
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 876,655 – 876,790 |
| Length | 135 |
| Max. P | 0.959578 |

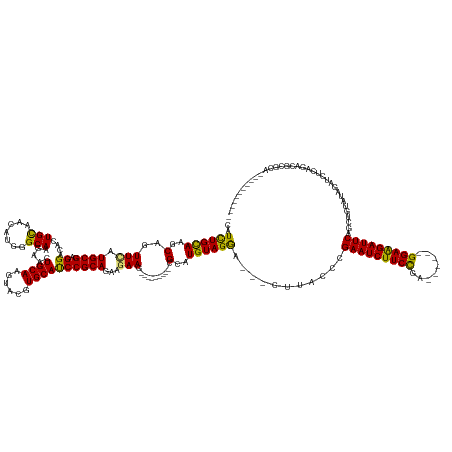
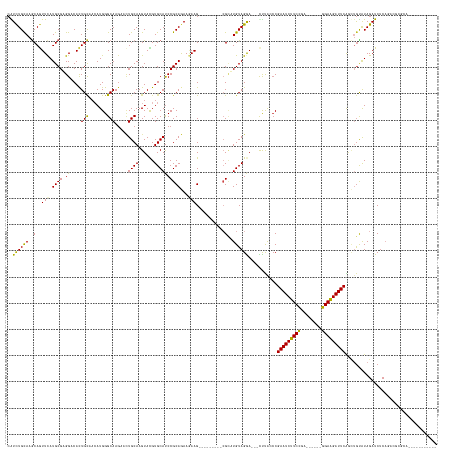
| Location | 876,655 – 876,790 |
|---|---|
| Length | 135 |
| Sequences | 12 |
| Columns | 164 |
| Reading direction | forward |
| Mean pairwise identity | 79.73 |
| Shannon entropy | 0.38851 |
| G+C content | 0.50391 |
| Mean single sequence MFE | -46.88 |
| Consensus MFE | -26.32 |
| Energy contribution | -25.95 |
| Covariance contribution | -0.37 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.56 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.67 |
| SVM RNA-class probability | 0.959578 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
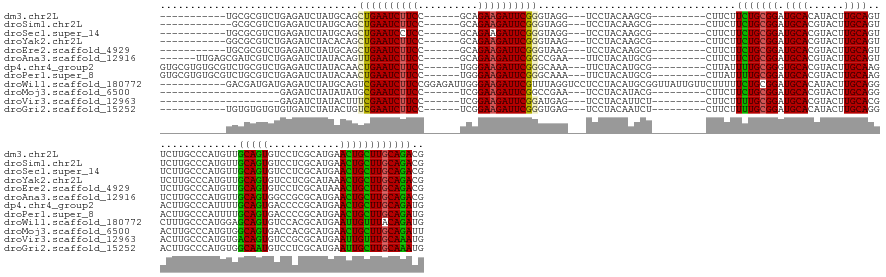
>dm3.chr2L 876655 135 + 23011544 CGUCUGCAAGCAGUUCAUGCGAGGACACUGCAACAUGGGCAAGAACUGCAAGUAUGUGCAUCCGCAGAAGAAG---------CGCUUGUAGGA---CCUACCCGAAUCUUCUGC------GGAAGAUUCAGCUGCAUAGAUCUCAGACGCGCA----------- ((((((...(((((((.(((..................))).))))))).((..(((((((((((((((((..---------((...((((..---.)))).))..))))))))------))).........))))))...))))))))....----------- ( -48.77, z-score = -1.86, R) >droSim1.chr2L 870156 134 + 22036055 CGUCUGCAAGCAGUUCAUGCGAGGACACUGCAACAUGGGCAAGAACUGCAAGUACGUGCAUCCGCAGAAGAAG---------CGCUUGUAGGA---CCUACCCGAAUCUUCUGC------GGAAGAUUCAGCUGCAUAGAUCUCAGACGCGC------------ ((((((...(((((((.(((..................))).)))))))......((((((((((((((((..---------((...((((..---.)))).))..))))))))------))).........)))))......))))))...------------ ( -47.47, z-score = -1.68, R) >droSec1.super_14 847368 135 + 2068291 CGUCUGCAAGCAGUUCAUGCGAGGACACUGCAACAUGGGCAAGAACUGCAAGUACGUGCAUCCGCAGAAGAAG---------CGCUUGUAGGA---CCUACCCGAAUCUUCUGC------GGAGGAUUCAGCUGCAUAGAUCUCAGACGCGCA----------- ((((((...(((((((.(((..................))).)))))))......((((((((((((((((..---------((...((((..---.)))).))..))))))))------))).........)))))......))))))....----------- ( -47.47, z-score = -1.28, R) >droYak2.chr2L 861188 135 + 22324452 CGUCUGCAAGCAGUUUAUGCGAGGACACUGCAACAUGGGCAAGAACUGCAAGUACGUGCAUCCGCAGAAGAAG---------CGCUUGUAGGA---CUUACCCGAAUCUUCUGC------GGAAGAUUCAGCUGUGUAGAUCUCAGACGCGCC----------- ((((((...(((.....)))((..((((.((.....(((.(((..(((((((..(.(((....)))......)---------..)))))))..---))).)))((((((((...------.)))))))).)).))))...)).))))))....----------- ( -50.40, z-score = -2.49, R) >droEre2.scaffold_4929 934777 135 + 26641161 CGUCUGCAAGCAGUUUAUGCGAGGACACUGCAACAUGGGCAAGAACUGCAAGUACGUGCAUCCGCAGAAGAAG---------CGCUUGUAGGA---CUUACCCGAAUCUUCUGC------GGAAGAUUCAGCUGCAUAGAUCUCAGACGCGCA----------- ((((((..((..(((((((((((....))((.....(((.(((..(((((((..(.(((....)))......)---------..)))))))..---))).)))((((((((...------.)))))))).)))))))))))))))))))....----------- ( -47.70, z-score = -1.84, R) >droAna3.scaffold_12916 2598448 140 + 16180835 CGUCUGCAAGCAGUUCAUGCGCGGCCACUGCAACAUGGGCAAGAACUGCAAGUACGUGCAUCCGCAGAAGAAG---------CGCAUGUAGAA---UUCGGCCGAAUCUUCUGC------GGAAGAUUCAACUGUAUAGAUCUCAGACGAUCGCUCAA------ ((((((...(((((((.(((.((((....))....)).))).)))))))..((((((.(.(((((((((((..---------((..((.....---..))..))..))))))))------))).).....)).))))......)))))).........------ ( -48.40, z-score = -1.68, R) >dp4.chr4_group2 773602 146 - 1235136 CAUCUGCAAGCAGUUCAUGCGGGGUCACUGCAAAAUGGGCAAGUCUUGCAAGUACGUGCACCCGCAAAAUAAG---------CGCAUGUAGAA---UUUGCCCGAAUCUUCCCA------GGAAGAUUCAGUUGUAUAGAUCUCAGACGCAGACGCACACGCAC ..(((((..(((.....)))((((((..(((((...((((((((..((((.(..(.(((....)))......)---------..).))))..)---)))))))((((((((...------.))))))))..)))))..))))))....))))).((....)).. ( -54.80, z-score = -4.37, R) >droPer1.super_8 3427463 146 + 3966273 CAUCUGCAAGCAGUUCAUGCGGGGUCACUGCAAAAUGGGCAAGUCUUGCAAGUACGUGCAUCCGCAAAAUAAG---------CGCAUGUAGAA---UUUGCCCGAAUCUUCCCA------GGAAGAUUCAGUUGUAUAGAUCUCAGACGCAGACGCACACGCAC ..(((((..(((.....)))((((((..(((((...((((((((..((((.(..(.(((....)))......)---------..).))))..)---)))))))((((((((...------.))))))))..)))))..))))))....))))).((....)).. ( -54.80, z-score = -4.43, R) >droWil1.scaffold_180772 6647328 153 - 8906247 CAUCUGUAAACAAUUCAUGCGUGGACACUGCUCCAUGGGCAAAGCCUGCAAGUAUGUGCAUCCGCAGAAAAAGAACAAUAACCGCAUGUAGGAGGACCUAAACGAAUCUUCCCAAUCUCCGGAAGAUUCGACUGCAUAGAUCUCAUCAUCGUC----------- .((((((........(((((((((((((((((...(((((...)))))..)))).)))..))))).(........).......)))))((((....))))..((((((((((........)))))))))).....))))))............----------- ( -40.70, z-score = -1.65, R) >droMoj3.scaffold_6500 8562702 126 - 32352404 AAUCUGCAAGCAGUUCAUGCGUGGUCACUGCCACAUGGGCAAGUCCUGCAAGUACGUGCAUCCGCAGAAGAAG---------CGUAUGUAGGA---UUCGGCCGAAUCUUCCGA------GGAAGAUUCGCAUAUAUAGAUCUC-------------------- ...(((....)))...(((((((((....)))))...(((.(((((((((..(((((...((....))....)---------)))))))))))---))..)))((((((((...------.))))))))))))...........-------------------- ( -44.60, z-score = -2.77, R) >droVir3.scaffold_12963 19424253 126 + 20206255 CAUUUGCAAACAAUUCAUGCGCGGACACUGUCACAUGGGCAAGUCGUGCAAGUACGUGCAUCCGCAAAAGAAG---------AGAAUGUAGGA---CUCAUCCGAAUCUUCCGA------GGAAGAUUCGAAAGUAUAGAUCUC-------------------- ..(((((..........(((((((....((((.....))))..))))))).((....))....)))))....(---------(((.(((((((---....)))((((((((...------.)))))))).....))))..))))-------------------- ( -34.90, z-score = -1.56, R) >droGri2.scaffold_15252 7288963 135 + 17193109 CAUUUGCAAGCAAUUCAUGCGAGGACAUUGCCACAUGGGCAAGUCCUGCAAGUAUGUGCAUCCGCAAAAGAAG---------AGAUUGUAGGA---CUCACCCGAAUCUUCCGA------GGAAGAUUCGACAGUAUAGAUCACACACACACA----------- .((((((..(((.....))).(((((.(((((.....)))))))))))))))).(((((.(((((((......---------...)))).)))---......(((((((((...------.)))))))))...)))))...............----------- ( -42.60, z-score = -3.70, R) >consensus CAUCUGCAAGCAGUUCAUGCGAGGACACUGCAACAUGGGCAAGAACUGCAAGUACGUGCAUCCGCAGAAGAAG_________CGCAUGUAGGA___CUUACCCGAAUCUUCCGA______GGAAGAUUCAGCUGUAUAGAUCUCAGACGCGCA___________ ..(((((..(((.....)))..((....(((.......))).....((((......)))).)))))))...................................(((((((((........)))))))))................................... (-26.32 = -25.95 + -0.37)

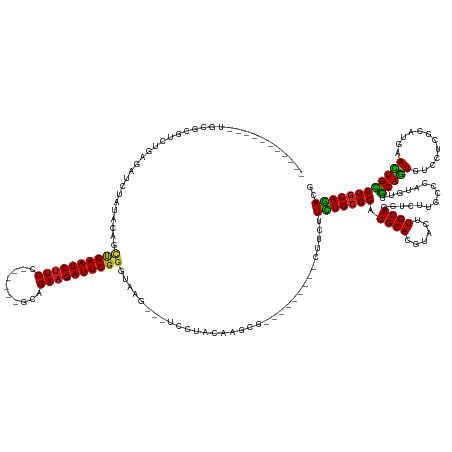
| Location | 876,655 – 876,790 |
|---|---|
| Length | 135 |
| Sequences | 12 |
| Columns | 164 |
| Reading direction | reverse |
| Mean pairwise identity | 79.73 |
| Shannon entropy | 0.38851 |
| G+C content | 0.50391 |
| Mean single sequence MFE | -46.50 |
| Consensus MFE | -26.86 |
| Energy contribution | -25.98 |
| Covariance contribution | -0.87 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.58 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.872822 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 876655 135 - 23011544 -----------UGCGCGUCUGAGAUCUAUGCAGCUGAAUCUUCC------GCAGAAGAUUCGGGUAGG---UCCUACAAGCG---------CUUCUUCUGCGGAUGCACAUACUUGCAGUUCUUGCCCAUGUUGCAGUGUCCUCGCAUGAACUGCUUGCAGACG -----------....((((((.(((...((((((.((((((((.------...))))))))(((((((---..((.((((.(---------(.(((.....))).)).....)))).))..)))))))..))))))..)))...(((.....)))...)))))) ( -47.50, z-score = -1.33, R) >droSim1.chr2L 870156 134 - 22036055 ------------GCGCGUCUGAGAUCUAUGCAGCUGAAUCUUCC------GCAGAAGAUUCGGGUAGG---UCCUACAAGCG---------CUUCUUCUGCGGAUGCACGUACUUGCAGUUCUUGCCCAUGUUGCAGUGUCCUCGCAUGAACUGCUUGCAGACG ------------...((((((.(((((((....((((((((((.------...)))))))))))))))---))...((((((---------.(((...(((((..((((((((..((((...))))....).))).))))..))))).))).)))))))))))) ( -47.90, z-score = -1.37, R) >droSec1.super_14 847368 135 - 2068291 -----------UGCGCGUCUGAGAUCUAUGCAGCUGAAUCCUCC------GCAGAAGAUUCGGGUAGG---UCCUACAAGCG---------CUUCUUCUGCGGAUGCACGUACUUGCAGUUCUUGCCCAUGUUGCAGUGUCCUCGCAUGAACUGCUUGCAGACG -----------....((((((.......((((.........(((------((((((((..((.((((.---..))))...))---------..)))))))))))))))....(..(((((((.(((.(((......))).....))).)))))))..))))))) ( -47.30, z-score = -1.08, R) >droYak2.chr2L 861188 135 - 22324452 -----------GGCGCGUCUGAGAUCUACACAGCUGAAUCUUCC------GCAGAAGAUUCGGGUAAG---UCCUACAAGCG---------CUUCUUCUGCGGAUGCACGUACUUGCAGUUCUUGCCCAUGUUGCAGUGUCCUCGCAUAAACUGCUUGCAGACG -----------....((((((.((...((((.((.((((((((.------...))))))))(((((((---..((.((((..---------(....((....)).....)..)))).))..))))))).....)).))))..))(((.....)))...)))))) ( -44.60, z-score = -1.21, R) >droEre2.scaffold_4929 934777 135 - 26641161 -----------UGCGCGUCUGAGAUCUAUGCAGCUGAAUCUUCC------GCAGAAGAUUCGGGUAAG---UCCUACAAGCG---------CUUCUUCUGCGGAUGCACGUACUUGCAGUUCUUGCCCAUGUUGCAGUGUCCUCGCAUAAACUGCUUGCAGACG -----------....((((((.(((...((((((.((((((((.------...))))))))(((((((---..((.((((..---------(....((....)).....)..)))).))..)))))))..))))))..)))...(((.....)))...)))))) ( -48.10, z-score = -2.08, R) >droAna3.scaffold_12916 2598448 140 - 16180835 ------UUGAGCGAUCGUCUGAGAUCUAUACAGUUGAAUCUUCC------GCAGAAGAUUCGGCCGAA---UUCUACAUGCG---------CUUCUUCUGCGGAUGCACGUACUUGCAGUUCUUGCCCAUGUUGCAGUGGCCGCGCAUGAACUGCUUGCAGACG ------.........((((((.......(((.(((((((((((.------...)))))))))))....---.....(((.((---------(.......))).)))...)))(..(((((((.(((((((......))))....))).)))))))..))))))) ( -46.60, z-score = -1.15, R) >dp4.chr4_group2 773602 146 + 1235136 GUGCGUGUGCGUCUGCGUCUGAGAUCUAUACAACUGAAUCUUCC------UGGGAAGAUUCGGGCAAA---UUCUACAUGCG---------CUUAUUUUGCGGGUGCACGUACUUGCAAGACUUGCCCAUUUUGCAGUGACCCCGCAUGAACUGCUUGCAGAUG (((((((((.((.(((((.((.....)).))..((((((((((.------...))))))))))))).)---)..))))))))---------)......((((((..(((.....(((((((........))))))))))..))))))....(((....)))... ( -50.90, z-score = -2.55, R) >droPer1.super_8 3427463 146 - 3966273 GUGCGUGUGCGUCUGCGUCUGAGAUCUAUACAACUGAAUCUUCC------UGGGAAGAUUCGGGCAAA---UUCUACAUGCG---------CUUAUUUUGCGGAUGCACGUACUUGCAAGACUUGCCCAUUUUGCAGUGACCCCGCAUGAACUGCUUGCAGAUG (((((.(((((((((((..((((..........((((((((((.------...))))))))))(((..---.......))).---------))))...))))))))))).((((.((((((........))))))))))....)))))...(((....)))... ( -52.20, z-score = -3.34, R) >droWil1.scaffold_180772 6647328 153 + 8906247 -----------GACGAUGAUGAGAUCUAUGCAGUCGAAUCUUCCGGAGAUUGGGAAGAUUCGUUUAGGUCCUCCUACAUGCGGUUAUUGUUCUUUUUCUGCGGAUGCACAUACUUGCAGGCUUUGCCCAUGGAGCAGUGUCCACGCAUGAAUUGUUUACAGAUG -----------((((((...((((((((......((((((((((........))))))))))..))))).)))...((((((..(((((((((......(((((((((......))))...)))))....)))))))))....)))))).))))))........ ( -48.10, z-score = -1.46, R) >droMoj3.scaffold_6500 8562702 126 + 32352404 --------------------GAGAUCUAUAUAUGCGAAUCUUCC------UCGGAAGAUUCGGCCGAA---UCCUACAUACG---------CUUCUUCUGCGGAUGCACGUACUUGCAGGACUUGCCCAUGUGGCAGUGACCACGCAUGAACUGCUUGCAGAUU --------------------..........(((((((((((((.------...))))))))((.(((.---((((.....((---------(.......)))...(((......))))))).))).))..((((......)))))))))..(((....)))... ( -39.60, z-score = -2.07, R) >droVir3.scaffold_12963 19424253 126 - 20206255 --------------------GAGAUCUAUACUUUCGAAUCUUCC------UCGGAAGAUUCGGAUGAG---UCCUACAUUCU---------CUUCUUUUGCGGAUGCACGUACUUGCACGACUUGCCCAUGUGACAGUGUCCGCGCAUGAAUUGUUUGCAAAUG --------------------((((.....((((((((((((((.------...))))))))))..)))---).......)))---------).......((((((((...(((..(((.....)))....)))...))))))))(((.........)))..... ( -37.30, z-score = -2.50, R) >droGri2.scaffold_15252 7288963 135 - 17193109 -----------UGUGUGUGUGUGAUCUAUACUGUCGAAUCUUCC------UCGGAAGAUUCGGGUGAG---UCCUACAAUCU---------CUUCUUUUGCGGAUGCACAUACUUGCAGGACUUGCCCAUGUGGCAAUGUCCUCGCAUGAAUUGCUUGCAAAUG -----------...((((((((.((((......((((((((((.------...))))))))))..(((---.........))---------).........))))))))))))((((((((((((((.....))))).))))..(((.....))).)))))... ( -47.90, z-score = -3.93, R) >consensus ___________UGCGCGUCUGAGAUCUAUACAGCUGAAUCUUCC______GCAGAAGAUUCGGGUAAG___UCCUACAAGCG_________CUUCUUCUGCGGAUGCACGUACUUGCAGGUCUUGCCCAUGUUGCAGUGUCCUCGCAUGAACUGCUUGCAGACG .................................((((((((((..........)))))))))).................................(((((((.((((......))))...............(((((............)))))))))))).. (-26.86 = -25.98 + -0.87)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:07:14 2011