
| Sequence ID | dm3.chrX |
|---|---|
| Location | 2,119,970 – 2,120,090 |
| Length | 120 |
| Max. P | 0.607629 |

| Location | 2,119,970 – 2,120,090 |
|---|---|
| Length | 120 |
| Sequences | 14 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.13 |
| Shannon entropy | 0.59074 |
| G+C content | 0.53516 |
| Mean single sequence MFE | -44.77 |
| Consensus MFE | -10.04 |
| Energy contribution | -10.56 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.69 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.22 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.607629 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
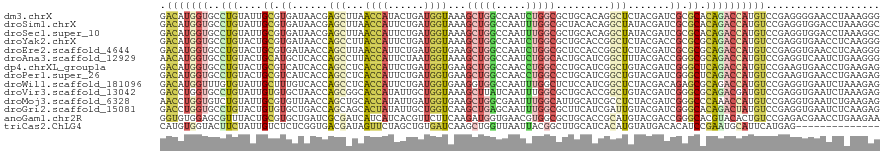
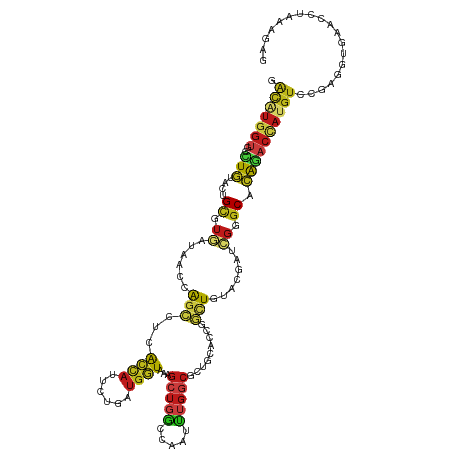
>dm3.chrX 2119970 120 + 22422827 GACAUGGUGCCUGUAUUGCGUGAUAACGAGCUUAACCAUACUGAUGGUAAAGCUGGCCAAUCUGGCGCUGCACAGGCUCUACGAUCGCGCACAGACCAUGUCCGAGGGGAACCUAAAGGG ((((((((..((((...(((((((...((((((.(((((....)))))...((.((((.....))).).))..))))))....)))))))))))))))))))..(((....)))...... ( -48.80, z-score = -3.76, R) >droSim1.chrX 1483049 120 + 17042790 GACAUGGUGCCUGUAUUGCGUGAUAACGAGCUUAACCAUUCUGAUGGUAAAGCUGGCCAAUUUGGCGCUACACAGGCUAUACGAUCGCGCACAGACCAUGUCCGAGGUGGACCUAAAGGC ((((((((..((((...(((((((..(((((((.(((((....))))).))))).(((.....)))(((.....)))....)))))))))))))))))))))..(((....)))...... ( -44.30, z-score = -2.98, R) >droSec1.super_10 1905772 120 + 3036183 GACAUGGUGCCUGUAUUGCGUGAUAACGAGCUUAACCAUUCUGAUGGUAAAGCUGGCCAAUUUGGCGCUGCACAGGCUAUACGAUCGCGCACAGACCAUGUCCGAGGUGGACCUAAAGGC ((((((((..((((...(((((((..(((((((.(((((....))))).))))).(((.....)))(((.....)))....)))))))))))))))))))))..(((....)))...... ( -44.60, z-score = -2.59, R) >droYak2.chrX 15561415 120 - 21770863 GACAUGGUGCCUGUACUGCGUGAUAACCAGCCUUACCAUUCUGAUGGUAAAGCUGGCCAAUCUGGCGCUGCACCGGCUCUACGACCGCGCGCAGACCAUGUCCGAGGUGAACCUCAAGGG ((((((((..((((...(((((....(((((.(((((((....))))))).)))))....(((((.((((...)))).))).)).))))))))))))))))).((((....))))..... ( -51.80, z-score = -3.89, R) >droEre2.scaffold_4644 2085098 120 + 2521924 GACAUGGUGCCUGUACUGCGUGAUAACCAGCUUAACCAUUCUGAUGGUGAAGCUGGCCAAUCUGGCGCUCCACCGGCUCUACGAUCGCGCGCAGACCAUGUCCGAGGUGAACCUCAAGGG ((((((((..((((...(((((((..(((((((.(((((....))))).)))))))......(((.(((.....))).)))..))))))))))))))))))).((((....))))..... ( -52.60, z-score = -4.24, R) >droAna3.scaffold_12929 2018335 120 + 3277472 AACAUGGUGCCUGUACUGCAUGCUCACCAGCCUUACCAUUCUAAUGGUAAAGCUGGCCAAUCUGGCACUGCAUCGGCUUUACGACCGGGCGCAGACCAUGUCCGAGGUCAAUCUGAAGGG ......(((((((...(((((((.(((((((.(((((((....))))))).)))))......))))).))))(((......))).))))))).((((........))))........... ( -42.60, z-score = -1.70, R) >dp4.chrXL_group1a 133579 120 + 9151740 GACAUGGUGCCUGUACUGCGUCAUCACCAGCCUCACCAUUCUGAUGGUGAAGCUGGCCAACCUGGCCCUGCAUCGGCUGUACGAUCGGGCUCAGACCAUGUCCGAAGUGAACCUGAAGAG ((((((((..................(((((.(((((((....))))))).))))).....(((((((.(.((((......))))))))).))))))))))).................. ( -46.20, z-score = -2.32, R) >droPer1.super_26 811570 120 - 1349181 GACAUGGUGCCUGUACUGCGUCAUCACCAGCCUCACCAUUCUGAUGGUGAAGCUGGCCAACCUGGCCCUGCAUCGGCUGUACGAUCGGGCUCAGACCAUGUCCGAAGUGAACCUGAAGAG ((((((((..................(((((.(((((((....))))))).))))).....(((((((.(.((((......))))))))).))))))))))).................. ( -46.20, z-score = -2.32, R) >droWil1.scaffold_181096 8753662 120 - 12416693 GACAUGGUUUGUGUAUUGCUUUGUCACCAGCCUCACCAUUCUGAUGGUGAAGGUGGCCAAUUUGGCUCUCCAUCGGCUCUACGACAGAGCGCAGACCAUGUCCGAGGUGAAUCUAAAGAG ((((((((((((.....((((((((...(((((((((((....))))))).((.((((.....))))..))...))))....)))))))))))))))))))).................. ( -51.20, z-score = -4.59, R) >droVir3.scaffold_13042 877506 120 + 5191987 GACCUGGUGCCUGUAUUGUGUGCUAACCAGCGGCACCAUAUUGCUGGUAAAGCUUAUCAAUUUGGCGCUGCACCGGCUGUACGAUCGGGCGCAGACGAUGUCCGAGGUGAAUCUAAAGAG .....((((((...((((...(((.((((((((.......))))))))..)))....))))..)))))).(((((......)..((((((((....).)))))))))))........... ( -39.00, z-score = -0.39, R) >droMoj3.scaffold_6328 2869795 120 + 4453435 AACCUGGUGUCUGUAUUGCGUGUUAACCAGCUGCACCAUAUUGAUGGUGAAGCUGGCGAAUUUGGCAUUGCAUCGCCUCUACGAUCGGGCCCAAACCAUGUCCGAGGUGAAUCUGAAGAG .(((.((((..((((.(((..(((..((((((.((((((....)))))).))))))..)))...))).)))).)))).......((((((.........)))))))))............ ( -42.60, z-score = -2.43, R) >droGri2.scaffold_15081 1258579 120 + 4274704 GACCUGGUGCCUGUACUGUGUGCUGACCAGCAGCACUAUAUUGCUGGUCAAGCUGAGCAAUUUGGCGCUUCAUCGAUUGUACGAUCGGGCACAGACUAUGUCCGAGGUGAAUCUCAAGAG ((...((((((.....(((..((((((((((((.......))))))))).)))...)))....))))))...))((((.(((..(((((((.......))))))).)))))))....... ( -44.20, z-score = -2.08, R) >anoGam1.chr2R 46776778 120 + 62725911 GGUGUGGAGCGUUUACUGCGUGCUGAUCGCGAUCAUCAUCACGUUCUUCAAGAUGGUGAACGUGGCGCUGCACCGCAUGUACGACCGGGCACGUACACUGUCCGAGACGAACCUGAAGAA ((((..(.((((((((((((((.((((.......)))).)))))((.....)).)))))))))....)..)))).((.((.((.(((((((.(....))))))).).)).)).))..... ( -39.70, z-score = 0.22, R) >triCas2.ChLG4 6796830 106 - 13894384 CAUGUGGUACUUCUAUUGUCUCUCGGUGACGAUAGUUCUAGCUGUGAUCAAGCUGGUUAAUUACGGCUUGCAUCACAUGUAUGACACAUCCGAAUGCAUUCAUGAG-------------- ((((..(((.(((...(((.(((((....)))........(((((((((((((((........))))))).)))))).))..)).)))...))))))...))))..-------------- ( -33.00, z-score = -2.25, R) >consensus GACAUGGUGCCUGUACUGCGUGAUAACCAGCCUCACCAUUCUGAUGGUAAAGCUGGCCAAUUUGGCGCUGCACCGGCUGUACGAUCGGGCACAGACCAUGUCCGAGGUGAACCUAAAGAG .(((((((..(((....((.((......(((...((((......))))...(((((.....))))).........))).......)).)).))))))))))................... (-10.04 = -10.56 + 0.52)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:08:36 2011