
| Sequence ID | dm3.chrX |
|---|---|
| Location | 1,920,400 – 1,920,520 |
| Length | 120 |
| Max. P | 0.628480 |

| Location | 1,920,400 – 1,920,520 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 121 |
| Reading direction | reverse |
| Mean pairwise identity | 78.13 |
| Shannon entropy | 0.53837 |
| G+C content | 0.61345 |
| Mean single sequence MFE | -47.61 |
| Consensus MFE | -17.98 |
| Energy contribution | -18.47 |
| Covariance contribution | 0.49 |
| Combinations/Pair | 1.61 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.628480 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
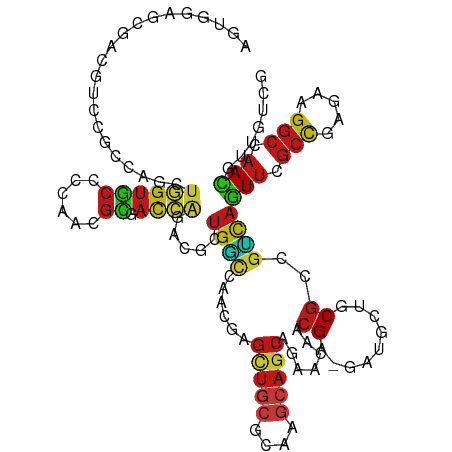
>dm3.chrX 1920400 120 - 22422827 AGUGGAGCGACGUCCGCCAACUGGUGCCCCAACGCGACCAGACUUUGGCCAACGAGCUGCGCAAGCAACAGAACAACGA-AAUGCUCCGCCGUCAGUUUGCCGAGAAGGCCAACAUUGUCG .((((((((.(((..((((((((((((......)).)))))...)))))..))).(.(((....))).)..........-..))))))))((.(((((.(((.....))).)))..)).)) ( -44.20, z-score = -2.39, R) >droSim1.chrX_random 1147514 120 - 5698898 AGUGGAGCGACGUCCGCCAACUGGUGCCCCAACGCGACCAGACUUUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUCCGCCGUCAGUUUGCCGAGAAGGCCAACAUUGUCG .((((((((.(((..((((((((((((......)).)))))...)))))..))).(((((....)))))..........-..))))))))((.(((((.(((.....))).)))..)).)) ( -50.80, z-score = -3.68, R) >droYak2.chrX 15364813 120 + 21770863 AGUGGAGCGACGUCCGCCAGCUGGUGCCCCAACGCGACCAGACUUUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUCCGCCGUCAGUUUGCCGAGAAGGCCAACAUUGUCG .((((((((.(((..((((.(((((((......)).)))))....))))..))).(((((....)))))..........-..))))))))((.(((((.(((.....))).)))..)).)) ( -50.70, z-score = -3.26, R) >droEre2.scaffold_4644 1892508 120 - 2521924 AGUGGAGCGACGUCCGCCAGCUGGUGCCCCAACGCGACCAGACCUUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUCCGCCGUCAGUUUGCCGAGAAGGCAAACAUUGUCG .((((((((.(((..((((.(((((((......)).)))))....))))..))).(((((....)))))..........-..))))))))((.(((((((((.....)))))))..)).)) ( -54.80, z-score = -4.50, R) >droAna3.scaffold_12929 1914416 120 - 3277472 AGUGGAGCGAUGUCCGCCAGCUGGUUCCCCAGCGGGACCAGACACUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUGCGCCGUCAGUUCGCCGAGAAGGCCAACAUUGUUG .....((((((((..(((((((((((((.....))))))))...)))))....((((((((...((((((.........-..))))))..)).))))))(((.....)))..)))))))). ( -51.10, z-score = -2.67, R) >droSec1.super_17 128218 91 + 1527944 ------------------------------AACCCUGCGAUCCCGAGAUCAUUGUCUCGCGCGUCCAGCAACUGGAGGAUGCUUACGCAGAGUUGGUCCGUUUGGCCGUGGAACGUCGCAG ------------------------------(((.(((((.....(((((....)))))((((.(((((...))))).).)))...))))).)))((((.....))))((((....)))).. ( -31.20, z-score = -0.52, R) >dp4.chrXL_group1a 2593913 120 - 9151740 AGUGGAGCGACGUCCGCCAGCUGGUGCCACAACGCGACCAGACGCUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUGCGCCGUCAGUUCGCCGAGAAGGCCAAUGUGGUGG .(((.((((.(((..((((((((((((......)).))))...))))))..))).(((((....)))))..........-..)))).)))(..(((((.(((.....))).))).))..). ( -48.50, z-score = -0.67, R) >droPer1.super_28 1031113 120 - 1111753 AGUGGAGCGACGUCCGCCAGCUGGUGCCACAACGCGACCAGACGCUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUGCGCCGUCAGUUCGCCGAGAAGGCCAAUGUGGUGG .(((.((((.(((..((((((((((((......)).))))...))))))..))).(((((....)))))..........-..)))).)))(..(((((.(((.....))).))).))..). ( -48.50, z-score = -0.67, R) >droWil1.scaffold_181096 5440550 120 + 12416693 AGUGGACUGAUGUGCGUCAGCUGGUGCCCCAGCGCGAUCAGACACUGGCCAACGAGCUGCGCAAGCAGCAAAAUAACGA-AAUGCUGCGCCGCCAGUUUGCCGAGAAGGCCAACGUGGUGG ............((((.(((((.((...((((.((.....).).))))...)).))))))))).((((((.........-..)))))).(((((((((.(((.....))).))).)))))) ( -48.50, z-score = -1.42, R) >droMoj3.scaffold_6473 14017219 120 + 16943266 AGUGGACCGAUGUCCGCCAGCUGGUGCCACAGCGCGACCAGACGUUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUGCGCCGCCAGUUCGCCGAGAAGGCCAAUGUGGUCG .((((((....))))))..((((......)))).(((((..(((((((((..(((((((.((..((((((.........-..))))))...))))))))).......)))))))))))))) ( -56.20, z-score = -3.35, R) >droVir3.scaffold_12928 7146656 120 - 7717345 AAUGGACCGAUGUCCGCCAGCUGGUGCCGCAGCGCGACCAGACGCUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUGCGCCGCCAGUUCGCUGAAAAGGCCAAUGUGGUCG ...((((....))))((.((((((((.(((((((((.((((...)))).).....(((((....)))))..........-).))))))).)))))))).))......((((.....)))). ( -54.80, z-score = -2.71, R) >droGri2.scaffold_14853 1385673 120 - 10151454 AAUGGAGCGAUGUCCGCCAAUUGGUGCCGCAACGCGAUCAGACGCUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUCCGUCGCCAGUUUGCCGAGAAGGCCAACGUUGUCG ..(((.(((.(((.((((....))))..))).)))..)))(((((((((..(((.(((((....)))))........((-(...)))))).))))))..(((.....))).......))). ( -43.60, z-score = -0.72, R) >anoGam1.chr2R 5647381 120 - 62725911 AGUGGUCGGAGGUGCGCGCCCUGGUGCCGCAGCGCGACCAGACGCUGGCGAACGAGCUGCGCAAGCAGCAGAACAACGA-GAUGCUGCGCCGCCAGUUCGCCGAGAAGGCGAACGCGGUCG ..((((((..(.(((((((....))).)))).).))))))((((((((((.....(((((....))))).......((.-.......)).)))))(((((((.....))))))))).))). ( -60.20, z-score = -1.65, R) >apiMel3.Group11 12277601 120 + 12576330 AGUGGAGCGAUGUCAGGCAACUCGUGCCCCAACGUGACGGCACGCUUCAAGCUGAGCUUCGUAAACAACAGAACAACGA-AUUGCUUCGACGACAAUUUGCCGAGAAGGCUAAUGCAGUUG ..(((((((.((((((....))(((......)))....)))))))))))(((((((((((((.............))))-)..))).........(((.(((.....))).))).))))). ( -30.82, z-score = 0.85, R) >triCas2.ChLG4 11023440 120 + 13894384 AAUGGGGCGAAGUCCGGCAACUGGUGCCCCAACGCGACGCCACCCUGCAAGGCGAGCUCCGCAAGCAGCAAAACAACGA-AAUGUUGCGUCGGCAAUUCGCGGAAAAAGCCAACGCUGUUG ..(((((((.(((......)))..)))))))..(((..((......))..(((....(((((..((((((.........-..))))))(....).....)))))....)))..)))..... ( -40.20, z-score = 0.40, R) >consensus AGUGGAGCGACGUCCGCCAGCUGGUGCCCCAACGCGACCAGACGCUGGCCAACGAGCUGCGCAAGCAGCAGAACAACGA_GAUGCUGCGCCGUCAGUUCGCCGAGAAGGCCAACGUUGUCG .....................((((((......)).)))).....((((......(((((....))))).......((.........))..))))(((.(((.....))).)))....... (-17.98 = -18.47 + 0.49)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:07:56 2011