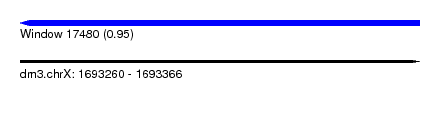
| Sequence ID | dm3.chrX |
|---|---|
| Location | 1,693,260 – 1,693,366 |
| Length | 106 |
| Max. P | 0.953462 |

| Location | 1,693,260 – 1,693,366 |
|---|---|
| Length | 106 |
| Sequences | 8 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 81.01 |
| Shannon entropy | 0.37132 |
| G+C content | 0.30664 |
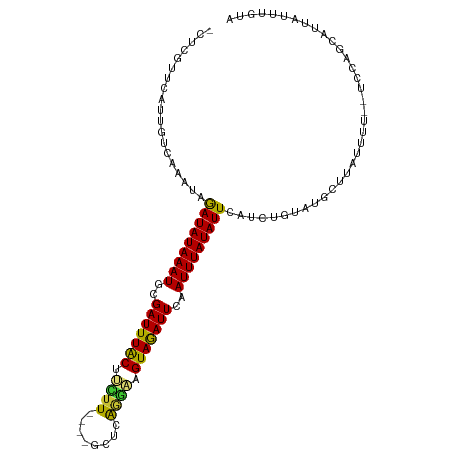
| Mean single sequence MFE | -20.84 |
| Consensus MFE | -11.97 |
| Energy contribution | -11.06 |
| Covariance contribution | -0.90 |
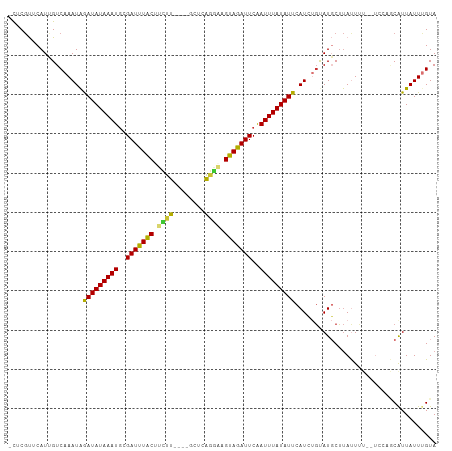
| Combinations/Pair | 1.40 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.57 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.59 |
| SVM RNA-class probability | 0.953462 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
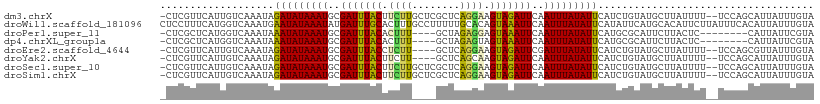
>dm3.chrX 1693260 106 - 22422827 -CUCGUUCAUUGUCAAAUAGAUAUAAAUGCGAUUUACUUCUUGCUCGCUCAGGAAGUAGAUUCAAUUUAUAUUCAUCUGUAUGCUUAUUUU--UCCAGCAUUAUUUGUA -............(((((((((((((((..(((((((((((((......)))))))))))))..))))))))).......(((((......--...))))))))))).. ( -24.40, z-score = -3.19, R) >droWil1.scaffold_181096 7770542 109 + 12416693 CUCCUUUCAUGGUCAAAUGAAUAUAAAUAUGAUUUGCACUUUGCCUUUUUGCACAGUAAAUUCAAUUUAUAUUCAUAUUCAUGCACAUUCUUAUUUCACAUUAUUUGUA .......(((((....((((((((((((.(((.((..(((.(((......))).))).)).)))))))))))))))..)))))..............(((.....))). ( -20.80, z-score = -3.23, R) >droPer1.super_11 854988 96 - 2846995 -CUCGCUCAUGGUCAAAUAAAUAUAAAUGCGAUUUACACUUU----GCUAGAGGAGUAAAUUCAAUUUAUAUUCAUGCGCAUUCUUACUC--------CAUUAUUCGUA -...((.(((((........((((((((..(((((((.((((----....)))).)))))))..))))))))))))).))..........--------........... ( -18.00, z-score = -1.90, R) >dp4.chrXL_group1a 4277520 96 - 9151740 -CUCGCUCAUGGUCAAAUAAAUAUAAAUGCGAUUUACACUUU----GCUAGAGUAGUAAAUUCAAUUUAUAUUCAUGCGCAUUCUUACUC--------CAUUAUUCGUA -...((.(((((........((((((((..(((((((((((.----....)))).)))))))..))))))))))))).))..........--------........... ( -18.10, z-score = -2.39, R) >droEre2.scaffold_4644 1671003 102 - 2521924 -CUCGUUCAUUGUCAAAUAGAUAUAAAUGCGAUUUACCUCUU----GCUCAGGAAGUAGAUUCGAUUUAUAUUCAUCUGUAUGCUUAUUUU--UCCAGCGUUAUUUGUA -............(((((((((((((((.((((((((.((((----....)))).))))).)))))))))))).......(((((......--...))))))))))).. ( -19.30, z-score = -1.30, R) >droYak2.chrX 1538830 102 - 21770863 -CUCGUUCAUUGUCAAAUAGAUAUAAAUGCGAUUUACUUCUU----GCUCAGCAAGUAGAUUCAAUUUAUAUUCAUCUGUAUGCUUAUUUU--UCCAGCAUUAUUUGUA -............(((((((((((((((..(((((((((((.----....)).)))))))))..))))))))).......(((((......--...))))))))))).. ( -17.30, z-score = -1.21, R) >droSec1.super_10 1488083 106 - 3036183 -CUCGUUCAUUGUCAAAUAGAUAUAAAUGCGAUUUACUUCUUGCUCGCUCAGGAAGUAGAUUCAAUUUAUAUUCAUCUGUAUGCUUAUUUU--UCCAGCAUUAUUUGUA -............(((((((((((((((..(((((((((((((......)))))))))))))..))))))))).......(((((......--...))))))))))).. ( -24.40, z-score = -3.19, R) >droSim1.chrX 1191210 106 - 17042790 -CUCGUUCAUUGUCAAAUAGAUAUAAAUGCGAUUUACUUCUUGCUCGCUCAGGAAGUAGAUUCAAUUUAUAUUCAUCUGUAUGCUUAUUUU--UCCAGCAUUAUUUGUA -............(((((((((((((((..(((((((((((((......)))))))))))))..))))))))).......(((((......--...))))))))))).. ( -24.40, z-score = -3.19, R) >consensus _CUCGUUCAUUGUCAAAUAGAUAUAAAUGCGAUUUACUUCUU____GCUCAGGAAGUAGAUUCAAUUUAUAUUCAUCUGUAUGCUUAUUUU__UCCAGCAUUAUUUGUA ...................(((((((((..(((((((.((((........)))).)))))))..))))))))).................................... (-11.97 = -11.06 + -0.90)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:07:24 2011