
| Sequence ID | dm3.chrX |
|---|---|
| Location | 1,675,802 – 1,675,880 |
| Length | 78 |
| Max. P | 0.852677 |

| Location | 1,675,802 – 1,675,880 |
|---|---|
| Length | 78 |
| Sequences | 15 |
| Columns | 78 |
| Reading direction | reverse |
| Mean pairwise identity | 88.39 |
| Shannon entropy | 0.29036 |
| G+C content | 0.40000 |
| Mean single sequence MFE | -20.70 |
| Consensus MFE | -15.40 |
| Energy contribution | -15.79 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.74 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.92 |
| SVM RNA-class probability | 0.852677 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 1675802 78 - 22422827 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.14, R) >droSim1.chrX 1184373 78 - 17042790 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.14, R) >droSec1.super_10 1470703 78 - 3036183 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.14, R) >droYak2.chrX 1521128 78 - 21770863 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.14, R) >droEre2.scaffold_4644 1653644 78 - 2521924 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.14, R) >droAna3.scaffold_12929 1681678 78 + 3277472 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.14, R) >dp4.chrXL_group1a 4244148 78 - 9151740 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.14, R) >droPer1.super_11 819615 78 - 2846995 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.14, R) >droWil1.scaffold_181096 7702525 78 + 12416693 UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUAAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.40, z-score = -2.20, R) >droVir3.scaffold_12942 110462 78 + 183257 UGAGGACUAAAUAUCCGUGCAUCCCCACAUGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.20, z-score = -1.88, R) >droMoj3.scaffold_6328 36897 78 + 4453435 UGAGGACUAAAUAUCCGUGCAUCCCCACAUGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.20, z-score = -1.88, R) >droGri2.scaffold_14853 9335297 78 + 10151454 UGAGGACUAAAUAUCCGUGCAUCCCCACAUGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((((......)))..((((((((((....))))).((....)))))))..)))))))))))). ( -20.20, z-score = -1.88, R) >anoGam1.chrX 19110708 78 + 22145176 UGCGGGCUGAAGACGCGCGCAUCGCCGCACGGUGCCGUGUUGAUGUACAGAUGGAAGUAGAUGCCGUCCUUCAGCGUG ..((.((((((((((((.((((((.....))))))))))).........(((((.........)))))))))))).)) ( -31.10, z-score = -1.01, R) >apiMel3.Group15 3289257 78 - 7856270 UGUGGUGAAAAUAUUCUUGCAUCACCACAUGGUGCGGUAUUUAUGUACAAAUGAAACUGAAUGCCUUGUUUUAGCUUA ((((((((.............)))))))).(((.((((.(((((......)))))))))...)))............. ( -17.82, z-score = -1.17, R) >triCas2.ChLG4 12729145 78 - 13894384 UGAGGACAAAAAAUUCGAGCGUCACCACACGGUGCGAUAUUUAUGUAAAGGUGGAAUUCGAUUCCAUCCUUAAGUUUG ((((((........(((((..((((((((..(........)..)))...)))))..))))).....))))))...... ( -17.42, z-score = -0.92, R) >consensus UGAGGACUAAAUAUCCGUGCAUCCCCACAAGGUGCUGUAUUUAUAUACAAAUGGAAAUGUACCCCGGAUUUUAGUUUA ...((((((((.(((((.(((((.......))))).((((....))))................))))))))))))). (-15.40 = -15.79 + 0.39)

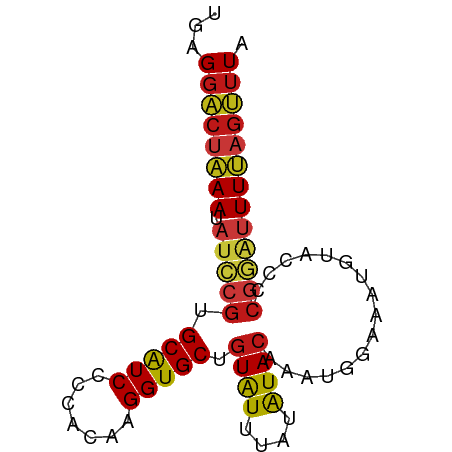
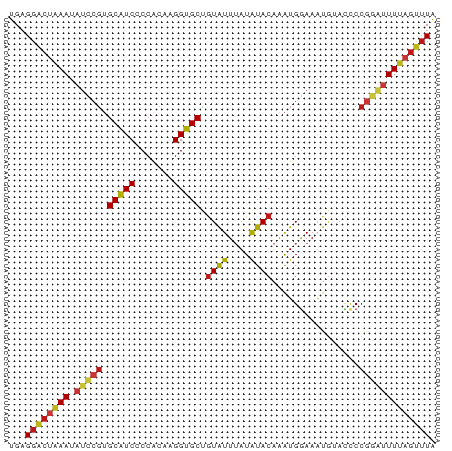
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:07:20 2011