
| Sequence ID | dm3.chrX |
|---|---|
| Location | 712,459 – 712,565 |
| Length | 106 |
| Max. P | 0.813726 |

| Location | 712,459 – 712,565 |
|---|---|
| Length | 106 |
| Sequences | 14 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 78.82 |
| Shannon entropy | 0.47331 |
| G+C content | 0.52398 |
| Mean single sequence MFE | -35.06 |
| Consensus MFE | -15.74 |
| Energy contribution | -15.25 |
| Covariance contribution | -0.49 |
| Combinations/Pair | 1.53 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.45 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.813726 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 712459 106 - 22422827 UGCGUUGGAUAUGCCCGAGCUACAGACGGAAAUGAUCUGCAUCCUGAUCAAGCAGACCUCCCGCCACUUGGGCCAGAAGCUCAGUGUGGGCGUCCAGGUAAACAAG--- (((.((((((..(((((.........(((....(.(((((...........))))).)..))).((((.((((.....))))))))))))))))))))))......--- ( -32.00, z-score = -0.13, R) >droSec1.super_10 557297 106 - 3036183 UGCGUUGGAUAUGCCCGAGCUACAGACGGAAAUGAUCUGCAUCCUGAUCAAGCAGACCUCCCGCCACUUGGGCCAGAAGCUCAGUGUCGGCGUCCAGGUAAACAAG--- (((.((((((.((((((.........)))...(((((........))))).)))......(((.((((.((((.....)))))))).))).)))))))))......--- ( -30.20, z-score = 0.06, R) >droYak2.chrX 636255 106 - 21770863 UGCGCUGGAUAUGCCCGAGCUACAGACGGAAAUGAUCUGCAUCCUGAUCAAGCAGACCUCCCGCCACAUGGGCCAGAAGCUCAGUGUCGGCGUCCAGGUAAACAAG--- (((.((((((.((((((.........)))...(((((........))))).)))......(((.(((.(((((.....)))))))).))).)))))))))......--- ( -32.90, z-score = -0.61, R) >droEre2.scaffold_4644 676855 106 - 2521924 UGCGUUGGAUAUGCCCGAGCUACAGACGGAAAUGAUCUGCAUCCUGAUCAAGCAGACCUCCCGCCACAUGGGCCAGAAGCUCAGUGUCGGCGUCCAGGUAAACAAG--- (((.((((((.((((((.........)))...(((((........))))).)))......(((.(((.(((((.....)))))))).))).)))))))))......--- ( -30.20, z-score = -0.01, R) >droAna3.scaffold_12929 457090 106 - 3277472 CGCCCUGGACAUGCCCGAGCUACAGACGGAGAUGAUCUGCAUCCUGAUCAAGCAGACCUCCCGGCACAUGGGCCAGAAGCUCAGUGUCGGCGUCCAGGUAAACAAG--- ...(((((((.((((((.........)))...(((((........))))).)))......(((((((.(((((.....)))))))))))).)))))))........--- ( -40.50, z-score = -2.20, R) >dp4.chrXL_group3a 2496733 106 - 2690836 CUCCCUGGACAUGCCCGAGCUGCAGACGGAGAUGAUCUGCAUAUUGAUCAAGCAGACCUCCCGGCACAUGGGCCAGAAGCUCAGUGUCGGCGUCCAGGUAAACAAG--- ...(((((((.(((.(((..((((((((....)).))))))..))).....)))......(((((((.(((((.....)))))))))))).)))))))........--- ( -41.00, z-score = -2.37, R) >droPer1.super_11 660565 106 + 2846995 CUCCCUGGACAUGCCCGAGCUGCAGACGGAGAUGAUCUGCAUAUUGAUCAAGCAGACCUCCCGGCACAUGGGCCAGAAGCUCAGUGUCGGCGUCCAGGUAAACAAG--- ...(((((((.(((.(((..((((((((....)).))))))..))).....)))......(((((((.(((((.....)))))))))))).)))))))........--- ( -41.00, z-score = -2.37, R) >droWil1.scaffold_181096 1480720 106 + 12416693 CUGUUUGGAUAUGCCGGAACUGCAAACCGAAAUGAUUUGCAUAUUGAUUAAACAAACUUCCCGACAUAUGGGACAAAAACUAAGUGUCGGCGUCCAGGUAAACAAG--- .((..(((((..(((((((.((((((.(.....).))))))..(((......)))..)))).(((((.(((........))).)))))))))))))..))......--- ( -27.30, z-score = -2.18, R) >droVir3.scaffold_12726 834605 106 + 2840439 UUGCCUGGACAUGCCGGAGCUACAGACCGAAAUGAUCUGCAUUUUGAUCAAGCAAACCUCCCGACAUAUGGGCCAGAAACUAAGUGUCGGUGUCCAGGUAAACAAG--- (((((((((((((((((.........)))...(((((........))))).)))......(((((((.(((........))).)))))))))))))))))).....--- ( -40.10, z-score = -4.56, R) >droMoj3.scaffold_6473 6466749 106 - 16943266 UUGCCUGGAUAUGCCGGAGCUGCAGACCGAAAUGAUCUGCAUUCUGAUAAAGCAAACCUCCCGACAUAUGGGCCAGAAACUAAGUGUCGGUGUCCAGGUAAACAAG--- (((((((((((((((((((.((((((.(.....).))))))))))).....)))......(((((((.(((........))).)))))))))))))))))).....--- ( -41.60, z-score = -4.99, R) >droGri2.scaffold_15203 11036463 106 + 11997470 UUGCCUGGACAUGCCGGAACUACAGACCGAAAUGAUUUGCAUUCUAAUUAAGCAAACCUCCCGACAUAUGGGUCAGAAACUAAGUGUCGGUGUCCAGGUAAACAAG--- (((((((((((((((((.........)))...(((((........))))).)))......(((((((.(((........))).)))))))))))))))))).....--- ( -35.20, z-score = -3.79, R) >anoGam1.chr2R 50109569 106 + 62725911 AUGCCUGGACAUGCCCGAGCUUCAGUCGGAGCUGAUCUGCGCACUGAUCAAGCAAACGUCCCGCCAUACCGGCCAGAAGCUGGGUGUAGGUGUCCAGGUAAACAAG--- .(((((((((((((((.((((((.(((((.(((((((........)))).)))....(.....)....)))))..))))))))))....)))))))))))......--- ( -47.00, z-score = -3.92, R) >apiMel3.Group10 7283018 100 + 11440700 AUGUAUAGACCAGCCUGAACUGCAAUCUGAAUUCAUAUGUGCAUUGGUAAAGCAGACAAGUCGUCACAC---UCAACACC---GUUUGGGCGUACAGGUAAAAAAG--- ........(((.(((..(((((((...((....))....))))..(((..((..(((.....)))...)---)....)))---)))..))).....))).......--- ( -19.90, z-score = 0.48, R) >triCas2.ChLG4 6824101 106 + 13894384 AUGUUUGGACAUGCCGGAGCUACAGGCCGAACUCAUUUGUGCGUUAAUUAAGCAAACGAGCAAAAGCUC--GCCCUUGCCCAAGC-UCGGAGUCCAGGUAUCUACACAA (((..(((((...(((.((((...(((............(((.........)))..(((((....))))--).....)))..)))-)))).)))))..)))........ ( -31.90, z-score = -1.35, R) >consensus UUCCCUGGACAUGCCCGAGCUACAGACGGAAAUGAUCUGCAUACUGAUCAAGCAGACCUCCCGACACAUGGGCCAGAAGCUCAGUGUCGGCGUCCAGGUAAACAAG___ ....((((((..(((...(((...........(((((........)))))..........((........))..........)))...)))))))))............ (-15.74 = -15.25 + -0.49)

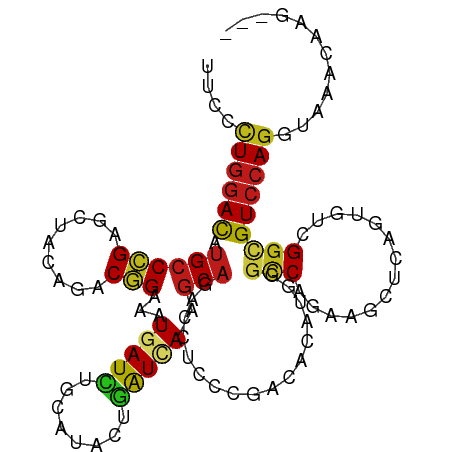
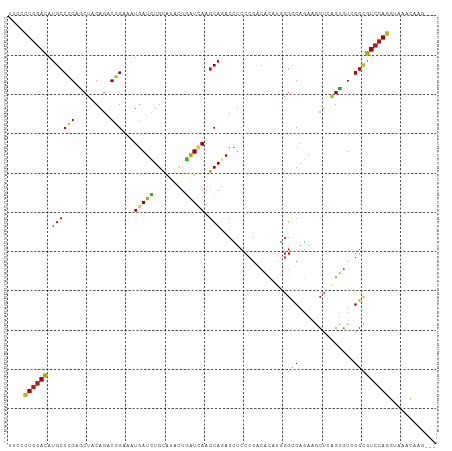
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:03:56 2011