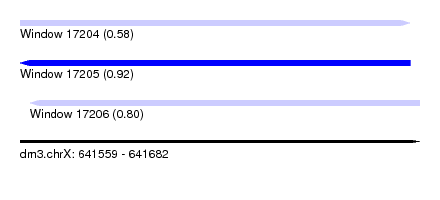
| Sequence ID | dm3.chrX |
|---|---|
| Location | 641,559 – 641,682 |
| Length | 123 |
| Max. P | 0.918203 |

| Location | 641,559 – 641,679 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.35 |
| Shannon entropy | 0.42033 |
| G+C content | 0.52615 |
| Mean single sequence MFE | -40.74 |
| Consensus MFE | -24.67 |
| Energy contribution | -24.61 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.61 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.580720 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
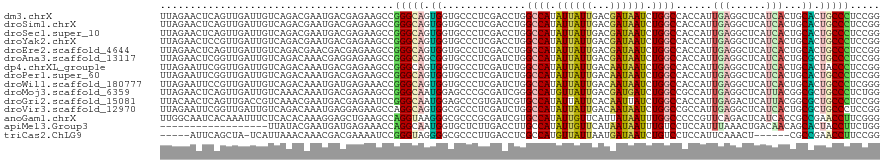
>dm3.chrX 641559 120 + 22422827 GAGCCGGAGGGCAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUCGUCAAUAAUAUGGCCAGGUCGAGGGCACCACUGCCCGGCUUCUCGUCAUUCGUCUGACAAUCAACUGAGUUC (((((((.((((((((((....)).((((......((((((.(((((.....))))).))))))......))))..))))))))........((((......))))......))).)))) ( -44.10, z-score = -1.54, R) >droSim1.chrX 528197 120 + 17042790 GAGCCGGAGGGCAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUCGUCAAUAAUAUGGCCAGGUCGAGGGCACCACUGCCCGGCUUCUCGUCAUUCGUCUGACAAUCAACUGAGUUC (((((((.((((((((((....)).((((......((((((.(((((.....))))).))))))......))))..))))))))........((((......))))......))).)))) ( -44.10, z-score = -1.54, R) >droSec1.super_10 491131 120 + 3036183 GAGCCGGAGGGCAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUCGUCAAUAAUAUGGCCAGGUCGAGGGCACCACUGCCCGGCUUCUCGUCAUUCGUCUGACAAUCAACUGAGUUC (((((((.((((((((((....)).((((......((((((.(((((.....))))).))))))......))))..))))))))........((((......))))......))).)))) ( -44.10, z-score = -1.54, R) >droYak2.chrX 567396 120 + 21770863 GAGCCGGAGGGCAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUCGUCAAUAAUAUGGCCAGGUCGAGGGCACCACUGCCCGGCUUCUCGUCAUUCGUCUGACAAUCAACGGAGUUC ...(((((((((.(.((((((.((..((((.((..((((((.(((((.....))))).)))))).)).)))).)).)))))).).)).))))((((......))))......)))..... ( -44.30, z-score = -1.51, R) >droEre2.scaffold_4644 605868 120 + 2521924 GAGCCGGAGGGCAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUCGUCAAUAAUAUGGCCAGGUCGAGGGCACCACUGCCCGGCUUCUCGUCGUUCGUCUGACAAUCAACUGAGUUC (((((((.((((((((((....)).((((......((((((.(((((.....))))).))))))......))))..))))))))........((((......))))......))).)))) ( -43.40, z-score = -1.15, R) >droAna3.scaffold_13117 678860 120 + 5790199 GAGCCGGAGGGCAGCGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUCGUCAAUAAUAUGGCCAGAUCGAGGGCACCGCUGCCCGGCUUCUCCUCGUUUGUCUGACAAUCAACCGAGUUC (((((...((((((((((....)).((((......((((((.(((((.....))))).))))))......))))..)))))))))))))...((((.(((.........))).))))... ( -45.90, z-score = -1.80, R) >dp4.chrXL_group1e 138850 120 + 12523060 GAGCCGGAGGGUAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUUGUCAAUAAUAUGGCCAGAUCGAGGGCACCACUGCCCGGCUUCUCGUCAUUUGUCUGACAAUCAACCGAAUUC (((((((..(((.((((..((((..(((.....)))(((((.(((((.....))))).)))))..))))...)))).)))..)))))))...((((......)))).............. ( -42.70, z-score = -1.92, R) >droPer1.super_60 207140 120 - 401158 GAGCCGGAGGGCAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUUGUCAAUAAUAUGGCCAGAUCGAGGGCACCACUGCCCGGCUUCUCGUCAUUUGUCUGACAAUCAACCGAAUUC (((((...((((((((((....)).((((......((((((.(((((.....))))).))))))......))))..)))))))))))))...((((......)))).............. ( -43.30, z-score = -1.83, R) >droWil1.scaffold_180777 2708637 120 + 4753960 GAGCCCGAGGGCAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUUGUCAAUAAUAUGGCCAGAUCGAGGGCACCACUGCCCGGUUUCUCAUCAUUUGUCUGACAAUCAACGGAAUUC ....(((.((((((((((....)).((((......((((((.(((((.....))))).))))))......))))..))))))))((((((.((.....))...)).))))..)))..... ( -40.80, z-score = -1.51, R) >droMoj3.scaffold_6359 2026044 120 - 4525533 GAGCCAGAGGGCAGCGCCGUAAUGAGCCUCAAUGGCGGCCAGAUCAUCGUCAAUAACAUGGCCCGAUCGCGGGCUCCAUUGCCCGGCUUCUCGUCAUUUGUUUGACAAUCAACUGAGUUC (((((((.(((.((((((((..((.....))...)))))..((((...((((......))))..)))).(((((......)))))))).)))((((......))))......))).)))) ( -42.90, z-score = -1.54, R) >droGri2.scaffold_15081 2380315 120 - 4274704 GAGCCGGAGGGCAGCGCCGUAAUGAGUCUCAAUGGUGGCCAGAUAAUUGUGAAUAAUAUGGCACGAUCACGGGCUCCAUUGCCCGGAUUCUCGUCAUUCGUUUGACGGUCAACUGAGUUG .((((((..(((.(.((((((.....((.((((.((......)).)))).))....)))))).)((((.(((((......)))))))))..(((((......))))))))..))).))). ( -38.50, z-score = -0.43, R) >droVir3.scaffold_12970 10594746 120 - 11907090 GAGCCGGAGGGCAGCGCAGUGAUGAGCCUCAAUGGCGGCCAGAUUAUUGUCAAUAAUAUGGCCAGAUCGAGGGCGCCACUGCCUGGCUUCUCCUCAUUUGUCUGACAAUCAACCGAAUUC (((..(((((.(((.((((((.((..((((.((...(((((.(((((.....))))).)))))..)).)))).)).))))))))).))))).)))(((((..((.....))..))))).. ( -44.10, z-score = -1.58, R) >anoGam1.chrX 10411598 120 - 22145176 GACCCCGAAGGUUCGGCGGUGAUGAGUCUGAACGGGGGCCAAAUUAUAAUGAACAAUAUGGCACGAUCGCGGGCGCCCUUACCUGGCUUCAGCUCCUUUGUGUGAGAAAUUUGUGAUUGC ..(((((.((..(((.......)))..))...)))))((((.(((.........))).)))).((((((((((((((.......)))....))))(((.....)))......))))))). ( -33.70, z-score = 0.57, R) >apiMel3.Group3 3425145 105 + 12341916 GAACCAGAAGGUAGUGCUGUUGUCAGUUUAAAUGGAGGACAAAUUAUUAUGAACAAUAUGGCAAGGUCAAGAGCACCAUUGCCUGGUUUCUCAUCAUUCGUAUAA--------------- (((((((..(((.(((((.(((((..((.....))..)))))................((((...))))..))))).)))..)))))))................--------------- ( -22.30, z-score = -0.04, R) >triCas2.ChLG9 3652548 111 + 15222296 GAACCGGAAGGUUCGGCG------AGUUUGAAUGGAGGACAGAUUAUCAUUAAUAACAUGGCGAGGUCAAGGGCGCCCCUACCCGGAUUUUCGUCGUUUGUUUAAUGA-UAGCUGAAU-- (((((....)))))....------...............(((.((((((((((....(((((((((....(((........)))....)))))))))....)))))))-))))))...-- ( -36.90, z-score = -2.40, R) >consensus GAGCCGGAGGGCAGUGCAGUGAUGAGCCUCAAUGGUGGCCAGAUUAUCGUCAAUAAUAUGGCCAGAUCGAGGGCACCACUGCCCGGCUUCUCGUCAUUUGUCUGACAAUCAACUGAGUUC (((((...(((((((((......).((((........((((.(((((.....))))).))))........))))..)))))))))))))............................... (-24.67 = -24.61 + -0.06)

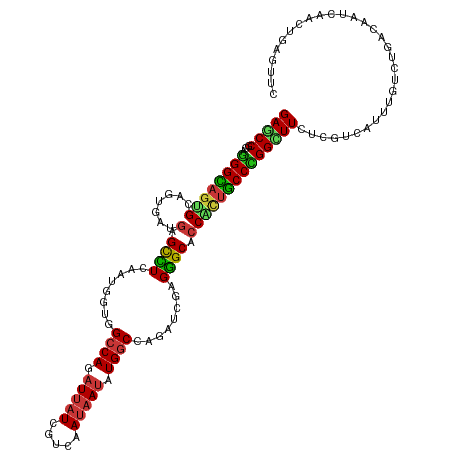
| Location | 641,559 – 641,679 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.35 |
| Shannon entropy | 0.42033 |
| G+C content | 0.52615 |
| Mean single sequence MFE | -42.63 |
| Consensus MFE | -16.19 |
| Energy contribution | -15.57 |
| Covariance contribution | -0.62 |
| Combinations/Pair | 1.61 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.918203 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 641559 120 - 22422827 GAACUCAGUUGAUUGUCAGACGAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGGCUC ............((((((......))))))..(((((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..)))))))))........)))))). ( -48.90, z-score = -4.03, R) >droSim1.chrX 528197 120 - 17042790 GAACUCAGUUGAUUGUCAGACGAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGGCUC ............((((((......))))))..(((((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..)))))))))........)))))). ( -48.90, z-score = -4.03, R) >droSec1.super_10 491131 120 - 3036183 GAACUCAGUUGAUUGUCAGACGAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGGCUC ............((((((......))))))..(((((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..)))))))))........)))))). ( -48.90, z-score = -4.03, R) >droYak2.chrX 567396 120 - 21770863 GAACUCCGUUGAUUGUCAGACGAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGGCUC .....(((((..((((((......))))))..)).)))(((((((((.((((((..((((((.((((((...)))))).))))))...))))))..)))))))))...(((....))).. ( -49.40, z-score = -4.28, R) >droEre2.scaffold_4644 605868 120 - 2521924 GAACUCAGUUGAUUGUCAGACGAACGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGGCUC ...(((.((((.(((.....))).))))))).(((((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..)))))))))........)))))). ( -50.70, z-score = -4.61, R) >droAna3.scaffold_13117 678860 120 - 5790199 GAACUCGGUUGAUUGUCAGACAAACGAGGAGAAGCCGGGCAGCGGUGCCCUCGAUCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCGCUGCCCUCCGGCUC ...((((.(((.((....))))).))))....(((((((..((((((((((((((.((((((.((((((...)))))).))))))..))))))).........)))))))..))))))). ( -48.80, z-score = -3.05, R) >dp4.chrXL_group1e 138850 120 - 12523060 GAAUUCGGUUGAUUGUCAGACAAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGAUCUGGCCAUAUUAUUGACAAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUACCCUCCGGCUC ....((((((..((((((......))))))..))))))(((((((((.(((((((.((((((.(((((.....))))).))))))..)))))))..)))))))))............... ( -51.00, z-score = -5.11, R) >droPer1.super_60 207140 120 + 401158 GAAUUCGGUUGAUUGUCAGACAAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGAUCUGGCCAUAUUAUUGACAAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGGCUC ....((((((..((((((......))))))..))))))(((((((((.(((((((.((((((.(((((.....))))).))))))..)))))))..)))))))))...(((....))).. ( -54.20, z-score = -5.48, R) >droWil1.scaffold_180777 2708637 120 - 4753960 GAAUUCCGUUGAUUGUCAGACAAAUGAUGAGAAACCGGGCAGUGGUGCCCUCGAUCUGGCCAUAUUAUUGACAAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCGGGCUC .....(((((..(..(((......)))..)..)).)))(((((((((.(((((((.((((((.(((((.....))))).))))))..)))))))..)))))))))...(((....))).. ( -49.40, z-score = -4.55, R) >droMoj3.scaffold_6359 2026044 120 + 4525533 GAACUCAGUUGAUUGUCAAACAAAUGACGAGAAGCCGGGCAAUGGAGCCCGCGAUCGGGCCAUGUUAUUGACGAUGAUCUGGCCGCCAUUGAGGCUCAUUACGGCGCUGCCCUCUGGCUC ...(((.((((.(((.....))).))))))).((((((((......))))(((.((((((((.((((((...)))))).)))))(((.....)))......))))))........)))). ( -42.60, z-score = -1.26, R) >droGri2.scaffold_15081 2380315 120 + 4274704 CAACUCAGUUGACCGUCAAACGAAUGACGAGAAUCCGGGCAAUGGAGCCCGUGAUCGUGCCAUAUUAUUCACAAUUAUCUGGCCACCAUUGAGACUCAUUACGGCGCUGCCCUCCGGCUC ...((((((.(..(((((......))))).((.(((((((......))))).))))(.((((...((........))..))))).).))))))............((((.....)))).. ( -30.70, z-score = -0.01, R) >droVir3.scaffold_12970 10594746 120 + 11907090 GAAUUCGGUUGAUUGUCAGACAAAUGAGGAGAAGCCAGGCAGUGGCGCCCUCGAUCUGGCCAUAUUAUUGACAAUAAUCUGGCCGCCAUUGAGGCUCAUCACUGCGCUGCCCUCCGGCUC ......((((..((.(((......))).))..))))((((((((..(.(((((((.((((((.(((((.....))))).))))))..)))))))..)..)))))).))(((....))).. ( -43.90, z-score = -1.74, R) >anoGam1.chrX 10411598 120 + 22145176 GCAAUCACAAAUUUCUCACACAAAGGAGCUGAAGCCAGGUAAGGGCGCCCGCGAUCGUGCCAUAUUGUUCAUUAUAAUUUGGCCCCCGUUCAGACUCAUCACCGCCGAACCUUCGGGGUC .........................(((((((((((((((...(((((........)))))(((........))).))))))).....))))).)))...(((.((((....))))))). ( -29.70, z-score = 0.16, R) >apiMel3.Group3 3425145 105 - 12341916 ---------------UUAUACGAAUGAUGAGAAACCAGGCAAUGGUGCUCUUGACCUUGCCAUAUUGUUCAUAAUAAUUUGUCCUCCAUUUAAACUGACAACAGCACUACCUUCUGGUUC ---------------....((((((.(((((......(((((.(((.(....)))))))))......)))))....))))))............(((....)))....(((....))).. ( -18.90, z-score = -0.43, R) >triCas2.ChLG9 3652548 111 - 15222296 --AUUCAGCUA-UCAUUAAACAAACGACGAAAAUCCGGGUAGGGGCGCCCUUGACCUCGCCAUGUUAUUAAUGAUAAUCUGUCCUCCAUUCAAACU------CGCCGAACCUUCCGGUUC --...(((.((-((((((((((..(((((......))(((((((....)))).))))))...)))..)))))))))..)))...............------....(((((....))))) ( -23.50, z-score = -0.40, R) >consensus GAACUCAGUUGAUUGUCAGACAAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGAUCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGGCUC ............................(((.....(((((.((..............((((.((((((...)))))).))))......(((......)))...)).))))).....))) (-16.19 = -15.57 + -0.62)

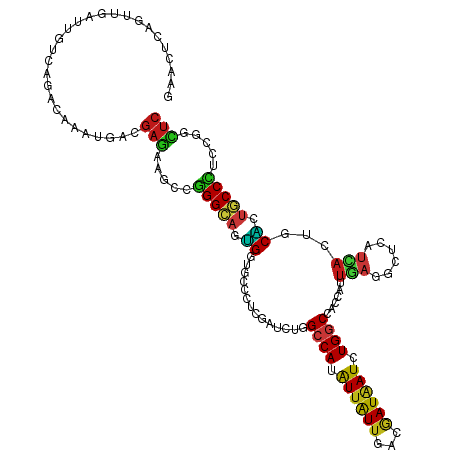
| Location | 641,562 – 641,682 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.93 |
| Shannon entropy | 0.42977 |
| G+C content | 0.51263 |
| Mean single sequence MFE | -40.33 |
| Consensus MFE | -14.91 |
| Energy contribution | -14.25 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.60 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.37 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.795574 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 641562 120 - 22422827 UUAGAACUCAGUUGAUUGUCAGACGAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGG ..........(((..((((((......))))))..)))((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..))))))))).)))........ ( -45.90, z-score = -3.62, R) >droSim1.chrX 528200 120 - 17042790 UUAGAACUCAGUUGAUUGUCAGACGAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGG ..........(((..((((((......))))))..)))((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..))))))))).)))........ ( -45.90, z-score = -3.62, R) >droSec1.super_10 491134 120 - 3036183 UUAGAACUCAGUUGAUUGUCAGACGAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGG ..........(((..((((((......))))))..)))((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..))))))))).)))........ ( -45.90, z-score = -3.62, R) >droYak2.chrX 567399 120 - 21770863 UUAGAACUCCGUUGAUUGUCAGACGAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGG ........(((((..((((((......))))))..)))((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..))))))))).)))......)) ( -46.80, z-score = -3.95, R) >droEre2.scaffold_4644 605871 120 - 2521924 UUAGAACUCAGUUGAUUGUCAGACGAACGACGAGAAGCCGGGCAGUGGUGCCCUCGACCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGG ......(((.((((.(((.....))).)))))))...(((((((((((((.((((((..((((((.((((((...)))))).))))))...))))))..)))))))))........)))) ( -45.50, z-score = -3.56, R) >droAna3.scaffold_13117 678863 120 - 5790199 UUAGAACUCGGUUGAUUGUCAGACAAACGAGGAGAAGCCGGGCAGCGGUGCCCUCGAUCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCGCUGCCCUCCGG .........((((..((.((........)).))..))))(((((((((((.(((((((.((((((.((((((...)))))).))))))..))))))).....)))..))))))))..... ( -46.50, z-score = -2.80, R) >dp4.chrXL_group1e 138853 120 - 12523060 UUAGAAUUCGGUUGAUUGUCAGACAAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGAUCUGGCCAUAUUAUUGACAAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUACCCUCCGG .(((...((((((..((((((......))))))..))))))(((((((((.(((((((.((((((.(((((.....))))).))))))..)))))))..))))))))).)))........ ( -51.60, z-score = -5.64, R) >droPer1.super_60 207143 120 + 401158 UUAGAAUUCGGUUGAUUGUCAGACAAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGAUCUGGCCAUAUUAUUGACAAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGG .(((...((((((..((((((......))))))..))))))(((((((((.(((((((.((((((.(((((.....))))).))))))..)))))))..))))))))).)))........ ( -51.30, z-score = -5.09, R) >droWil1.scaffold_180777 2708640 120 - 4753960 UUAGAAUUCCGUUGAUUGUCAGACAAAUGAUGAGAAACCGGGCAGUGGUGCCCUCGAUCUGGCCAUAUUAUUGACAAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCGGG ........(((((..(..(((......)))..)..)))((((((((((((.(((((((.((((((.(((((.....))))).))))))..)))))))..))))))))).))).....)). ( -46.10, z-score = -3.97, R) >droMoj3.scaffold_6359 2026047 120 + 4525533 UUAGAACUCAGUUGAUUGUCAAACAAAUGACGAGAAGCCGGGCAAUGGAGCCCGCGAUCGGGCCAUGUUAUUGACGAUGAUCUGGCCGCCAUUGAGGCUCAUUACGGCGCUGCCCUCUGG .((((.....(((..((((((......))))))..))).(((((..((...))(((.((((((((.((((((...)))))).)))))(((.....)))......))))))))))))))). ( -41.60, z-score = -1.40, R) >droGri2.scaffold_15081 2380318 120 + 4274704 UUACAACUCAGUUGACCGUCAAACGAAUGACGAGAAUCCGGGCAAUGGAGCCCGUGAUCGUGCCAUAUUAUUCACAAUUAUCUGGCCACCAUUGAGACUCAUUACGGCGCUGCCCUCCGG ......((((((.(..(((((......))))).((.(((((((......))))).))))(.((((...((........))..))))).).))))))........(((.(.....).))). ( -29.80, z-score = -0.23, R) >droVir3.scaffold_12970 10594749 120 + 11907090 UUAGAAUUCGGUUGAUUGUCAGACAAAUGAGGAGAAGCCAGGCAGUGGCGCCCUCGAUCUGGCCAUAUUAUUGACAAUAAUCUGGCCGCCAUUGAGGCUCAUCACUGCGCUGCCCUCCGG .........((((..((.(((......))).))..))))((((((((..(.(((((((.((((((.(((((.....))))).))))))..)))))))..)..)))))).))......... ( -40.70, z-score = -1.31, R) >anoGam1.chrX 10411601 120 + 22145176 UUGGCAAUCACAAAUUUCUCACACAAAGGAGCUGAAGCCAGGUAAGGGCGCCCGCGAUCGUGCCAUAUUGUUCAUUAUAAUUUGGCCCCCGUUCAGACUCAUCACCGCCGAACCUUCGGG (((((............((.......))(((((((((((((((...(((((........)))))(((........))).))))))).....))))).)))......))))).((....)) ( -29.90, z-score = 0.07, R) >apiMel3.Group3 3425148 102 - 12341916 ------------------UUAUACGAAUGAUGAGAAACCAGGCAAUGGUGCUCUUGACCUUGCCAUAUUGUUCAUAAUAAUUUGUCCUCCAUUUAAACUGACAACAGCACUACCUUCUGG ------------------....((((((.(((((......(((((.(((.(....)))))))))......)))))....))))))............(((....))).....((....)) ( -17.40, z-score = -0.37, R) >triCas2.ChLG9 3652551 108 - 15222296 -----AUUCAGCUA-UCAUUAAACAAACGACGAAAAUCCGGGUAGGGGCGCCCUUGACCUCGCCAUGUUAUUAAUGAUAAUCUGUCCUCCAUUCAAACU------CGCCGAACCUUCCGG -----...(((.((-((((((((((..(((((......))(((((((....)))).))))))...)))..)))))))))..)))...............------..(((.......))) ( -20.10, z-score = 0.42, R) >consensus UUAGAACUCAGUUGAUUGUCAGACAAAUGACGAGAAGCCGGGCAGUGGUGCCCUCGAUCUGGCCAUAUUAUUGACGAUAAUCUGGCCACCAUUGAGGCUCAUCACUGCACUGCCCUCCGG .......................................(((((.((..............((((.((((((...)))))).))))......(((......)))...)).)))))..... (-14.91 = -14.25 + -0.66)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:03:42 2011