
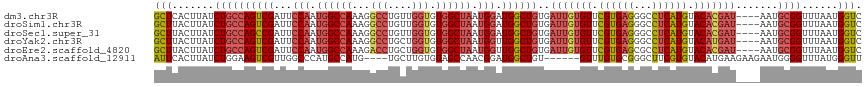
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 27,498,558 – 27,498,708 |
| Length | 150 |
| Max. P | 0.930944 |

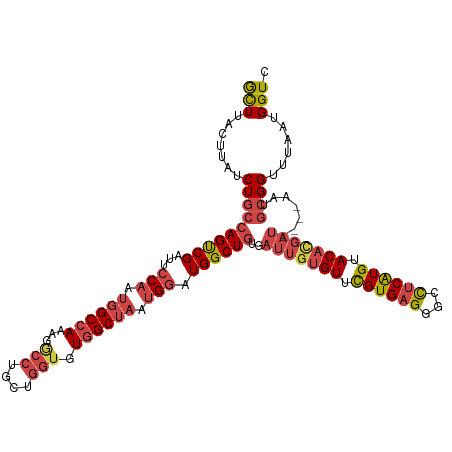
| Location | 27,498,558 – 27,498,672 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 85.72 |
| Shannon entropy | 0.26828 |
| G+C content | 0.49407 |
| Mean single sequence MFE | -40.48 |
| Consensus MFE | -28.39 |
| Energy contribution | -30.17 |
| Covariance contribution | 1.78 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.70 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.36 |
| SVM RNA-class probability | 0.930944 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 27498558 114 + 27905053 GCUCACUUAUCUGCCAGUCGAUUCCAAUGGCCAAAGGCCUGUUGGUGUGGCUAAUGGAUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACACGAU----AAUGCGGUUUAAUGGUC ..(((.(((.((((((((((..((((.((((((...(((....))).)))))).))))))))))..(((((((.((((((...)))))).)))))))----...))))..)))))).. ( -41.80, z-score = -2.49, R) >droSim1.chr3R 27118557 114 + 27517382 GCUUACUUAUCUGCCAGUCGAUUCCAAUGGCCAAAGGCCUGUUGGUGUGGCUAAUGGAUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACACGAU----AAUGCGGUUUAAUGGUC (((.......((((((((((..((((.((((((...(((....))).)))))).))))))))))..(((((((.((((((...)))))).)))))))----...))))......))). ( -42.12, z-score = -2.86, R) >droSec1.super_31 158198 114 + 568655 GCUUACUUAUCUGCCAGCCGAUUCCAAUGGCCAAAGGCCUGUUGGUGUGGCUAAUGGAUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACACGAU----AAUGCGGUUUAAUGGUC (((.......((((((((((..((((.((((((...(((....))).)))))).))))))))))..(((((((.((((((...)))))).)))))))----...))))......))). ( -44.82, z-score = -3.31, R) >droYak2.chr3R 28435969 114 + 28832112 GCUUACUUAUCUGCCAGUCGAUUCCAAUGGCCAAAGGCCUGCUGGUGUGGCUAAUGGUUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACAUGAU----AAUGCGGUUUAAUGGUC (((.......(((((((((((..(((.((((((...(((....))).)))))).))))))))))..((..(((.((((((...)))))).)))..))----...))))......))). ( -39.12, z-score = -1.87, R) >droEre2.scaffold_4820 9993836 114 - 10470090 GCUUACUUAUCUGCCAGUCGAUUCCAAUGGCCAAAGACCUGCUGGUGUGGCUAAUGGUUGGCUGUGAUUGUGUUCGUGAGCGCCUCAUGUACACGAU----AAUGCGGUUUAAUGGUC (((.......(((((((((((..(((.((((((...(((....))).)))))).))))))))))..(((((((.((((((...)))))).)))))))----...))))......))). ( -41.22, z-score = -2.91, R) >droAna3.scaffold_12911 1768764 108 - 5364042 AUUCACUUAUCUGGAAGUCGUUGGCCCAUGCCAUG----UGCUUGUGUAGCCAACGGAUGGCUGU------GUUUGUGCGGGCUUCGUGUACAUGAAGAAGAAUGGGGUUUAUGGGUU ..............((.((((..((((..(((.((----..(..(..((((((.....)))))).------.)..)..)))))(((((....)))))........))))..)))).)) ( -33.80, z-score = -0.75, R) >consensus GCUUACUUAUCUGCCAGUCGAUUCCAAUGGCCAAAGGCCUGCUGGUGUGGCUAAUGGAUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACACGAU____AAUGCGGUUUAAUGGUC (((.......((((((((((...(((.((((((...(((....))).)))))).))).))))))..(((((((.((((((...)))))).))))))).......))))......))). (-28.39 = -30.17 + 1.78)
| Location | 27,498,598 – 27,498,708 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.35 |
| Shannon entropy | 0.28497 |
| G+C content | 0.44496 |
| Mean single sequence MFE | -33.13 |
| Consensus MFE | -22.71 |
| Energy contribution | -23.38 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.69 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.571368 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 27498598 110 + 27905053 --GUUGGUGUGGCUAAUGGAUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACACGAU----AAUGCGGUUUAAUGGUCUGAGCUACGCAA----AUAGACUGUUCAAUUCUAUCUUAC --....((((((((...((((((((((.(((((((.((((((...)))))).)))))))----...)))))).....)))).))))))))..----(((((.(.....).)))))..... ( -34.10, z-score = -2.23, R) >droSim1.chr3R 27118597 110 + 27517382 --GUUGGUGUGGCUAAUGGAUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACACGAU----AAUGCGGUUUAAUGGUCUGAGCUACGCAA----AUAGACUGUUCAAUUCUAUCUUAU --....((((((((...((((((((((.(((((((.((((((...)))))).)))))))----...)))))).....)))).))))))))..----(((((.(.....).)))))..... ( -34.10, z-score = -2.23, R) >droSec1.super_31 158238 110 + 568655 --GUUGGUGUGGCUAAUGGAUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACACGAU----AAUGCGGUUUAAUGGUCUGAGCUACGCAA----AUAGACUGUUCAAUUCUAUCUUAU --....((((((((...((((((((((.(((((((.((((((...)))))).)))))))----...)))))).....)))).))))))))..----(((((.(.....).)))))..... ( -34.10, z-score = -2.23, R) >droYak2.chr3R 28436009 110 + 28832112 --GCUGGUGUGGCUAAUGGUUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACAUGAU----AAUGCGGUUUAAUGGUCUGAGCUACGCAA----AUAGACUGUUCAAUUCCAUCUUAC --....((((((((.((.(((((((((.((..(((.((((((...)))))).)))..))----...)))).))))).))...))))))))..----........................ ( -31.20, z-score = -0.88, R) >droEre2.scaffold_4820 9993876 110 - 10470090 --GCUGGUGUGGCUAAUGGUUGGCUGUGAUUGUGUUCGUGAGCGCCUCAUGUACACGAU----AAUGCGGUUUAAUGGUCUGAGCUACGCAA----AUAGACUGUUCAAUUCUAUCUUAC --....((((((((.((.(((((((((.(((((((.((((((...)))))).)))))))----...)))).))))).))...))))))))..----(((((.(.....).)))))..... ( -34.50, z-score = -2.03, R) >droAna3.scaffold_12911 1768798 113 - 5364042 GUGCUUGUGUAGCCAACGGAUGGCUGUGUUUGUGC------GGGCUUCGUGUACAUGAAGAAGAAUGGGGUUUAUGGGUUCCAACUUCAUCAUAUUAUAGGGUUUUCAACCUUUUCCUA- (..(..(..((((((.....))))))..)..)..)------(((..((.((((.((((.((((..(((..((....))..))).)))).))))..)))).))..)))............- ( -30.80, z-score = -0.48, R) >consensus __GCUGGUGUGGCUAAUGGAUGGCUGUGAUUGUGUUCGUGAGGGCCUCAUGUACACGAU____AAUGCGGUUUAAUGGUCUGAGCUACGCAA____AUAGACUGUUCAAUUCUAUCUUAC ......((((((((...((((((((((((((((((.((((((...)))))).)))))))......))))))).....)))).)))))))).............................. (-22.71 = -23.38 + 0.67)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:00:26 2011