
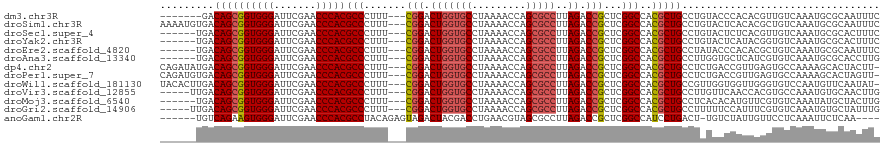
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 26,074,338 – 26,074,448 |
| Length | 110 |
| Max. P | 0.997905 |

| Location | 26,074,338 – 26,074,448 |
|---|---|
| Length | 110 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.75 |
| Shannon entropy | 0.38960 |
| G+C content | 0.57271 |
| Mean single sequence MFE | -35.20 |
| Consensus MFE | -26.88 |
| Energy contribution | -26.47 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.18 |
| Mean z-score | -0.95 |
| Structure conservation index | 0.76 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.563825 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 26074338 110 + 27905053 -------GACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUGUACCCACACGUUGUCAAAUGCGCAAUUUC -------((((((((((((............(((....---(((.(((((((.........)))))..)).)))...)))...((.....)).))))).))))))).............. ( -34.70, z-score = -1.01, R) >droSim1.chr3R 25740612 117 + 27517382 AAAAUGUGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUGUACUCACACGCUGUCAAAUGCGCAAUUUC ......(((((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))...................))))))))............. ( -36.20, z-score = -1.02, R) >droSec1.super_4 4924914 111 + 6179234 ------UGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUGUACUCUCACGUUGUCAAAUGCGCACUUUC ------(((((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))...................))))))))............. ( -33.60, z-score = -0.57, R) >droYak2.chr3R 27008267 111 + 28832112 ------UGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUGUACUCAUACGGUGUCAAAUGCGCACUUUC ------(((((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))..))))(((.(((....))))))))))............. ( -34.30, z-score = -0.41, R) >droEre2.scaffold_4820 8548481 111 - 10470090 ------UGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUAUACCCACACGCUGUCAAAUGCGCAAUUUC ------(((((((((((((...................---(((.(((((((.........)))))..)).)))...(((......)))....))))).))))))))............. ( -37.40, z-score = -2.19, R) >droAna3.scaffold_13340 21115470 111 + 23697760 ------UGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUUGGUGCUCAUCGUGUCAAAUGCGCACCUUG ------...((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))..)))))....(((((.(((........))).)))))... ( -39.90, z-score = -1.53, R) >dp4.chr2 29360400 116 - 30794189 CAGAUAUGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUCUGACCGUUGAGUGCCAAAAGCACUACUU- ((((...(.((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))..)))))).)))).......(((((.....)))))....- ( -38.70, z-score = -1.89, R) >droPer1.super_7 211230 116 - 4445127 CAGAUGUGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUCUGACCGUUGAGUGCCAAAAGCACUAGUU- ((((.(.(.((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))..))))))))))).......(((((.....)))))....- ( -39.90, z-score = -1.37, R) >droWil1.scaffold_181130 9554707 116 + 16660200 UACACUUGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCGUUGGUGGUUGGGUGUCCAAUGUUCAAUAU- .....(((((((((.((((.......))))))).....---.((((((((.......)))))(((((..(((((((((((......))))..)))))))))))))))..)).))))...- ( -40.90, z-score = -0.81, R) >droVir3.scaffold_12855 3169213 112 + 10161210 -----UUGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUUGUUCAACCACGUGCCAAAUGUGCAACUUG -----....((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))..)))))...........(((((.....)))))....... ( -33.50, z-score = -0.48, R) >droMoj3.scaffold_6540 24115909 111 - 34148556 ------UGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUCACACAUGUUCGUGUCAAAUAUGCUACUUG ------...((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))..)))))....((((......))))............... ( -32.40, z-score = -0.68, R) >droGri2.scaffold_14906 5859175 112 + 14172833 -----UUGACAGCGGUGGGAUUCGAACCCACGCCCUUU---CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUUUUUCCAUUUCGUGUCAAAUGUGCUAUUUG -----....((((((((((.......)))))(((....---(((.(((((((.........)))))..)).)))...)))..)))))........(((((......)))))......... ( -30.50, z-score = -0.22, R) >anoGam1.chr2R 37003837 109 - 62725911 ------UGUCAGAAGUGGGAUUCGAACCCACGCCUACAGAGUAGACUACGACCUGAACGUAGCGCCUUAGACCGCUCGGCCAUCCUGACU-UGUCUAUUGUUCCUCAAAUUCUCAA---- ------.(((((..(((((.......)))))(((..(((.((........)))))...(.((((........))))))))....))))).-.........................---- ( -25.60, z-score = -0.22, R) >consensus ______UGACAGCGGUGGGAUUCGAACCCACGCCCUUU___CGGACUGGUGCCUAAAACCAGCGCCUUAGACCGCUCGGCCACGCUGCCUGUACCCACACGGUGUCAAAUGCGCAAUUUC .........((((((((((.......)))))(((........((.(((((.......)))))..))...((....)))))..)))))................................. (-26.88 = -26.47 + -0.41)


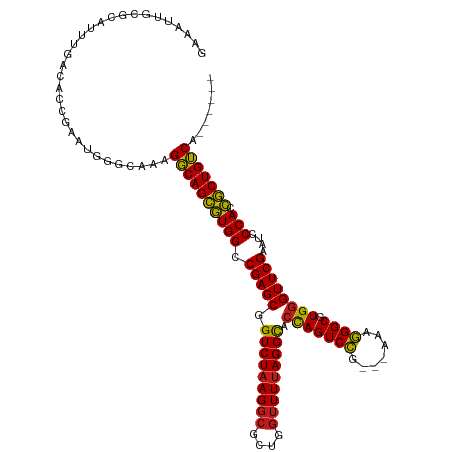
| Location | 26,074,338 – 26,074,448 |
|---|---|
| Length | 110 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.75 |
| Shannon entropy | 0.38960 |
| G+C content | 0.57271 |
| Mean single sequence MFE | -48.00 |
| Consensus MFE | -38.42 |
| Energy contribution | -38.02 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.87 |
| Structure conservation index | 0.80 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.21 |
| SVM RNA-class probability | 0.997905 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 26074338 110 - 27905053 GAAAUUGCGCAUUUGACAACGUGUGGGUACAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUC------- .....(((.(((.((....)).))).)))..((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).)))))))------- ( -45.80, z-score = -2.04, R) >droSim1.chr3R 25740612 117 - 27517382 GAAAUUGCGCAUUUGACAGCGUGUGAGUACAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCACAUUUU .............((((((((.(((......(((......)))((((.(((((((((....))))))))).(((((((.---...)))).)))))))......)))))))))))...... ( -48.70, z-score = -2.70, R) >droSec1.super_4 4924914 111 - 6179234 GAAAGUGCGCAUUUGACAACGUGAGAGUACAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCA------ ....((((.(((........)))...)))).((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).))))))).------ ( -48.50, z-score = -2.97, R) >droYak2.chr3R 27008267 111 - 28832112 GAAAGUGCGCAUUUGACACCGUAUGAGUACAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCA------ ....((((.(((.((....)).))).)))).((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).))))))).------ ( -49.90, z-score = -3.29, R) >droEre2.scaffold_4820 8548481 111 + 10470090 GAAAUUGCGCAUUUGACAGCGUGUGGGUAUAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCA------ .............((((((((.(((((....(((......)))((((.(((((((((....))))))))).(((((((.---...)))).)))))))....)))))))))))))------ ( -54.10, z-score = -4.22, R) >droAna3.scaffold_13340 21115470 111 - 23697760 CAAGGUGCGCAUUUGACACGAUGAGCACCAAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCA------ ...(((((.((((......)))).)))))..((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).))))))).------ ( -55.60, z-score = -4.45, R) >dp4.chr2 29360400 116 + 30794189 -AAGUAGUGCUUUUGGCACUCAACGGUCAGAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCAUAUCUG -..(.(((((.....))))))......((((((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).)))))))...)))) ( -51.00, z-score = -3.32, R) >droPer1.super_7 211230 116 + 4445127 -AACUAGUGCUUUUGGCACUCAACGGUCAGAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCACAUCUG -....(((((.....))))).......((((((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).)))))))...)))) ( -50.30, z-score = -2.82, R) >droWil1.scaffold_181130 9554707 116 - 16660200 -AUAUUGAACAUUGGACACCCAACCACCAACGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCAAGUGUA -.......(((((((....))..........((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).))))))).))))). ( -47.10, z-score = -2.37, R) >droVir3.scaffold_12855 3169213 112 - 10161210 CAAGUUGCACAUUUGGCACGUGGUUGAACAAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCAA----- ...........((..((.....))..))...((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).)))))))..----- ( -45.80, z-score = -1.90, R) >droMoj3.scaffold_6540 24115909 111 + 34148556 CAAGUAGCAUAUUUGACACGAACAUGUGUGAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCA------ ((((((...))))))((((......))))..((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).))))))).------ ( -47.40, z-score = -3.11, R) >droGri2.scaffold_14906 5859175 112 - 14172833 CAAAUAGCACAUUUGACACGAAAUGGAAAAAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG---AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCAA----- .........(((((......)))))......((((((((((.(((((.(((((((((....))))))))).(((((((.---...)))).))))))))....))).)))))))..----- ( -45.50, z-score = -3.22, R) >anoGam1.chr2R 37003837 109 + 62725911 ----UUGAGAAUUUGAGGAACAAUAGACA-AGUCAGGAUGGCCGAGCGGUCUAAGGCGCUACGUUCAGGUCGUAGUCUACUCUGUAGGCGUGGGUUCGAAUCCCACUUCUGACA------ ----.........................-.(((((((((((((((((.............))))).)))))).((((((...))))))(((((.......))))).)))))).------ ( -34.32, z-score = -0.85, R) >consensus GAAAUUGCGCAUUUGACACCGAAUGGGCAAAGGCAGCGUGGCCGAGCGGUCUAAGGCGCUGGUUUUAGGCACCAGUCCG___AAAGGGCGUGGGUUCGAAUCCCACCGCUGUCA______ ...............................((((((((((.(((((.(((((((((....))))))))).(((((((.......)))).))))))))....))).)))))))....... (-38.42 = -38.02 + -0.40)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:56:51 2011