
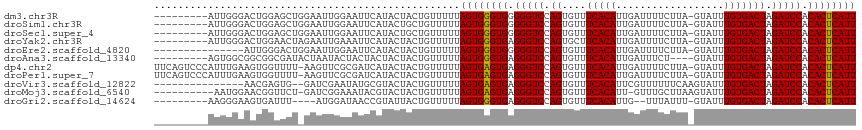
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 25,041,285 – 25,041,414 |
| Length | 129 |
| Max. P | 0.998337 |

| Location | 25,041,285 – 25,041,392 |
|---|---|
| Length | 107 |
| Sequences | 11 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 79.66 |
| Shannon entropy | 0.42926 |
| G+C content | 0.39109 |
| Mean single sequence MFE | -28.85 |
| Consensus MFE | -20.13 |
| Energy contribution | -19.90 |
| Covariance contribution | -0.23 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.70 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.43 |
| SVM RNA-class probability | 0.939453 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 25041285 107 + 27905053 ---------AUUGGGACUGGAGCUGGAAUUGGAAUUCAUACUACUGUUUUUAGUGGGUGGGGGUCCAGUGUUUCACAUUGAUUUUCUUA-GUAUUUGUGACUAGAUCCACACUCAUU ---------..((((..((((.((((.((((((.(((((.((((((....)))))))))))..))))))...(((((...(((.....)-))...))))))))).))))..)))).. ( -32.60, z-score = -2.23, R) >droSim1.chr3R 24712176 107 + 27517382 ---------AUUGGGACUGGAGCUGGAAUUGGAAUUCAUACUGCUGUUUUUAGUGGGUGGGGGUCCAGUGUUUCACAUUGAUUUUCUUA-GUAUUUGUGACUAGAUCCACACUCAUU ---------..((((..((((.((((.((((((.(((((.(..(((....)))..))))))..))))))...(((((...(((.....)-))...))))))))).))))..)))).. ( -31.70, z-score = -1.77, R) >droSec1.super_4 3904065 107 + 6179234 ---------AUUGGGACUGGAGCUGGAAUUGGAAUUCAUACUGCUGUUUUUAGUGGGUGGGGGUCCAGUGUUUCACAUUGAUUUUCUUA-GUAUUUGUGACUAGAUCCACACUCAUU ---------..((((..((((.((((.((((((.(((((.(..(((....)))..))))))..))))))...(((((...(((.....)-))...))))))))).))))..)))).. ( -31.70, z-score = -1.77, R) >droYak2.chr3R 7375940 107 - 28832112 ---------AUUGGGACUGGAACUAGAAUUGAAAUUCAUACUACUGUUUUUAGUGGGUGAGGGUCCAGUGCUUCACAUUGAUUUUCUUA-GUAUUUGUGACUAGAUCCACACUCAUU ---------..((((..((((.((((....((..(((((.((((((....)))))))))))..)).......(((((...(((.....)-))...))))))))).))))..)))).. ( -28.40, z-score = -1.51, R) >droEre2.scaffold_4820 7480911 101 - 10470090 ---------------AUUGGGACUGGAAUUGGAAUUCAUACUACUGUUUUUAGUGGGUGGGGGUCCAGUGUUUCACAUUGAUUUUCUUA-GUAUUUGUGACUAGAUCCACACUCAUU ---------------..((((.((((.((((((.(((((.((((((....)))))))))))..))))))...(((((...(((.....)-))...))))))))).))))........ ( -28.30, z-score = -1.67, R) >droAna3.scaffold_13340 9497332 104 - 23697760 ---------AGUGGCGGCGGCGAUACUAAUACUACUACUACUACUGUUUUUAGUGGGUGAGGGUCCAGUGUUUCACAUUGAUUUCU----GUAUUUGUGACUAGAUCCACACUCAUU ---------(((((..(..(...........)..)..))))).........((((((((.(((((.((....(((((...((....----))...))))))).))))).)))))))) ( -25.90, z-score = -0.46, R) >dp4.chr2 1881884 115 + 30794189 UUCAGUCCCAUUUGAAGUGGUUUU-AAGUUCGCGAUCAUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUUGAUUUUCUUA-GUAUUUGUGACUAGAUCCACACUCAUU ..((((......(((.((((....-....))))..)))....)))).....((((((((.(((((.((....(((((...(((.....)-))...))))))).))))).)))))))) ( -28.60, z-score = -1.93, R) >droPer1.super_7 2045282 115 + 4445127 UUCAGUCCCAUUUGAAGUGGUUUU-AAGUUCGCGAUCAUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUUGAUUUUCUUA-GUAUUUGUGACUAGAUCCACACUCAUU ..((((......(((.((((....-....))))..)))....)))).....((((((((.(((((.((....(((((...(((.....)-))...))))))).))))).)))))))) ( -28.60, z-score = -1.93, R) >droVir3.scaffold_12822 2330905 100 + 4096053 ---------------AACGAGUG--GAUCGAAUAUGCGUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUUCGUUUUUUCAAGUAUUUGUGACUAGAUCCACACUCAUU ---------------(((.((((--(..((......))..))))))))...((((((((.(((((.((....(((((..................))))))).))))).)))))))) ( -25.67, z-score = -1.43, R) >droMoj3.scaffold_6540 26447111 105 + 34148556 ----------AAUGGAACGGUUCU-GAUCGGAAAUACGUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUU-GUUUGCUUAAGUAUUUGUGACUAGAUCCACACUCAUU ----------...((((((((...-...((......))....)))))))).((((((((.(((((.((....(((((..-...(((....)))..))))))).))))).)))))))) ( -29.40, z-score = -1.83, R) >droGri2.scaffold_14624 3249725 101 + 4233967 ---------AAGGGAAGUGAUUU----AUGGAUAACCGUAUUACUGUUUUUAGUGGGUGAGGGUCCAGUGUUUCACAUUG--UUUAUUU-GUAUUUGUGACUAGAUCCACACUCAUU ---------(((((.(((((..(----((((....)))))))))).)))))((((((((.(((((.((....(((((...--.......-.....))))))).))))).)))))))) ( -26.46, z-score = -1.55, R) >consensus _________AUUGGGACUGGAUCU_GAAUUGGAAUUCAUACUACUGUUUUUAGUGGGUGAGGGUCCAGUGUUUCACAUUGAUUUUCUUA_GUAUUUGUGACUAGAUCCACACUCAUU ...................................................((((((((.(((((.((....(((((..................))))))).))))).)))))))) (-20.13 = -19.90 + -0.23)
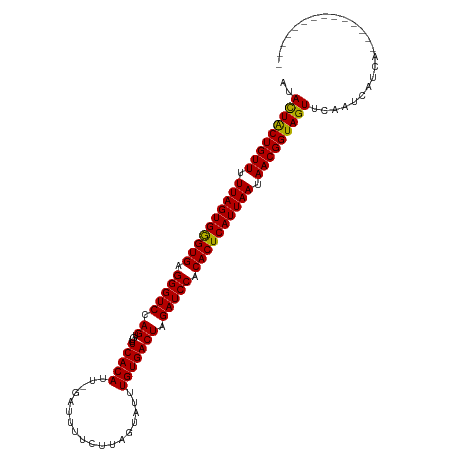
| Location | 25,041,313 – 25,041,414 |
|---|---|
| Length | 101 |
| Sequences | 12 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 90.00 |
| Shannon entropy | 0.20536 |
| G+C content | 0.36461 |
| Mean single sequence MFE | -37.04 |
| Consensus MFE | -35.36 |
| Energy contribution | -34.90 |
| Covariance contribution | -0.46 |
| Combinations/Pair | 1.10 |
| Mean z-score | -5.73 |
| Structure conservation index | 0.95 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.33 |
| SVM RNA-class probability | 0.998337 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 25041313 101 + 27905053 AUACUACUGUUUUUAGUGGGUGGGGGUCCAGUGUUUCACAUU-GAUUUUCUUAGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAUCA-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.(((.....)))...))))))).))))).)))))))))).)))))))))..........-------------- ( -36.40, z-score = -5.60, R) >droSim1.chr3R 24712204 101 + 27517382 AUACUGCUGUUUUUAGUGGGUGGGGGUCCAGUGUUUCACAUU-GAUUUUCUUAGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAUCA-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.(((.....)))...))))))).))))).)))))))))).)))))))))..........-------------- ( -36.40, z-score = -5.41, R) >droSec1.super_4 3904093 101 + 6179234 AUACUGCUGUUUUUAGUGGGUGGGGGUCCAGUGUUUCACAUU-GAUUUUCUUAGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAUCA-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.(((.....)))...))))))).))))).)))))))))).)))))))))..........-------------- ( -36.40, z-score = -5.41, R) >droYak2.chr3R 7375968 101 - 28832112 AUACUACUGUUUUUAGUGGGUGAGGGUCCAGUGCUUCACAUU-GAUUUUCUUAGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAUCA-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.(((.....)))...))))))).))))).)))))))))).)))))))))..........-------------- ( -36.40, z-score = -5.62, R) >droEre2.scaffold_4820 7480933 101 - 10470090 AUACUACUGUUUUUAGUGGGUGGGGGUCCAGUGUUUCACAUU-GAUUUUCUUAGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAUCA-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.(((.....)))...))))))).))))).)))))))))).)))))))))..........-------------- ( -36.40, z-score = -5.60, R) >droAna3.scaffold_13340 9497360 98 - 23697760 CUACUACUGUUUUUAGUGGGUGAGGGUCCAGUGUUUCACAUU-GAUUUCU---GUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAGCA-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.((....---))...))))))).))))).)))))))))).)))))))))..........-------------- ( -35.80, z-score = -4.95, R) >dp4.chr2 1881920 101 + 30794189 AUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUU-GAUUUUCUUAGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAACA-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.(((.....)))...))))))).))))).)))))))))).)))))))))..........-------------- ( -37.30, z-score = -5.97, R) >droPer1.super_7 2045318 101 + 4445127 AUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUU-GAUUUUCUUAGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAACA-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.(((.....)))...))))))).))))).)))))))))).)))))))))..........-------------- ( -37.30, z-score = -5.97, R) >droWil1.scaffold_181108 1734361 100 + 4707319 CUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUU-AAAUAUUU-AGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAUCG-------------- ..(((((((((.((((((((((.(((((.((....(((((..-.(((((..-.)))))))))))).))))).)))))))))).)))))))))..........-------------- ( -36.00, z-score = -6.26, R) >droVir3.scaffold_12822 2330925 106 + 4096053 GUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUUCGUUUUUUCAAGUAUUUGUGACUAGAUCCACACUCAUUAACAACGGUAGUUCAAUCAGCAAAUA---------- (.(((((((((.((((((((((.(((((.((....(((((..................))))))).))))).)))))))))).))))))))).)............---------- ( -36.27, z-score = -5.77, R) >droMoj3.scaffold_6540 26447137 97 + 34148556 GUACUACUGUUUUUAGUGAGUGAGGGUCCAGUGUUUCACAUU-GUUUGCUUAAGUAUUUGUGACUAGAUCCACACUCAUUAAAAACGGUAGUUCAAUC------------------ (.((((((((((((((((((((.(((((.((....(((((..-...(((....)))..))))))).))))).)))))))))))))))))))).)....------------------ ( -41.70, z-score = -7.45, R) >droGri2.scaffold_14624 3249749 113 + 4233967 GUAUUACUGUUUUUAGUGGGUGAGGGUCCAGUGUUUCACAUU-GUUUAUUU--GUAUUUGUGACUAGAUCCACACUCAUUAAAAACGGUAGUUCAAUCAACAGUUAUUGCAUGACA (.((((((((((((((((((((.(((((.((....(((((..-........--.....))))))).))))).)))))))))))))))))))).)........(((((...))))). ( -38.06, z-score = -4.73, R) >consensus AUACUACUGUUUUUAGUGGGUGAGGGUCCAGUGUUUCACAUU_GAUUUUCUUAGUAUUUGUGACUAGAUCCACACUCAUUAAUAACGGUAGUUCAAUCAUCA______________ ..(((((((((.((((((((((.(((((.((....(((((..................))))))).))))).)))))))))).)))))))))........................ (-35.36 = -34.90 + -0.46)
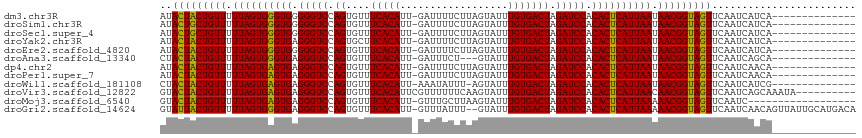
| Location | 25,041,313 – 25,041,414 |
|---|---|
| Length | 101 |
| Sequences | 12 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 90.00 |
| Shannon entropy | 0.20536 |
| G+C content | 0.36461 |
| Mean single sequence MFE | -25.49 |
| Consensus MFE | -22.37 |
| Energy contribution | -22.62 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.18 |
| Structure conservation index | 0.88 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.98 |
| SVM RNA-class probability | 0.996771 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 25041313 101 - 27905053 --------------UGAUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACUAAGAAAAUC-AAUGUGAAACACUGGACCCCCACCCACUAAAAACAGUAGUAU --------------..........(((((.(((.(((.((.(((.((.((((((((((...............-..)))))....))))).)).))).)).))).))).))))).. ( -23.13, z-score = -2.85, R) >droSim1.chr3R 24712204 101 - 27517382 --------------UGAUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACUAAGAAAAUC-AAUGUGAAACACUGGACCCCCACCCACUAAAAACAGCAGUAU --------------..........(((.(.(((.(((.((.(((.((.((((((((((...............-..)))))....))))).)).))).)).))).))).).))).. ( -18.53, z-score = -1.33, R) >droSec1.super_4 3904093 101 - 6179234 --------------UGAUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACUAAGAAAAUC-AAUGUGAAACACUGGACCCCCACCCACUAAAAACAGCAGUAU --------------..........(((.(.(((.(((.((.(((.((.((((((((((...............-..)))))....))))).)).))).)).))).))).).))).. ( -18.53, z-score = -1.33, R) >droYak2.chr3R 7375968 101 + 28832112 --------------UGAUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACUAAGAAAAUC-AAUGUGAAGCACUGGACCCUCACCCACUAAAAACAGUAGUAU --------------..........(((((.(((.(((.((.(((.((.((((((((((...............-..)))))....))))).)).))).)).))).))).))))).. ( -23.93, z-score = -2.65, R) >droEre2.scaffold_4820 7480933 101 + 10470090 --------------UGAUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACUAAGAAAAUC-AAUGUGAAACACUGGACCCCCACCCACUAAAAACAGUAGUAU --------------..........(((((.(((.(((.((.(((.((.((((((((((...............-..)))))....))))).)).))).)).))).))).))))).. ( -23.13, z-score = -2.85, R) >droAna3.scaffold_13340 9497360 98 + 23697760 --------------UGCUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUAC---AGAAAUC-AAUGUGAAACACUGGACCCUCACCCACUAAAAACAGUAGUAG --------------..........(((((.(((.(((.((.(((.((.((((((((((.....---.......-..)))))....))))).)).))).)).))).))).))))).. ( -24.14, z-score = -2.59, R) >dp4.chr2 1881920 101 - 30794189 --------------UGUUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACUAAGAAAAUC-AAUGUGAAACACUGGACCCUCACUCACUAAAAACAGUAGUAU --------------..........(((((.(((.(((.((((((.((.((((((((((...............-..)))))....))))).)).)))))).))).))).))))).. ( -28.73, z-score = -3.96, R) >droPer1.super_7 2045318 101 - 4445127 --------------UGUUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACUAAGAAAAUC-AAUGUGAAACACUGGACCCUCACUCACUAAAAACAGUAGUAU --------------..........(((((.(((.(((.((((((.((.((((((((((...............-..)))))....))))).)).)))))).))).))).))))).. ( -28.73, z-score = -3.96, R) >droWil1.scaffold_181108 1734361 100 - 4707319 --------------CGAUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACU-AAAUAUUU-AAUGUGAAACACUGGACCCUCACUCACUAAAAACAGUAGUAG --------------..........(((((.(((.(((.((((((.((.((((((((((((((..-...)))).-..)))))....))))).)).)))))).))).))).))))).. ( -29.10, z-score = -4.54, R) >droVir3.scaffold_12822 2330925 106 - 4096053 ----------UAUUUGCUGAUUGAACUACCGUUGUUAAUGAGUGUGGAUCUAGUCACAAAUACUUGAAAAAACGAAUGUGAAACACUGGACCCUCACUCACUAAAAACAGUAGUAC ----------..............(((((.(((.(((.((((((.((.((((((((((.....((....)).....)))))....))))).)).)))))).))).))).))))).. ( -29.80, z-score = -4.08, R) >droMoj3.scaffold_6540 26447137 97 - 34148556 ------------------GAUUGAACUACCGUUUUUAAUGAGUGUGGAUCUAGUCACAAAUACUUAAGCAAAC-AAUGUGAAACACUGGACCCUCACUCACUAAAAACAGUAGUAC ------------------......(((((.(((((((.((((((.((.((((((((((...............-..)))))....))))).)).)))))).))))))).))))).. ( -32.23, z-score = -5.48, R) >droGri2.scaffold_14624 3249749 113 - 4233967 UGUCAUGCAAUAACUGUUGAUUGAACUACCGUUUUUAAUGAGUGUGGAUCUAGUCACAAAUAC--AAAUAAAC-AAUGUGAAACACUGGACCCUCACCCACUAAAAACAGUAAUAC ..(((..((((....))))..)))..(((.(((((((.((.(((.((.((((((((((.....--........-..)))))....))))).)).))).)).))))))).))).... ( -25.96, z-score = -2.49, R) >consensus ______________UGAUGAUUGAACUACCGUUAUUAAUGAGUGUGGAUCUAGUCACAAAUACUAAGAAAAUC_AAUGUGAAACACUGGACCCUCACCCACUAAAAACAGUAGUAU ........................(((((.(((.(((.((.(((.((.((((((((((..................)))))....))))).)).))).)).))).))).))))).. (-22.37 = -22.62 + 0.25)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:53:50 2011