
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 24,925,522 – 24,925,625 |
| Length | 103 |
| Max. P | 0.607217 |

| Location | 24,925,522 – 24,925,625 |
|---|---|
| Length | 103 |
| Sequences | 13 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 67.88 |
| Shannon entropy | 0.71551 |
| G+C content | 0.57869 |
| Mean single sequence MFE | -40.48 |
| Consensus MFE | -9.21 |
| Energy contribution | -9.65 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.72 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.23 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.607217 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 24925522 103 - 27905053 UCGUCAUCGUCUCCGGUGAUACCGCAA--AACUUCAUCACAUCGCCG---AGAUCCAGCUUUCCAUUGCUACUUCGCUGGAUCGUCGUUGGGGGUGGCGAUGGAGACU---- ..(((((((....))))))).......--..(((((((.((((.(((---(((((((((................))))))))....)))).))))..)))))))...---- ( -38.39, z-score = -2.20, R) >droSim1.chr2L 18724378 80 - 22036055 --GCGGCAGCCACAUGAGCAGCCUCAU--AGCCGC-CCGGAAAACUG---GGACUCAACUUGCCGGCCAAAUGUGGCUGAACC--CGUUG---------------------- --((((((((((((((((....)))..--.(((((-((((....)))---))...........))))...)))))))))...)--)))..---------------------- ( -30.90, z-score = -2.01, R) >droSec1.super_4 3790204 103 - 6179234 UCGUCAUCGUCUCCAGUGAUGCCGCAA--AACUUCAUCACAUCGCCG---AGAUCCAGCUUUCCAUUGCUACUUCGCUGGAUCGUUGUUGGCGGUGGCGAUGGAGACU---- ((..(((((((.((.((((((..(...--..)..))))))...((((---(((((((((................))))))))....))))))).)))))))..))..---- ( -41.29, z-score = -3.35, R) >droEre2.scaffold_4820 7363946 103 + 10470090 UCGUCAUCGUCUCCCGUGAUUCCGCAA--AACUUCAUCACAUCGCCG---AGAUCGAGCUUUCCAUUGCUACUUCGCUGGAUCGUCGUUGGUGGUGGCGAGGGAGACU---- ........((((((((((((.......--......))))).((((((---..(((((((..((((..((......))))))..))..)))))..))))))))))))).---- ( -37.42, z-score = -1.78, R) >droYak2.chr3R 7259392 103 + 28832112 UCGUCAUCAUCUCCCGUGAUUCCGCAA--AACUUCAUCACAUCGCCG---AGAUCCAGCUUUCCAUUGCUACUUCGUUGGAUCGUCGUUGGCGGUGGCGAGGGAGACU---- .........(((((((((....)))..--.......((.((((((((---(((((((((................))))))))....)))))))))..))))))))..---- ( -36.79, z-score = -2.30, R) >droAna3.scaffold_13340 4047283 103 + 23697760 UCGUCAUCGUCGCCCGUGAUGCCGCAA--AACUUCAUCACGUCGCCG---AGAUCCAGCUUCCCGUUGCUGCUCCUCUGGAUCGUGGUGGGCGGUGGCGAUGGCGACU---- (((((((((((((((((((((..(...--..)..)))))))((((((---.(((((((......((....))....))))))).))))))..))))))))))))))..---- ( -49.10, z-score = -3.51, R) >dp4.chr2 21034145 103 + 30794189 UCGUCGUCGUCGCCCGUGAUGCCGCAG--AACUUCAUUACGUCCCCG---AGGUCCAGUUUCCCGUUGCUGCUCCUCUGGAUGUGCGUGGGCGGCGGCGAUGGCGACU---- ..((((((((((((......(((((..--....(((((((((((..(---(((..((((........))))..)))).))))))).))))))))))))))))))))).---- ( -50.70, z-score = -3.52, R) >droPer1.super_0 10719661 103 - 11822988 UCAUCGUCGUCGCCCGUGAUGCCGCAG--AACUUCAUUACGUCCCCG---AGGUCCAGUUUCCCGUUGCUGCUCCUCUGGAUGUGCGUGGGCGGCGGCGAUGGCGACU---- ((((((((((((((((((....)))..--........(((((((..(---(((..((((........))))..)))).)))))))...))))))))))))))).....---- ( -50.40, z-score = -3.69, R) >droWil1.scaffold_181108 4308895 103 + 4707319 UCAUCGUCGUCGCCGGUGAUUCCACAA--AAUUUCAUCACAUCGCCG---AGAUCAAGUUUGCCAUUGCUGGCACGUUGGAUAUGCGUUGGAGGUGGUGAGGGGGAUU---- ..(((.((.((((((((((........--....))))).((.(((..---..(((.((..(((((....)))))..)).)))..))).)).....))))).)).))).---- ( -31.10, z-score = 0.11, R) >droMoj3.scaffold_6540 7632880 103 - 34148556 UCGUCGUCAUCCUCCGUGAUGCCGCAG--AACUUCAUCACAUCGCCG---AGAUCGAGUUUGCCGUUGCUCGAGCGCUGGAUGUGCGUGGGCGGCGGCGACGGCGACU---- ((((((((...(((.((((((......--..........)))))).)---)).........(((((((((((.((((.....)))).)))))))))))))))))))..---- ( -48.49, z-score = -1.85, R) >droGri2.scaffold_14906 12188186 103 + 14172833 UCGUCGUCGUCCUCAGUUAUGCCGCAG--AACUUCAUCACAUCGCCG---AGAUCUAGUUUGCCAUUGCUGGCACGCUGUAUGUGCGUCGGCGGCGGCGAUGGCGAUU---- (((((((((....)......(((((.(--(......))....(((((---(.........(((((....)))))(((.......))))))))))))))))))))))..---- ( -39.90, z-score = -1.24, R) >droVir3.scaffold_12855 9267655 103 + 10161210 UCGUCGUCGUCCUCCGUAAUGCCGCAG--AACUUCAUCACAUCCCCG---AGAUCUAGUUUGCCAUUGCUGGCGCGUUGUAUGUGUGUGGGCGGCGGUGAUGGUGACU---- ((..((((((((.(....(((((((.(--((((..(((.(......)---.)))..)))))((((....)))))))..))))....).))))))))..)).(....).---- ( -34.90, z-score = -0.48, R) >anoGam1.chr2R 19522282 112 - 62725911 CGGCCGUGGCGACCGGCGGUUGCAUGAUUGGUUUCACUUUCUUCUUGUUUCGUCGCUCCUUUUUAGGUUUUUCUACCUCAAUCCGCGCGGCCGGCAAGGAUAGGGCCGCCGC (((((.((((((((...))))))..((..(......)..))((((((((..(((((.(...((.((((......)))).))...).))))).)))))))))).))))).... ( -36.90, z-score = 0.08, R) >consensus UCGUCGUCGUCGCCCGUGAUGCCGCAA__AACUUCAUCACAUCGCCG___AGAUCCAGCUUGCCAUUGCUGCUUCGCUGGAUCUGCGUGGGCGGCGGCGAUGGCGACU____ ((((((((.......(((((...............)))))......................(((.((......)).)))............))))))))............ ( -9.21 = -9.65 + 0.44)

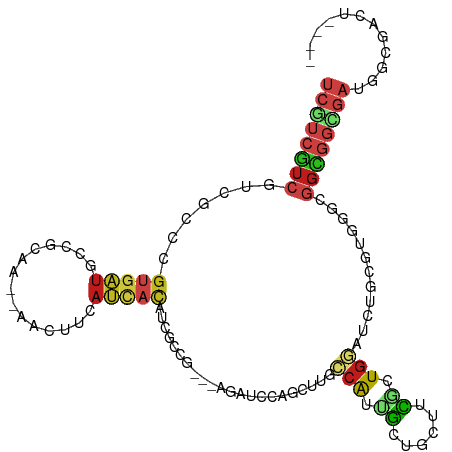
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:53:24 2011