
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 24,661,593 – 24,661,695 |
| Length | 102 |
| Max. P | 0.991990 |

| Location | 24,661,593 – 24,661,695 |
|---|---|
| Length | 102 |
| Sequences | 11 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 91.82 |
| Shannon entropy | 0.16437 |
| G+C content | 0.56863 |
| Mean single sequence MFE | -45.54 |
| Consensus MFE | -34.17 |
| Energy contribution | -34.26 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.75 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.850779 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
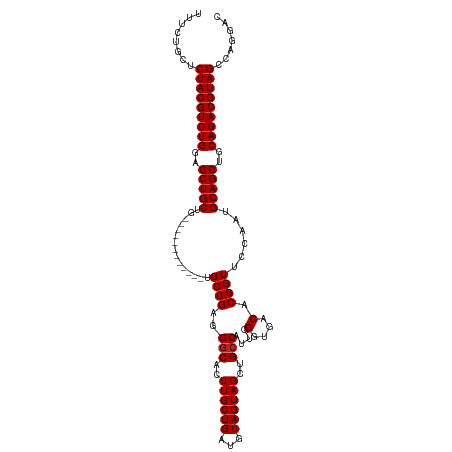
>dm3.chr3R 24661593 102 + 27905053 UUUCUGCUCUACGUCUGGAGCUGCUG------------UGCUGAGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCCGCAGACGUAGCCAGGAC .(((((..(((((((((((((((.((------------((((.((((((.((((((...)))))).)))).)).))).))))))))))).(((....)))))))))).))))). ( -43.10, z-score = -1.90, R) >droSim1.chr3R 24344749 102 + 27517382 UUUCUGCUCUACGUCUGGAGCUGCUG------------UGCUGAGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCCGCAGACGUAGCCAGGAC .(((((..(((((((((((((((.((------------((((.((((((.((((((...)))))).)))).)).))).))))))))))).(((....)))))))))).))))). ( -43.10, z-score = -1.90, R) >droSec1.super_4 3525084 102 + 6179234 UUUCUGCUCUACGUCUGGAGCUGCUG------------UGCUGAGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCCGCAGACGUAGCCAGGAC .(((((..(((((((((((((((.((------------((((.((((((.((((((...)))))).)))).)).))).))))))))))).(((....)))))))))).))))). ( -43.10, z-score = -1.90, R) >droYak2.chr3R 6988356 102 - 28832112 UUUCUGCUCUACGUCUGGAGCUGCUG------------UGCUGAGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCCGCAGACGUAGCCAGGAC .(((((..(((((((((((((((.((------------((((.((((((.((((((...)))))).)))).)).))).))))))))))).(((....)))))))))).))))). ( -43.10, z-score = -1.90, R) >droEre2.scaffold_4820 7093923 102 - 10470090 UUUCUGCUCUACGUCUGGAGCUGCUG------------UGCUGAGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCCGCAGACGUAGCCAGGAC .(((((..(((((((((((((((.((------------((((.((((((.((((((...)))))).)))).)).))).))))))))))).(((....)))))))))).))))). ( -43.10, z-score = -1.90, R) >droAna3.scaffold_13340 17459316 98 + 23697760 ----UGCUCUACGUCUGGAGCUGCUG------------UGCUGGGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCUGCAGACGUAGCCUGGAC ----....(((((((((.((((((..------------...((((((((.((((((...)))))).))))(((((....).))))))))..)))))).)))))))))....... ( -42.80, z-score = -1.87, R) >dp4.chr2 17038635 105 - 30794189 UUUCUGCUCUACGUCUGGAGCUGCUGCU---------GUGCUGGGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCUGCAGACGUAGCCAGGAC .(((((..(((((((((.((((((.(((---------((((..(.((((.((((((...)))))).)))).)..)....))))))......)))))).))))))))).))))). ( -49.10, z-score = -2.90, R) >droPer1.super_0 6692043 105 + 11822988 UUUCUGCUCUACGUCUGGAGCUGCUGCU---------GUGCUGGGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCUGCAGACGUAGCCAGGAC .(((((..(((((((((.((((((.(((---------((((..(.((((.((((((...)))))).)))).)..)....))))))......)))))).))))))))).))))). ( -49.10, z-score = -2.90, R) >droWil1.scaffold_181089 9365873 111 + 12369635 UUUCUGAUCUACGUCUGGAGCUGCUGCUACUGCU---GUGCUGCUGGCACUUGCUGAUGUAGUAGCCGCCAUUGGGUGACACGGUUCCAAUGCAGCUGCAGACGUAGCCUGAAC .(((....(((((((((.((((((((((((.((.---((((.....))))..))....)))))))).((.((((((.........)))))))))))).)))))))))...))). ( -49.70, z-score = -3.72, R) >droVir3.scaffold_13047 8993161 102 - 19223366 UUUCUGCUCUACGUCUGGAGCUGCUG------------UGCUGUUGGCACUUGCUGAUGUAGUAGCCGCCAUUGGGUGACACGGUUCCAAUGCAGCUGCAGACGUAGCUUGCGC .....((.(((((((((.((((((..------------.(((((((((..((((((...))))))..))))...(....))))))......)))))).)))))))))...)).. ( -43.80, z-score = -2.14, R) >droMoj3.scaffold_6540 5769096 114 - 34148556 UUUCUGCUCUACGUCUGGAGCUGCUGCUGCUGCUGCUGUGCUGUGGACACUUGCUGAUGUAGUAGCCGCCAUUGGGUGACACGGUUCCAAUGCAGCUGCAGACGUAGCCUGCCC .....((.(((((((((.((((((.((.((((((((...((.((....))..))....)))))))).)).((((((.........)))))))))))).)))))))))...)).. ( -50.90, z-score = -2.90, R) >consensus UUUCUGCUCUACGUCUGGAGCUGCUG____________UGCUGAGGGCACUUGCUGAUGUAGUAGCUGCCAUUGGGUGACACGGUUCCAAUGCAGCUGCAGACGUAGCCAGGAC ........(((((((((..(((((.....................(((..((((((...))))))..)))((((((.........)))))))))))..)))))))))....... (-34.17 = -34.26 + 0.09)


| Location | 24,661,593 – 24,661,695 |
|---|---|
| Length | 102 |
| Sequences | 11 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 91.82 |
| Shannon entropy | 0.16437 |
| G+C content | 0.56863 |
| Mean single sequence MFE | -41.17 |
| Consensus MFE | -36.55 |
| Energy contribution | -36.03 |
| Covariance contribution | -0.53 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.89 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.51 |
| SVM RNA-class probability | 0.991990 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 24661593 102 - 27905053 GUCCUGGCUACGUCUGCGGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCUCAGCA------------CAGCAGCUCCAGACGUAGAGCAGAAA ...(((.(((((((((.((((((..........(((((.....)))))(((........)))..((((.....)))------------).)))))).)))))))))..)))... ( -40.80, z-score = -2.79, R) >droSim1.chr3R 24344749 102 - 27517382 GUCCUGGCUACGUCUGCGGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCUCAGCA------------CAGCAGCUCCAGACGUAGAGCAGAAA ...(((.(((((((((.((((((..........(((((.....)))))(((........)))..((((.....)))------------).)))))).)))))))))..)))... ( -40.80, z-score = -2.79, R) >droSec1.super_4 3525084 102 - 6179234 GUCCUGGCUACGUCUGCGGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCUCAGCA------------CAGCAGCUCCAGACGUAGAGCAGAAA ...(((.(((((((((.((((((..........(((((.....)))))(((........)))..((((.....)))------------).)))))).)))))))))..)))... ( -40.80, z-score = -2.79, R) >droYak2.chr3R 6988356 102 + 28832112 GUCCUGGCUACGUCUGCGGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCUCAGCA------------CAGCAGCUCCAGACGUAGAGCAGAAA ...(((.(((((((((.((((((..........(((((.....)))))(((........)))..((((.....)))------------).)))))).)))))))))..)))... ( -40.80, z-score = -2.79, R) >droEre2.scaffold_4820 7093923 102 + 10470090 GUCCUGGCUACGUCUGCGGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCUCAGCA------------CAGCAGCUCCAGACGUAGAGCAGAAA ...(((.(((((((((.((((((..........(((((.....)))))(((........)))..((((.....)))------------).)))))).)))))))))..)))... ( -40.80, z-score = -2.79, R) >droAna3.scaffold_13340 17459316 98 - 23697760 GUCCAGGCUACGUCUGCAGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCCCAGCA------------CAGCAGCUCCAGACGUAGAGCA---- .......(((((((((.((((((..........(((((.....)))))(((........)))..((((.....)))------------).)))))).)))))))))....---- ( -37.30, z-score = -2.73, R) >dp4.chr2 17038635 105 + 30794189 GUCCUGGCUACGUCUGCAGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCCCAGCAC---------AGCAGCAGCUCCAGACGUAGAGCAGAAA ...(((.(((((((((.(((((((((((...........))))).((.(((........)))..((((.....))))---------.)).)))))).)))))))))..)))... ( -43.20, z-score = -3.65, R) >droPer1.super_0 6692043 105 - 11822988 GUCCUGGCUACGUCUGCAGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCCCAGCAC---------AGCAGCAGCUCCAGACGUAGAGCAGAAA ...(((.(((((((((.(((((((((((...........))))).((.(((........)))..((((.....))))---------.)).)))))).)))))))))..)))... ( -43.20, z-score = -3.65, R) >droWil1.scaffold_181089 9365873 111 - 12369635 GUUCAGGCUACGUCUGCAGCUGCAUUGGAACCGUGUCACCCAAUGGCGGCUACUACAUCAGCAAGUGCCAGCAGCAC---AGCAGUAGCAGCAGCUCCAGACGUAGAUCAGAAA .(((.(((((((((((.(((((((((((...........)))))....((((((......((..((((.....))))---.)))))))).)))))).))))))))).)).))). ( -43.80, z-score = -3.11, R) >droVir3.scaffold_13047 8993161 102 + 19223366 GCGCAAGCUACGUCUGCAGCUGCAUUGGAACCGUGUCACCCAAUGGCGGCUACUACAUCAGCAAGUGCCAACAGCA------------CAGCAGCUCCAGACGUAGAGCAGAAA ..((...(((((((((.((((((.........(((((.((....)).))).))...........((((.....)))------------).)))))).))))))))).))..... ( -39.80, z-score = -2.82, R) >droMoj3.scaffold_6540 5769096 114 + 34148556 GGGCAGGCUACGUCUGCAGCUGCAUUGGAACCGUGUCACCCAAUGGCGGCUACUACAUCAGCAAGUGUCCACAGCACAGCAGCAGCAGCAGCAGCUCCAGACGUAGAGCAGAAA ..((...(((((((((.((((((.........(((((.((....)).))).)).......((..((((.....)))).((....)).)).)))))).))))))))).))..... ( -41.60, z-score = -1.16, R) >consensus GUCCUGGCUACGUCUGCAGCUGCAUUGGAACCGUGUCACCCAAUGGCAGCUACUACAUCAGCAAGUGCCCUCAGCA____________CAGCAGCUCCAGACGUAGAGCAGAAA .......(((((((((.(((((((((((...........)))))(((((((........)))...)))).....................)))))).)))))))))........ (-36.55 = -36.03 + -0.53)


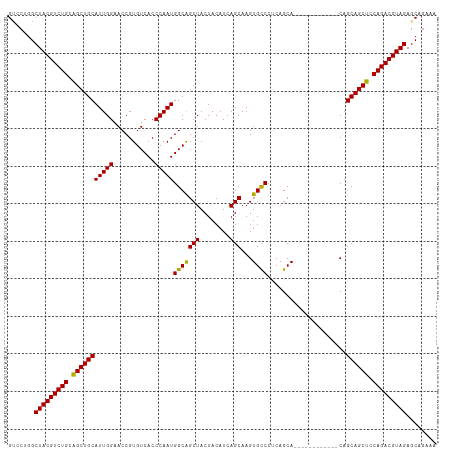
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:52:39 2011