| Sequence ID | dm3.chr3R |
|---|---|
| Location | 24,428,266 – 24,428,414 |
| Length | 148 |
| Max. P | 0.995174 |

| Location | 24,428,266 – 24,428,414 |
|---|---|
| Length | 148 |
| Sequences | 11 |
| Columns | 167 |
| Reading direction | forward |
| Mean pairwise identity | 76.07 |
| Shannon entropy | 0.49315 |
| G+C content | 0.42815 |
| Mean single sequence MFE | -45.10 |
| Consensus MFE | -28.14 |
| Energy contribution | -27.25 |
| Covariance contribution | -0.89 |
| Combinations/Pair | 1.46 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.62 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.77 |
| SVM RNA-class probability | 0.995174 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
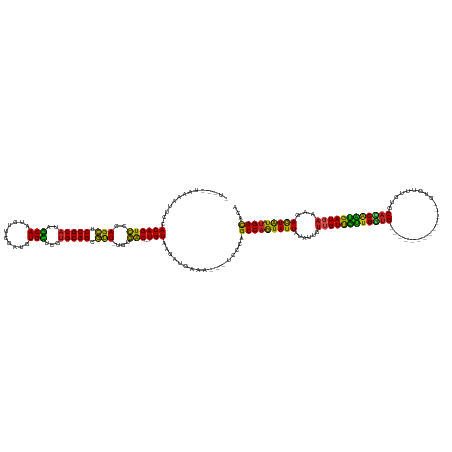
>dm3.chr3R 24428266 148 + 27905053 UUAGCUAAAAUUCCACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGCUCGUGCGGCGUU-UGUGAC--UGUAAGAUGAAA---UCCCAGCCAGUAUUAUAUUGUUGUUGUUGGUG-------------GUGUUUGUGCACCAAUGAUGAAAGAGUAUAUGGCACA .........(((..(((((((.(((.((((((((((..........)))).)))))).)))-.).)))--)))..)))....---.....(((((((((......(..(..((((((-------------.........))))))..)..)...))))).))))... ( -44.00, z-score = -1.57, R) >droSim1.chr3R 24119369 148 + 27517382 UUAGCUAAAAUUCCACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGCUCGUGCGGCGUU-UGUGAC--UGUAAGAUGAAA---UCCCAGCCAGUAUUAUAUUGUUGUUGUUGGUG-------------GUGUUUGUGCACCAAUGAUGAAAGAGUAUAUGGCACA .........(((..(((((((.(((.((((((((((..........)))).)))))).)))-.).)))--)))..)))....---.....(((((((((......(..(..((((((-------------.........))))))..)..)...))))).))))... ( -44.00, z-score = -1.57, R) >droSec1.super_22 1030648 148 + 1066717 UUAGAUAAAAUUCCACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGCUCGUGCGGCGUU-UGUGAC--UGUAAGAUGAAA---UCCCAGCCAGUAUUAUAUUGUUGUUGUUGGUG-------------GUGUUUGUGCACCAAUGAUGAAAGAGUAUAUGGCACA ...(((...(((..(((((((.(((.((((((((((..........)))).)))))).)))-.).)))--)))..)))...)---))...(((((((((......(..(..((((((-------------.........))))))..)..)...))))).))))... ( -45.70, z-score = -2.38, R) >droYak2.chr3R 6766277 148 - 28832112 GUUGCUAUGAUUCCACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGCUCAUGCGGCGUU-UGUGAC--UGUAAGAUGAAA---CCCCAGCCAGUAUUAUAUUGUUGUUGUUGGUG-------------GUGUAUGUGCACCAAUGAUGAAAGAGUAUAUGGCACA ..(((((((((((.(((((((.(((.((((((((((..........)))).)))))).)))-.).)))--))).........---..((((((((((......)))))..)))))((-------------((((....)))))).........)))).))))))).. ( -45.30, z-score = -1.26, R) >droEre2.scaffold_4820 6864513 148 - 10470090 UUAGCUACGAUUCCACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGCUCGUGCGGCGUU-UGUGAC--UGUAAGAUGAAA---UCCCAGCCAGUAUUAUAUUAUUGUUGUUGGUG-------------GUGUAUGUGCAUCAAUGAUGAAAGAGUAUAUGGCACA ...((((..((((.(((((((.(((.((((((((((..........)))).)))))).)))-.).)))--)))..((((..(---((((((((((((......)))))..))))).)-------------)).......))))..........))))...))))... ( -44.10, z-score = -1.27, R) >droAna3.scaffold_13340 17232742 142 + 23697760 ----AUAAAAUUC-ACAGUCCCAGCUCCGCAUAGCAAUGCUCGAUGUGCUCUUGCGGCGUU-UGUGAC--UGUGAGAUGAAA---AACCAGCCAGUAUUAUAUUGCUGUUGUUGGUG-------------CUCU-UGUGCACCAAUGAUGAAAAAGUAUAUGGCACA ----......(((-(((((((.(((.(((((.((((..........))))..))))).)))-.).)))--))))))......---.....((((....((((((..(((..((((((-------------(...-...)))))))..)))....))))))))))... ( -50.70, z-score = -3.91, R) >dp4.chr2 27182345 134 - 30794189 --------------ACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGUCUGUGCGGCGUUUCAUGAC--UGUAAAAUGAAA---UCCCAGCCAGUAUUAUAUCAUUGUUGUUGGUG--------------CUUGCGUGCAUCAGCGAUGAAAGAGUAUUUGGCACA --------------((((((..(((.((((((((((..........)).)))))))).)))....)))--))).........---.....(((((((((...(((((((((...(..--------------(....)..)..)))))))))...)))).)))))... ( -45.40, z-score = -2.63, R) >droPer1.super_7 736839 139 + 4445127 ---------AAUACACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGUCUGUGCGGCGUUUCAUGAC--UGUAAAAUGAAA---ACCCAGCCAGUAUUAUAUCAUUGUUGCUGGUG--------------CUUGCGUGCAUCAGCGAUGAAAGAGUAUUUGGCACA ---------.....((((((..(((.((((((((((..........)).)))))))).)))....)))--))).........---.....(((((((((......(..(((((((((--------------(......))))))))))..)...)))).)))))... ( -47.90, z-score = -2.79, R) >droWil1.scaffold_181089 9124455 151 + 12369635 -------------CACAGUCCCAGCUCCGCAUGGCACUAUUCAAUGUGCUCAUGCGGCGUUCUACGAC--UGUAAGAUGAACAU-UUUUGGCCAGUAUUAUAUUAUUGUUGUUGAUGUUCAGUUAUUUAUAUAACUAUGCAUCGAUGAUGAAAAAGUAUUUGGUACA -------------.((((((..(((.(((((((((((........)))).))))))).)))....)))--)))...........-.....(((((...(((.((.(..(..(((((((..((((((....))))))..)))))))..)..).)).))).)))))... ( -52.20, z-score = -4.93, R) >droVir3.scaffold_13047 6807744 147 - 19223366 -UUAGGCAAAAUACACAGUCCCAGCUCCGCAUUGCAAUGUUUGAUGUGUCUCUGCGGCGUUUUAUGGC--UGUAAGAUUAAA---ACCCAGCCCAUAUUAUAUUUUAGUUAUUGUUG--------------UAAUUGUGCAUCAAUGAUCAAAAAGUAUUUGGCACA -....((.((((((...(((.(((...((((((.........))))))...))).)))((..((((((--((..........---...))).)))))..)).((((.(((((((.((--------------((....)))).))))))).)))).)))))).))... ( -33.22, z-score = -0.18, R) >droGri2.scaffold_14830 715640 144 + 6267026 ----------UUGAACAGUUCCAGACCCGCUCUACA----CACGUGUGUUUUUGCGGUUCUUUACAACACUGUAAAAGCUGUUAAGCCCAGCCAAUAUUAUAUUUUAGUCAUCGGUGG--------CUAUUUUCGCUUGCCGCAAUGAUCAAUGAAUGU-UGGCCCA ----------...(((((((...((.((((...(((----(....))))....)))).))((((((....))))))))))))).......(((((((((.((((...(((((..((((--------(...........))))).))))).)))))))))-))))... ( -43.60, z-score = -3.32, R) >consensus _U___UAAAAUUCCACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGCUCGUGCGGCGUU_UGUGAC__UGUAAGAUGAAA___UCCCAGCCAGUAUUAUAUUGUUGUUGUUGGUG_____________GUGUUUGUGCACCAAUGAUGAAAGAGUAUAUGGCACA ..............((((((..(((.((((((.(((........)))....)))))).)))....)))..))).................(((((((((......((((((((((((......................))))))))))))...))))).))))... (-28.14 = -27.25 + -0.89)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:51:49 2011