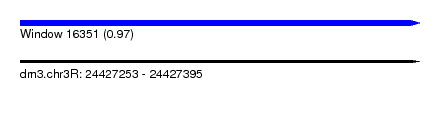
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 24,427,253 – 24,427,395 |
| Length | 142 |
| Max. P | 0.972225 |

| Location | 24,427,253 – 24,427,395 |
|---|---|
| Length | 142 |
| Sequences | 12 |
| Columns | 158 |
| Reading direction | forward |
| Mean pairwise identity | 75.94 |
| Shannon entropy | 0.49494 |
| G+C content | 0.43789 |
| Mean single sequence MFE | -42.82 |
| Consensus MFE | -22.85 |
| Energy contribution | -21.39 |
| Covariance contribution | -1.45 |
| Combinations/Pair | 1.47 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.53 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.972225 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 24427253 142 + 27905053 ACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGCUUGUGCGGCGUUU-GUGACUGUAAGAAUAAAUUC-CAGCCAGUAUUAUAUCAUUGUUGUU--GGUGCUCUUCGUGCAUCAACGAUGAAAGAGUAUAUGGCACACUAUAGCU------------ (((((((.(((.((((((((((..........))).))))))).))).-).))))))............-..(((((((((...(((((((((..--.(..(.....)..)..)))))))))...))))).))))...........------------ ( -47.50, z-score = -2.92, R) >droSim1.chr3R 24118376 142 + 27517382 ACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGCUCGUGCGGCGUUU-GUGACUGUAAGAUGAAAUCC-CAGCCAGUAUUAUAUCAUUGUUGUU--GGUGCUGUUCGUGCAUCAACGAUGAAAGAGUAUAUGGCACAAUAUAGCU------------ (((((((.(((.((((((((((..........)))).)))))).))).-).))))))............-..(((((((((...(((((((((..--.(..(.....)..)..)))))))))...))))).))))...........------------ ( -47.50, z-score = -2.68, R) >droSec1.super_22 1029649 142 + 1066717 ACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGCUCAUGCGGCGUUU-GUGACUGUAAGAUGAAAUCC-CAGCCAGUAUUAUAUCAUUGUUGUU--GGUGCUCUUCGUGCAUCAACGAUGAAAGAGUAUAUGGCACACUAUAGCU------------ (((((((.(((.((((((((((..........)))).)))))).))).-).))))))............-..(((((((((...(((((((((..--.(..(.....)..)..)))))))))...))))).))))...........------------ ( -47.50, z-score = -2.71, R) >droEre2.scaffold_4820 6863762 142 - 10470090 ACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGCUCUUGCGGCGUUU-GUGACUGUAAGAUAAAAUCC-CAGCCAGUAUUAUAUCAUUGUUGUU--GGUGCUCUUCGUGCAUCAACGAUGAAAGAGUAUUUGGCACACUAUAGCU------------ (((((((.(((.(((((.((((..........))))..))))).))).-).))))))............-..(((((((((...(((((((((..--.(..(.....)..)..)))))))))...)))).)))))...........------------ ( -46.10, z-score = -2.87, R) >droYak2.chr3R 6765543 141 - 28832112 ACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGCUCGUGCGGCGUUU-GUGACUGUAAGAUGAAAUCC-CAGCCAGUAUUAUAUCGU-GUUGUU--GGUGCUCUUCGUGCAUCAACGAUGAAAGAGUAUUUGGCACACUAUAGCU------------ (((((((.(((.((((((((((..........)))).)))))).))).-).))))))............-..(((((((((......(-((((((--(((((.......))))))))))))....)))).)))))...........------------ ( -46.90, z-score = -2.37, R) >droAna3.scaffold_13340 17232065 133 + 23697760 ACAGUCCCAGCUCCGCAUAGCAAUGCUCGAUGUGCUCUUGCGGCGUUU-GCGACUGUUAGAAUAACAUA---GCUCGUAUUAUAUCUUUGUCGGCUGGUCAUAUUCAAUGGGUAGUUGAUGAAAAUGUACGGGCCAC--------------------- (((((((.(((.(((((.((((..........))))..))))).))).-).))))))............---((((((((......((..(((((((.((((.....)))).)))))))..))...))))))))...--------------------- ( -45.90, z-score = -2.72, R) >dp4.chr2 27180778 142 - 30794189 ACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGUCUGUGCGGCGUUUCAUGACUGUAAAAUGAAAUCC-CAGCCAGUAUUAUAUCAUUGUUGCU--GGUGAU-UCCAUGCAUCAGCGAUGAAAGAGUAUUUGGCACACUAUACCC------------ ((((((..(((.((((((((((..........)).)))))))).)))....))))))............-..(((((((((...(((((((((((--((....-.))).)))...)))))))...)))).)))))...........------------ ( -43.40, z-score = -2.41, R) >droPer1.super_7 735219 154 + 4445127 ACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGUCUGUGCGGCGUUUCAUGACUGUAAAAUGAAAACC-CAGUCAGUAUUAUAUCAUUGUUGCU--GAUGUU-UUCGUGCAUCAGCGAUGAAAGAGUAUUUGGCACACUCCCUACGAAAUUACAUCU ((((((..(((.((((((((((..........)).)))))))).)))....))))))............-..(((((((((......(..(((((--(((((.-.....))))))))))..)...)))).)))))....................... ( -43.40, z-score = -2.03, R) >droWil1.scaffold_181089 9123742 137 + 12369635 ACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGUCUAUGCGGCGUUUUUUGGCUGUAAAAUGAAUGUAUUGACUCAUAUUAUAUCUAUGUUGACUAGUCCC----AAUGACUAAUUGAUGAAAACGUAUGCGCCACACUA----------------- ((((((..(((.((((((((((..........)).)))))))).)))....)))))).............((.(.(((((....((...((..(.(((((..----...))))).)..))))....))))).).)).....----------------- ( -29.80, z-score = -0.11, R) >droVir3.scaffold_13047 6806981 138 - 19223366 ACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGUCUGUGCGGCGUUUCAUGACUGUAAGAGAAAUGUU---GCACAUAUUAUAUCUUUGUUAAUUGGGCACCCUGUAUGCCUAAUUGAUGAAAACGUAUGUUCCACAAUU----------------- ((((((..(((.((((((((((..........)).)))))))).)))....))))))........(((.---(.((((((......((..(((((((((((.......)))))))))))..))...)))))).).)))...----------------- ( -42.00, z-score = -3.03, R) >droMoj3.scaffold_6540 29400872 138 + 34148556 ACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGUCUGUGCGGCGUUCAUUGACUGUAAUAGAAAUCUC---GCACGUAUUAUAUCUUUGUUGCUUAGGCACCCAGAAUGCCUAACUGAUGAAAACGUAUGUGCCACAAUU----------------- ((((((..(((.((((((((((..........)).)))))))).)))....))))))............---((((((((......((..((..(((((((.......)))))))..))..))...)))))))).......----------------- ( -40.90, z-score = -2.51, R) >droGri2.scaffold_14830 714582 139 + 6267026 ACAGUCCCAGCUCCGCAUAGCAAUGUUUGAUGUGUCUGUGCGGCGUUUCAUGGCUGUAACAUAAAUGUUAUUGCACAUAUUAUAUCGCUGUUGAUGAGACACCCUGCAUGCCUUAUUGAUGAAAACGUAUGCGCCACAA------------------- ..............((((((((..........)).))))))(((((((((((((.((((((....)))))).)).))).....((((....))))))))).....((((((...............)))))))))....------------------- ( -32.96, z-score = 0.99, R) >consensus ACAGUCCCAGCUCCGCAUAGCAAUGUUCGAUGUGCCUGUGCGGCGUUU_GUGACUGUAAGAUGAAAUCC_CAGCCAGUAUUAUAUCAUUGUUGUU__GGUGCUCUUCAUGCAUCAACGAUGAAAGAGUAUGUGGCACACUAUA_C_____________ ((((((..(((.((((((.(((........)))....)))))).)))....))))))..................(((((.......((((((((...(((.......)))...))))))))....)))))........................... (-22.85 = -21.39 + -1.45)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:51:45 2011