
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 23,501,805 – 23,501,918 |
| Length | 113 |
| Max. P | 0.523393 |

| Location | 23,501,805 – 23,501,918 |
|---|---|
| Length | 113 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.45 |
| Shannon entropy | 0.40325 |
| G+C content | 0.66124 |
| Mean single sequence MFE | -55.52 |
| Consensus MFE | -25.10 |
| Energy contribution | -25.40 |
| Covariance contribution | 0.30 |
| Combinations/Pair | 1.62 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.45 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.523393 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
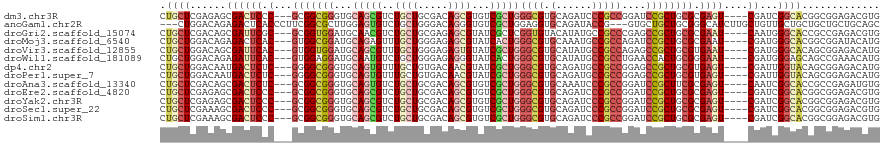
>dm3.chr3R 23501805 113 - 27905053 CUGCUCGAGAGCGACUCCC---GCGGCGGGUGCAGCGUCUGCUGCGACAGCGUGUCGCUGGGCGUGCAGAUCCCGCCGGAUCCGCUGCGCGAGU----CGAUCGGCACGGCGGAGACGUG .(((.(((...((((((.(---(((((((((.(.(((((((((((..(((((...))))).))).)))))...))).).)))))))))).))))----)).))))))..(((....))). ( -65.60, z-score = -2.42, R) >anoGam1.chr2R 56416775 114 + 62725911 ---CUGGACAGAGACUCACCCUUCGGCGCUUGGAGUGUCUGCUGGGACAGGGUGUCGCUGGAGGUGCAGAUACCG---GUGCUGCUGCGGGCAUCUUGCUGUUGCUGCUGCUGCUGCAGC ---(..(.((((.((((..((....).)....)))).)))))..)(((((((((((((.......)).))))))(---(((((......))))))...)))))...(((((....))))) ( -45.20, z-score = 0.87, R) >droGri2.scaffold_15074 5630934 113 + 7742996 CUGCUCGACAGCGAUUCGC---GCGGUGGAUGCAACGUCUGCUGCGAGAGCGUAUCGCUCGGUGUACAUAUGCCGCCCGAGCCGCUGCGCGAAU----CAAUGGGCACCGCCGAGACGUG ...(((..((..(((((((---(((((((((((...(....).((....)))))))((((((.((.........))))))))))))))))))))----)..))(((...))))))..... ( -50.00, z-score = -1.10, R) >droMoj3.scaffold_6540 1744485 113 + 34148556 CUGCUGGACAGAGACUCAC---GUGGCGGAUGCAGAGUUUGCUGGGAGAGCGUAUCACUGGGCGUGCAAAUGCCGCCAGAUCCGCUGCGCGAAU----CGAUGGGCACGGCGGAUACAUG ((((((..((..((.((.(---(..((((((.(((.(..((((.....))))...).)))((((.((....))))))..))))))..)).)).)----)..))....))))))....... ( -47.50, z-score = -2.08, R) >droVir3.scaffold_12855 8049995 113 + 10161210 CUGCUGGACAGCGAUUCAC---GUGGUGGAUGCAGCGUUUGCUGGGAGAGUGUAUCGCUGGGCGUGCAUAUGCCGCCAGAGCCGCUGCGUGAAU----CGAUGGGCACAGCGGAGACAUG ((((((..((.((((((((---(..((((...(((((..((((.....))))...)))))((((.((....))))))....))))..)))))))----)).))....))))))....... ( -55.80, z-score = -3.74, R) >droWil1.scaffold_181089 3742939 113 - 12369635 CUGCUGGACAGAGAUUCAC---GUGGAGGAUGCAAUGUCUGCUGGGAGAGGGUAUCACUGGGCGUGCAUAUGCCGCCUGAACCACUGCGGGAAU----CGAUGGGAGCAGCCGAAACAUG (((((...((..(((((.(---(..((((((((..(.(((....))).)..))))).))(((((.((....)))))))......)..)).))))----)..))..))))).......... ( -40.40, z-score = -1.28, R) >dp4.chr2 3856657 113 - 30794189 CUGCUGGACAAUGACUCUC---GGGGCGGGUGCAGUGUUUGCUGUGACAACGUAUCGCUGGGCGUGCAGAUGCCGCCGGAGCCGCUGCGUGAGU----CGAUUGGUACAGCGGAGACAUG ((((((..(((((((((.(---(.(((((.(.(((((..(((.((....))))).)))))((((.((....))))))..).))))).)).))))----).))))...))))))....... ( -53.10, z-score = -2.83, R) >droPer1.super_7 3950887 113 + 4445127 CUGCUGGACAAUGACUCUC---GGGGCGGGUGCAGUGUUUGCUGUGACAACGUAUCGCUGGGCGUGCAGAUGCCGCCGGAGCCGCUGCGUGAGU----CGAUUGGUACAGCGGAGACAUG ((((((..(((((((((.(---(.(((((.(.(((((..(((.((....))))).)))))((((.((....))))))..).))))).)).))))----).))))...))))))....... ( -53.10, z-score = -2.83, R) >droAna3.scaffold_13340 4511766 113 + 23697760 CUGCUCGACAGCGACUCUC---GCGGCGGGUGCAGUGUCUGCUGCGACAGCGUGUCGCUGGGCGUGCAAAUCCCGCCGGAUCCGCUUCGCGAGU----CAAUCGGCACCGCCGAGAUGUG ..(((((..((((....((---.(((((((((((.(((((...(((((.....))))).)))))))))...)))))))))..))))...)))))----...(((((...)))))...... ( -50.80, z-score = -0.58, R) >droEre2.scaffold_4820 5934561 113 + 10470090 CUGCUCGAGAGCGACUCCC---GCGGCGGGUGCAGCGUCUGCUGCGACAGCGUGUCGCUGGGCGUGCAGAUCCCGCCGGAUCCGCUGCGCGAGU----CGAUCGGCACGGCGGAGACGUG .(((.(((...((((((.(---(((((((((.(.(((((((((((..(((((...))))).))).)))))...))).).)))))))))).))))----)).))))))..(((....))). ( -65.60, z-score = -2.42, R) >droYak2.chr3R 5846877 113 + 28832112 CUGCUCGAGAGCGACUCCC---GCGGCGGGUGCAGCGUCUGCUGCGACAGCGUGUCGCUGGGCGUGCAGAUCCCGCCGGAUCCGCUGCGCGAGU----CGAUCGGCACGGCGGAGACGUG .(((.(((...((((((.(---(((((((((.(.(((((((((((..(((((...))))).))).)))))...))).).)))))))))).))))----)).))))))..(((....))). ( -65.60, z-score = -2.42, R) >droSec1.super_22 145027 113 - 1066717 CUGCUCGAAAGCGACUCCC---GCGGCGGGUGCAGCGUCUGCUGCGACAGCGUGUCGCUGGGCGUGCAGAUCCCGCCGGAUCCGCUGCGCGAGU----CGAUCGGCACGGCGGAGACGUG .(((.(((...((((((.(---(((((((((.(.(((((((((((..(((((...))))).))).)))))...))).).)))))))))).))))----)).))))))..(((....))). ( -64.50, z-score = -2.60, R) >droSim1.chr3R 23228960 113 - 27517382 CUGCUCGAAAGCGACUCCC---GCGGCGGGUGCAGCGUCUGCUGCGACAGCGUGUCGCUGGGCGUGCAGAUCCCGCCGGAUCCGCUGCGCGAGU----CGAUCGGCACGGCGGAGACGUG .(((.(((...((((((.(---(((((((((.(.(((((((((((..(((((...))))).))).)))))...))).).)))))))))).))))----)).))))))..(((....))). ( -64.50, z-score = -2.60, R) >consensus CUGCUCGACAGCGACUCCC___GCGGCGGGUGCAGCGUCUGCUGCGACAGCGUGUCGCUGGGCGUGCAGAUCCCGCCGGAUCCGCUGCGCGAGU____CGAUCGGCACGGCGGAGACGUG ..........(((.((((....(((((((...(((((..((((.....))))...)))))((((.(......)))))....)))))))((..((......))..)).....)))).))). (-25.10 = -25.40 + 0.30)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:48:55 2011