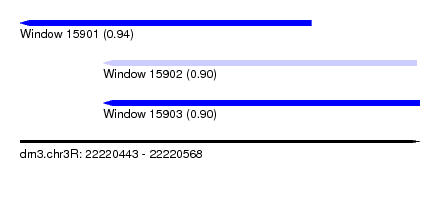
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 22,220,443 – 22,220,568 |
| Length | 125 |
| Max. P | 0.938649 |

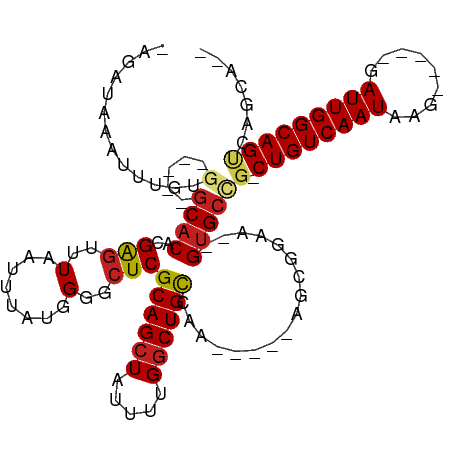
| Location | 22,220,443 – 22,220,534 |
|---|---|
| Length | 91 |
| Sequences | 10 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 80.16 |
| Shannon entropy | 0.38287 |
| G+C content | 0.57465 |
| Mean single sequence MFE | -38.29 |
| Consensus MFE | -27.39 |
| Energy contribution | -27.23 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.72 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.42 |
| SVM RNA-class probability | 0.938649 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
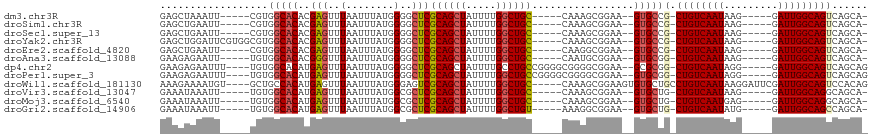
>dm3.chr3R 22220443 91 - 27905053 GGGCUCGCAGCUAUUUUGGCUGCCAAAGCGGAAGUG---CCGCUGUCAAUAAG-----GAUUGGCAGU---CAGCAUGCGGGUU-GGUUCAGACUUGGGGGCC-------- .(((((((((((.....((((((((((((((.....---))))).((......-----))))))))))---)))).))))))))-(((((........)))))-------- ( -38.00, z-score = -2.06, R) >droSim1.chr3R 22058132 91 - 27517382 GGGCUCGCAGCUAUUUUGGCUGCCAAAGCGGAAGUG---CCGCUGUCAAUAAG-----GAUUGGCAGU---CAGCAUGCGGGUU-GGUUCAGACUUGGGGGCA-------- .(((((((((((.....((((((((((((((.....---))))).((......-----))))))))))---)))).))))))))-.((((........)))).-------- ( -36.40, z-score = -1.70, R) >droSec1.super_13 999057 91 - 2104621 GGGCUCGCAGCUAUUUUGGCUGCCAAAGCGGAAGUG---CCGCUGUCAAUAAG-----GAUUGGCAGU---CAGCAUGCGGGUU-GGUUCAGACUUGGGGGCA-------- .(((((((((((.....((((((((((((((.....---))))).((......-----))))))))))---)))).))))))))-.((((........)))).-------- ( -36.40, z-score = -1.70, R) >droYak2.chr3R 4562872 91 + 28832112 GGGCUCGCAGCUAUUUUGGCUGCCAAAGCGGAAGUG---CCGCUGUCAAUAAG-----GAUUGGCAGU---CAGCAUGCGGGUU-GGUUCAGACUUGGGGUCC-------- ((((((((((((.....((((((((((((((.....---))))).((......-----))))))))))---)))).)))(((((-......))))).))))))-------- ( -36.80, z-score = -2.02, R) >droEre2.scaffold_4820 4638728 91 + 10470090 GGGCUCGCAGCUAUUUUGGCUGCCAAGGCGGAAGUG---CCGCUGUCAAUAAG-----GAUUGGCAGU---CAGCAUGCGGGUU-GGCUCAGACUUGUGGGCC-------- .(((((((((((.....((((((((((((((.....---))))).((......-----))))))))))---)))).))))))))-((((((......))))))-------- ( -41.60, z-score = -2.55, R) >droAna3.scaffold_13088 93773 84 - 569066 GGGCUCGCAGCUAUUUUGGCUGCCAAUGCGGAAGUG---CGGCUGUCAAUAAG-----GAUUGGCAGU---CAGCAUGCGGGUU-AGUUCAGGCUU--------------- (((((.((((((.....)))))).(((.((...(((---(((((((((((...-----.)))))))))---).)))).)).)))-)))))......--------------- ( -33.30, z-score = -2.02, R) >droWil1.scaffold_181130 7069225 103 - 16660200 GGAGUCGCAGCUAUUUUGGCUGCCAAAGCGGAAGUGUGCUGCCUGUCAAUAAGGAUUCGAUUGGCAGUCCACAGAAUGCGGCCU-GGCUGGAUCAACCUUGCCC------- ((.(((((((((.....))))))....(((...(((..(((((.((((((....))).))).)))))..)))....))))))))-(((.((.....))..))).------- ( -36.90, z-score = -0.79, R) >droVir3.scaffold_13047 4749764 100 - 19223366 GCGCUCGCAGCUAUUUUGGCUGCCAAAGCGGAAGUG---CUGCUGUCAAUAAG-----GAUUGGCAGG---CAGCAUGCGGGCUGAGCUGGUCUGGGCUGAGUUCACGGCU (((((((((...(((((.(((.....))).)))))(---(((((((((((...-----.))))))).)---)))).)))))))(((((((((....))).))))))..)). ( -44.90, z-score = -1.51, R) >droMoj3.scaffold_6540 12494759 93 + 34148556 GCGCUCGCAGCUAUUUUGGCUGCCAAAGCGGAAGUG---CUGCUGUCAAUGAG-----GAUUGGCAGG---CAGCAUGCGGGCUGAGCAGGACUGGGCUCGGAU------- .((((.((((((.....))))))...))))...(((---(((((((((((...-----.))))))).)---)))))).((((((.((.....)).))))))...------- ( -43.60, z-score = -2.38, R) >droGri2.scaffold_14906 10187856 87 - 14172833 GCGCUCGCAGCUAUUUUGGCUGUAAAGGCGGAAGUG---CUGCUGUCAAUAUG-----GAUUGGCAGC---CAGCAUGCGGGUUCGGCUCACACAGCU------------- (.(((((((...(((((.(((.....))).)))))(---(((((((((((...-----.)))))))).---)))).))))))).)((((.....))))------------- ( -35.00, z-score = -1.30, R) >consensus GGGCUCGCAGCUAUUUUGGCUGCCAAAGCGGAAGUG___CCGCUGUCAAUAAG_____GAUUGGCAGU___CAGCAUGCGGGUU_GGCUCAGACUUGGGGGCC________ ..(((((((((((((((.(((.....))).))))).......((((((((.........)))))))).....))).)))))))............................ (-27.39 = -27.23 + -0.16)
| Location | 22,220,469 – 22,220,567 |
|---|---|
| Length | 98 |
| Sequences | 12 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 86.25 |
| Shannon entropy | 0.26583 |
| G+C content | 0.49205 |
| Mean single sequence MFE | -38.08 |
| Consensus MFE | -23.30 |
| Energy contribution | -23.48 |
| Covariance contribution | 0.18 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.61 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.898997 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 22220469 98 - 27905053 -AGCUAAAUUCGUG-----GCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCAA-----AGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA-- -.(((........(-----((((..((((((........))))))((((((.....))))))...-----.......--)))))(-((((((((...-----.))))))))).))).-- ( -37.10, z-score = -2.55, R) >droSim1.chr3R 22058158 98 - 27517382 -AGCUGAAUUCGUG-----GCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCAA-----AGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA-- -.(((((....(((-----((((..((((((........))))))((((((.....))))))...-----.......--))))))-)(((((((...-----.))))))).))))).-- ( -41.20, z-score = -3.55, R) >droSec1.super_13 999083 98 - 2104621 -AGCUGAAUUCGUG-----GCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCAA-----AGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA-- -.(((((....(((-----((((..((((((........))))))((((((.....))))))...-----.......--))))))-)(((((((...-----.))))))).))))).-- ( -41.20, z-score = -3.55, R) >droYak2.chr3R 4562898 103 + 28832112 -AGCUGGAUUCGUGGCGUGGCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCAA-----AGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA-- -.((((((((((((((...)).))))))))..........(((..((((((.....))))))...-----.((....--)))))(-((((((((...-----.))))))))))))).-- ( -41.90, z-score = -2.64, R) >droEre2.scaffold_4820 4638754 98 + 10470090 -AGCUGAAUUCGUG-----GCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCAA-----GGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA-- -.(((((....(((-----((((..((((((........))))))((((((.....))))))...-----.......--))))))-)(((((((...-----.))))))).))))).-- ( -41.20, z-score = -3.39, R) >droAna3.scaffold_13088 93792 98 - 569066 AAGAGAA-UUUGUG-----GCACACGGGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCAA-----UGCGGAA--GUGCGG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA-- .......-..(((.-----((((..((((((........))))))((((((.....))))))...-----.......--))))((-((((((((...-----.)))))))))).)))-- ( -33.40, z-score = -1.89, R) >dp4.chr2 12011459 106 + 30794189 AAGAGAAUUUUGUG-----GCACAUGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGCCUGCCGGGGCGGGGCGGAA--GCGCGG-CUGUCAAUAGG-----GAUUGGCAGUCAGCAGC ...(((((.(((((-----((.(((((((...)))))))..)).)))))..))))).(((((....)))))((....--))((((-((((((((...-----.)))))))))).))... ( -41.10, z-score = -2.65, R) >droPer1.super_3 5860794 106 + 7375914 AAGAGAAUUUUGUG-----GCACAUGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCGGGGCGGGGCGGAA--GUGCGG-CUGUCAAUAGG-----GAUUGGCAGUCAGCAGC ...(((((.(((((-----((.(((((((...)))))))..)).)))))..))))).(((((.......(.((....--)).)((-((((((((...-----.)))))))))).))))) ( -37.60, z-score = -2.29, R) >droWil1.scaffold_181130 7069253 109 - 16660200 AAGAAAAUGUGCUG-----CCACAUGAGUUUAAUUUAUGGGAGUCGCAGCUAUUUUGGCUGCCAA-----AGCGGAAGUGUGCUGCCUGUCAAUAAGGAUUCGAUUGGCAGUCCACAGA .......((..(((-----((((((((((...))))))).((((((((((((((((.(((.....-----))).)))))).)))))((.......)))))))...))))))..)).... ( -38.70, z-score = -2.62, R) >droVir3.scaffold_13047 4749799 98 - 19223366 -AAAUAAAUUUGUG-----GCACAUGAGUUUAAUUUAUGGCGCUCGCAGCUAUUUUGGCUGCCAA-----AGCGGAA--GUGCUG-CUGUCAAUAAG-----GAUUGGCAGGCAGCA-- -...((((((..((-----...))..))))))........((((.((((((.....))))))...-----))))...--.(((((-((((((((...-----.))))))).))))))-- ( -34.90, z-score = -2.03, R) >droMoj3.scaffold_6540 12494787 98 + 34148556 -AAAUAAAUUUGUG-----GCACAUGAGUUUAAUUUAUGGCGCUCGCAGCUAUUUUGGCUGCCAA-----AGCGGAA--GUGCUG-CUGUCAAUGAG-----GAUUGGCAGGCAGCA-- -...((((((..((-----...))..))))))........((((.((((((.....))))))...-----))))...--.(((((-((((((((...-----.))))))).))))))-- ( -35.00, z-score = -1.85, R) >droGri2.scaffold_14906 10187878 98 - 14172833 -AAAUAAAUUUGUG-----GCACAUGAGUUUAAUUUAUGGCGCUCGCAGCUAUUUUGGCUGUAAA-----GGCGGAA--GUGCUG-CUGUCAAUAUG-----GAUUGGCAGCCAGCA-- -...((((((..((-----...))..)))))).....((((....(((((.(((((.(((.....-----))).)))--))))))-)(((((((...-----.)))))))))))...-- ( -33.60, z-score = -1.90, R) >consensus _AGAUAAAUUUGUG_____GCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCAA_____AGCGGAA__GUGCCG_CUGUCAAUAAG_____GAUUGGCAGUCAGCA__ ...................((((..(((..(........)..)))((((((.....)))))).................))))...((((((((.........))))))))........ (-23.30 = -23.48 + 0.18)

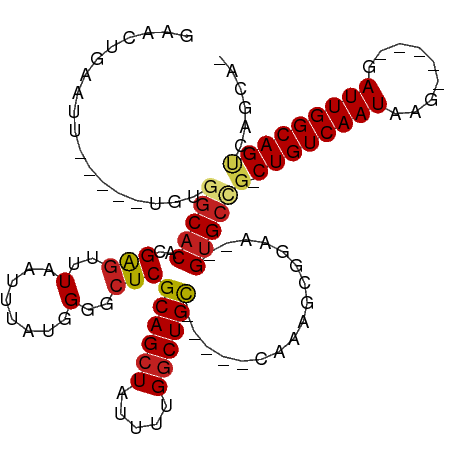
| Location | 22,220,469 – 22,220,568 |
|---|---|
| Length | 99 |
| Sequences | 12 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 86.90 |
| Shannon entropy | 0.25601 |
| G+C content | 0.49594 |
| Mean single sequence MFE | -38.05 |
| Consensus MFE | -24.13 |
| Energy contribution | -24.43 |
| Covariance contribution | 0.29 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.63 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.904392 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 22220469 99 - 27905053 GAGCUAAAUU-----CGUGGCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGC-----CAAAGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA- ..(((.....-----...(((((..((((((........))))))((((((.....))))))-----..........--)))))(-((((((((...-----.))))))))).))).- ( -37.10, z-score = -2.52, R) >droSim1.chr3R 22058158 99 - 27517382 GAGCUGAAUU-----CGUGGCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGC-----CAAAGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA- ..(((((...-----.(((((((..((((((........))))))((((((.....))))))-----..........--))))))-)(((((((...-----.))))))).))))).- ( -41.20, z-score = -3.52, R) >droSec1.super_13 999083 99 - 2104621 GAGCUGAAUU-----CGUGGCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGC-----CAAAGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA- ..(((((...-----.(((((((..((((((........))))))((((((.....))))))-----..........--))))))-)(((((((...-----.))))))).))))).- ( -41.20, z-score = -3.52, R) >droYak2.chr3R 4562898 104 + 28832112 GAGCUGGAUUCGUGGCGUGGCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGC-----CAAAGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA- ..((((((((((((((...)).))))))))..........(((..((((((.....))))))-----....((....--)))))(-((((((((...-----.))))))))))))).- ( -41.90, z-score = -2.57, R) >droEre2.scaffold_4820 4638754 99 + 10470090 GAGCUGAAUU-----CGUGGCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGC-----CAAGGCGGAA--GUGCCG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA- ..(((((...-----.(((((((..((((((........))))))((((((.....))))))-----..........--))))))-)(((((((...-----.))))))).))))).- ( -41.20, z-score = -3.37, R) >droAna3.scaffold_13088 93792 99 - 569066 GAAGAGAAUU-----UGUGGCACACGGGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGC-----CAAUGCGGAA--GUGCGG-CUGUCAAUAAG-----GAUUGGCAGUCAGCA- ..........-----(((.((((..((((((........))))))((((((.....))))))-----..........--))))((-((((((((...-----.)))))))))).)))- ( -33.40, z-score = -1.78, R) >dp4.chr2 12011460 106 + 30794189 GAAGAGAAUUU----UGUGGCACAUGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGCCUGCCGGGGCGGGGCGGAA--GCGCGG-CUGUCAAUAGG-----GAUUGGCAGUCAGCAG ....(((((.(----((((((.(((((((...)))))))..)).)))))..))))).(((((....)))))((....--))((((-((((((((...-----.)))))))))).)).. ( -41.10, z-score = -2.78, R) >droPer1.super_3 5860795 106 + 7375914 GAAGAGAAUUU----UGUGGCACAUGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGCCGGGGCGGGGCGGAA--GUGCGG-CUGUCAAUAGG-----GAUUGGCAGUCAGCAG ...........----....((.(((((((...))))))).(.(((((((((.....))))).)))).).(.((....--)).)((-((((((((...-----.)))))))))).)).. ( -36.80, z-score = -2.15, R) >droWil1.scaffold_181130 7069254 109 - 16660200 AAAGAAAAUGU----GCUGCCACAUGAGUUUAAUUUAUGGGAGUCGCAGCUAUUUUGGCUGC-----CAAAGCGGAAGUGUGCUGCCUGUCAAUAAGGAUUCGAUUGGCAGUCCACAG ........((.----.(((((((((((((...))))))).((((((((((((((((.(((..-----...))).)))))).)))))((.......)))))))...))))))..))... ( -38.70, z-score = -2.81, R) >droVir3.scaffold_13047 4749799 99 - 19223366 GAAAUAAAUU-----UGUGGCACAUGAGUUUAAUUUAUGGCGCUCGCAGCUAUUUUGGCUGC-----CAAAGCGGAA--GUGCUG-CUGUCAAUAAG-----GAUUGGCAGGCAGCA- ((((((...(-----((((((.(((((((...)))))))..)).))))).)))))).(((((-----(..(((((..--...)))-))((((((...-----.)))))).)))))).- ( -35.10, z-score = -2.08, R) >droMoj3.scaffold_6540 12494787 99 + 34148556 GAAAUAAAUU-----UGUGGCACAUGAGUUUAAUUUAUGGCGCUCGCAGCUAUUUUGGCUGC-----CAAAGCGGAA--GUGCUG-CUGUCAAUGAG-----GAUUGGCAGGCAGCA- ((((((...(-----((((((.(((((((...)))))))..)).))))).)))))).(((((-----(..(((((..--...)))-))((((((...-----.)))))).)))))).- ( -35.20, z-score = -1.83, R) >droGri2.scaffold_14906 10187878 99 - 14172833 GAAAUAAAUU-----UGUGGCACAUGAGUUUAAUUUAUGGCGCUCGCAGCUAUUUUGGCUGU-----AAAGGCGGAA--GUGCUG-CUGUCAAUAUG-----GAUUGGCAGCCAGCA- (((.((((((-----..((...))..)))))).))).((((....(((((.(((((.(((..-----...))).)))--))))))-)(((((((...-----.)))))))))))...- ( -33.70, z-score = -1.94, R) >consensus GAACUGAAUU_____UGUGGCACACGAGUUUAAUUUAUGGGGCUCGCAGCUAUUUUGGCUGC_____CAAAGCGGAA__GUGCCG_CUGUCAAUAAG_____GAUUGGCAGUCAGCA_ ..................(((((..(((..(........)..)))((((((.....)))))).................)))))..((((((((.........))))))))....... (-24.13 = -24.43 + 0.29)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:45:30 2011