
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 20,463,670 – 20,463,802 |
| Length | 132 |
| Max. P | 0.772725 |

| Location | 20,463,670 – 20,463,802 |
|---|---|
| Length | 132 |
| Sequences | 13 |
| Columns | 140 |
| Reading direction | forward |
| Mean pairwise identity | 76.53 |
| Shannon entropy | 0.51862 |
| G+C content | 0.61521 |
| Mean single sequence MFE | -57.08 |
| Consensus MFE | -28.30 |
| Energy contribution | -27.95 |
| Covariance contribution | -0.35 |
| Combinations/Pair | 1.62 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.50 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.772725 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 20463670 132 + 27905053 CAUCUGUACCAUGGGCCGCGGUGGACGGGG--UGAGUUCUUGGCUCCCAAUGGCCAAGGCGUAUGCU------UCGCACCGCGACGCGGAAAGUACGGUCCCAGAGGACCGAUCGCAUUGGGAGCAUUCGCCACGGACAG ...((((.(....).(((.(((((((((((--.....)))))((((((((((((....))((((..(------((((........)))))..))))(((((....))))).....)))))))))).)))))).))))))) ( -57.20, z-score = -1.73, R) >droSim1.chr3R 20302501 132 + 27517382 CAUCUGUACCAUGGGCCGCGGUGGACGUGG--UGAGUUCUUGGCUCCCAAUGGCCAAGGCGUAUGCU------UCGCACCGCGACGCGGGAAGUACGGUCCGAGAGGACCGAUCGCAUUGGGAGCAUUUGCCACGGACAG ...((((.((((.(....).)))).(((((--..(((.....((((((((((((....))...((((------((...((((...))))))))))((((((....))))))....)))))))))))))..))))).)))) ( -63.70, z-score = -3.47, R) >droSec1.super_0 20798033 132 + 21120651 CAUCUGUACCAUGGGCCGCGGUGGACGGGG--UGAGUUCUUGGCUCCCAAUGGCCAAGGCGUAUGCU------UCGCACCGCGACGCGGGAAAUACGGUCCCAGAGGACCGAUCGCAUUGGGAGCAUUUGCCACAGACAG ..(((((.((((.(....).))))....((--..(((.....((((((((((.((...((((.(((.------.......))))))).)).....((((((....))))))....)))))))))))))..)))))))... ( -53.90, z-score = -1.34, R) >droYak2.chr3R 2798013 132 - 28832112 CAUCUGUACCAUGGGUCGCGGUGGACGGGG--UGAGUUCUUGGCGCCCAAUGGCCAAGGCGUAUGCU------UUGCUCCGCGACGCGGGAAGUACGGUCCCAGAGGACCGAUCGCAUUGGGAGCAUUCGCCACGGACAG ..(((((.(((((((.(((.(.((((....--...)))).).)))))).))))....((((.(((((------(((((((((...))))).....((((((....))))))...)))....)))))).)))))))))... ( -56.80, z-score = -1.34, R) >droEre2.scaffold_4820 2875042 132 - 10470090 CAUCUGUACCAUGGGUCGCGGUGGACGGGG--UGAGUUCUUGGCGCCUAAUGGCCAAGGCGUGUGCU------UCGCUCCGCGACGCGGGAAAUACGGUCCCAGAGGACCGAUCGCAUUGGGAGCAUUCGCCACGGACAG ...((((.((.((((((((((.((.(((((--((.((.((((((........))))))))...))))------))))))))))))((((......((((((....)))))).))))..............))).)))))) ( -56.30, z-score = -1.21, R) >droAna3.scaffold_13340 18986354 132 - 23697760 CAUCUGGACCAUCGGACGCGGUGGUCUUGG--UGAAUUCUUAGCCCCCAAUGGCCAUGGCGAGUGCU------UUGCACCGCGACGUGGAAAAUAGGGUCCCAAGGGACCAAUGGCAUUGGGAGCGUUCGCGACAGAAAG ..((((((((((((....))))))))...(--(((((.....((.(((((((.((((.(((.((((.------..)))))))..............(((((....))))).))))))))))).))))))))..))))... ( -59.60, z-score = -3.67, R) >droPer1.super_7 2517224 132 - 4445127 CAUCUGGACCAUGGGCCGGGGCGGCCGUGG--CGAGUUCUUGGCCCCCAGCGGCCAGGGUGUGUGCU------UGGCUCCGCGACGUGGGAAGAACGGACCCAGGGGUCCGAUGGCAUUCGGCGCAUUGGCCACGGACAA ..(((((.(((((.((((((...((((((.--(((((((((((((......)))))))).....)))------)).)(((((...))))).....((((((....))))))))))).)))))).)).)))))).)).... ( -60.20, z-score = 0.48, R) >droWil1.scaffold_181089 2731571 138 - 12369635 CAUCUGCACCAUUGGUCUUGGUGGCCGAGG--UGAAUUCUUGGAACCCAAUGGCCAAGGUGUGUGCUGGUGCUUGGCUCCACGACGUGGAAAGUAUGGACCCAAUGGUCCAAUGGCAUUCGGGGCAUUGGCCACUGACAA .....(((((.((((((((((...((((((--.....))))))...)))).)))))))))))((((..((((((.(.(((((...)))))..((.((((((....))))))...))...).))))))..).)))...... ( -57.50, z-score = -1.84, R) >droVir3.scaffold_13047 756684 132 + 19223366 CAUUUGCACCAUUGGCCUUGGGGGUCGAGG--UGAAUUUUUGGCACCUAACGGCCAGGACGUCUGCU------UUGCACCGCGACGCGGAAAAUAAGGUCCUAACGGGCCUAUUGCAUUUGGUGCAUUGGCCACGGACAA ((..((((((((((((((((((.(((((((--.....))))))).))))).))))))....(((((.------((((...)))).))))).....((((((....))))))........))))))).))........... ( -53.10, z-score = -2.62, R) >droMoj3.scaffold_6540 4753393 132 + 34148556 CAUUUGCACCAUUGGCCUCGGGGGACGAGG--CGAGUUCUUAGCACCUAAAGGCCAGGGCGUCUGCU------UGGCACCGCGACGUGGAAAAUAUGGUCCUAAUGGGCCAAUUGCAUUGGGCGCAUUGGCCACUGAUAA ....(((...(((.((((((.....)))))--).))).....)))......((((((.(((((((..------..(((((((...))))......((((((....))))))..)))..))))))).))))))........ ( -51.50, z-score = -1.43, R) >dp4.chr2 29020063 132 - 30794189 CAUCUGGACCAUGGGCCGGGGCGGCCGUGG--CGAGUUCUUGGCCCCCAGCGGCCAGGGCGUGUGCU------UGGCUCCACGACGUGGGAAAAACGGACCCAGGGGUCCGAUGGCGUUCGGUGCAUUGGCCACGGACAA ((((.(((((.(((((((....(((((.((--((.((.(((((((......)))))))))...))))------)))))((((...))))......))).))))..)))))))))..(((((..((....))..))))).. ( -61.20, z-score = 0.08, R) >droGri2.scaffold_14906 8617237 132 - 14172833 CAUCUGCACCAUCGGCCUGGGUGGUCGUGG--CGAGUUCUUGGCACCCAGUGGCCACGGCGUUUGUU------UGGCACCGCGACGUGGAAAAUAGGGUCCCAAGGGGCCAAUUGCGUUGGGUGCAUUGGCCACAGACAA ..(((((.(((.(((((.....))))))))--.)...((((((.((((.((..(((((.(((.((..------...))..))).)))))...)).)))).))))))(((((((.(((.....)))))))))).))))... ( -55.20, z-score = -0.60, R) >triCas2.ChLG7 8785613 123 - 17478683 CAUCUGCACCAUCGGUCUUAGGGGCCGUGCGCCCCCUUUCCCGGCCACAACGGGCAGAGAAUGCCCC--------------CGUCGAGGGAAAUAGGGGCCCAGGGGCACCGCAUCGUGGCUGUCCUGGAAGGCAGA--- ..(((((..((.(((((.....))))))).(((((.((((((..(.((...(((((.....))))).--------------.)).).))))))..)))))((((((.((((((...)))).))))))))...)))))--- ( -55.90, z-score = -0.54, R) >consensus CAUCUGCACCAUGGGCCGCGGUGGACGUGG__UGAGUUCUUGGCACCCAAUGGCCAAGGCGUGUGCU______UGGCACCGCGACGCGGGAAAUACGGUCCCAGAGGACCGAUCGCAUUGGGAGCAUUGGCCACGGACAA ..((((.......(((((..(.((.....................)))..)))))..(((..((((............((((...))))......((((((....))))))............))))..))).))))... (-28.30 = -27.95 + -0.35)

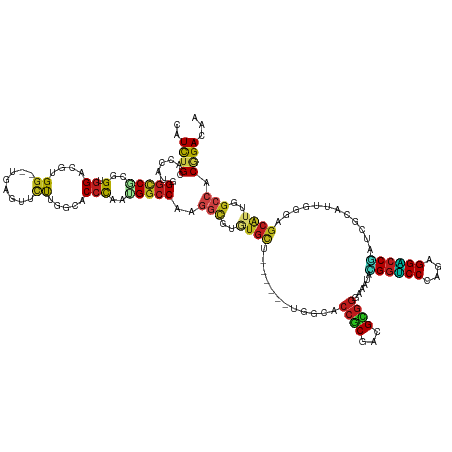
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:41:18 2011