
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 20,277,919 – 20,278,062 |
| Length | 143 |
| Max. P | 0.988421 |

| Location | 20,277,919 – 20,278,039 |
|---|---|
| Length | 120 |
| Sequences | 11 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.38 |
| Shannon entropy | 0.17656 |
| G+C content | 0.56591 |
| Mean single sequence MFE | -53.94 |
| Consensus MFE | -38.20 |
| Energy contribution | -38.15 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.71 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.784540 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 20277919 120 + 27905053 GCGAUGAUGCGGGUGGUGUUGUCGCUGGCGUUGAUUAUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCUAUGGUGCUGCUUUAUAGCAGCACACCACCUGU ........(((((((((......((((.((((((....((((.(.((.((((.......)))).)).)))))....)))))).))))....((((((((....))))))))))))))))) ( -58.80, z-score = -3.83, R) >droGri2.scaffold_14906 295462 120 + 14172833 GCGAUGAUGUGGAUGAUGUUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCCGCUUUACAGCAGCACGCAACCUUC ((......((.(((...))).))((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).))))....((((.(((....))).))))))....... ( -47.10, z-score = -0.63, R) >droMoj3.scaffold_6540 20652663 120 + 34148556 ACGAUGGUGCGGAUGAUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCCGCUUUAAAGCAGCACGCAGCCUUC ..((.((((((.....)))..((((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).))))....((((.(((....))).)))))).))).)) ( -51.20, z-score = -1.18, R) >droVir3.scaffold_13047 3639935 120 + 19223366 ACGAUGAUGUGGAUGAUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGAAGCAAUGGUGCCGCUUAAAAGCAGCACGCAAACUUC .......((((.........(((((((.(((((.......))))).)))))))((((((.....((((((.(((((...........))))))))))).....)))))).))))...... ( -44.00, z-score = -0.88, R) >droWil1.scaffold_181108 285409 120 - 4707319 GCGAUGAUGUGGAUGAUGCUGGCGCUGGCGUUGAUUAUGACGACGGCAGCGUUGUUGUUGCACUGCGGCGUCUAUUUUAAUGGCAGCUAUUGUGCUGCUUUACAGCAGCACUCAAUCUAC ........((((((((.......((((.((((((....((((.(.((((.((.......)).)))).)))))....)))))).))))....((((((((....)))))))))).)))))) ( -47.20, z-score = -2.40, R) >dp4.chr2 12270309 120 - 30794189 GCGAUGGUGUGGAUGGUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCUGCUGUAUAGCAGCACGCCACCUGC ((.((.((....)).))))((((((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).))))....((((((((....))))))))))))..... ( -56.10, z-score = -1.99, R) >droAna3.scaffold_13088 319749 120 + 569066 GCGAUGGUGCGGAUGGUGUUGUCGCUGGCGCUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCUGCUUUACAGCAGCACACCACCUAC (((....)))((.((((((.((.((((((((((...(((.(((((((......))))))))))..))))))..........(((((((....)))))))...)))).))))))))))... ( -52.80, z-score = -1.69, R) >droEre2.scaffold_4820 2688638 120 - 10470090 GCGAUGAUGCGGGUGGUGCUGUCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCUAUGGUGCUGCUUUACAGCAGCACACCACCUGU ........((((((((((.....((((.(((((.......))))).))))((((((((.((((((..(((((.........))).))..))))))......)))))))).)))))))))) ( -58.90, z-score = -3.14, R) >droYak2.chr3R 2606656 120 - 28832112 GCGAUGAUGCGGGUGGUGUUGUCGCUGGCGUUGAUUGUGACGACGACAGCGUUGCUGUUGCGCCGCGGCGUCUAUUGUAAUGGCAGCUAUGGUGCUGCUUUACAGCAGCACACCACCUGU ........(((((((((((.(((((.(((((.....(..(((.(....))))..)....))))))))))(.((.((((((.((((((......)))))))))))).)))))))))))))) ( -58.20, z-score = -3.22, R) >droSec1.super_0 20614460 120 + 21120651 GCGAUGAUGCGGGUGGUGUUGUCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCUAUGGUGCUGCUUUACAGCAGCACACCACCUGU ........(((((((((((....((((.(((((.......))))).))))((((((((.((((((..(((((.........))).))..))))))......))))))))))))))))))) ( -60.10, z-score = -3.75, R) >droSim1.chr3R 20118977 120 + 27517382 GCGAUGAUGCGGGUGGUGCUGUCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCUAUGGUGCUGCUUUACAGCAGCACACCACCUGU ........((((((((((.....((((.(((((.......))))).))))((((((((.((((((..(((((.........))).))..))))))......)))))))).)))))))))) ( -58.90, z-score = -3.14, R) >consensus GCGAUGAUGCGGAUGGUGCUGUCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCUAUGGUGCUGCUUUACAGCAGCACACCACCUGC ..............(((..((..((((.(((((.......))))).))))((((((((.((((((..(((((.........))).))..))))))......)))))))).))..)))... (-38.20 = -38.15 + -0.05)

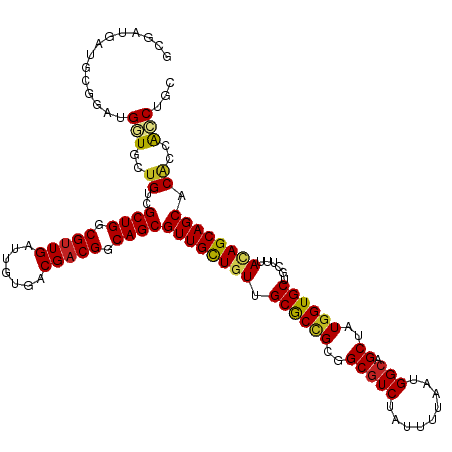
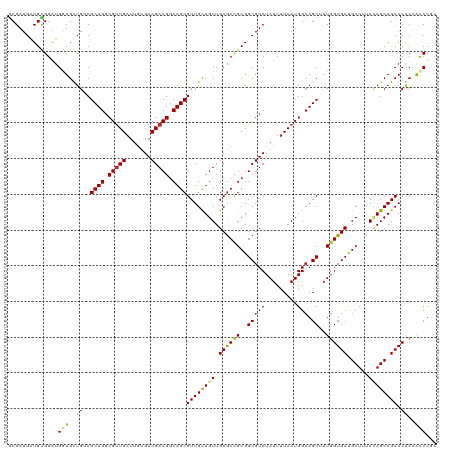
| Location | 20,277,919 – 20,278,039 |
|---|---|
| Length | 120 |
| Sequences | 11 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.38 |
| Shannon entropy | 0.17656 |
| G+C content | 0.56591 |
| Mean single sequence MFE | -43.15 |
| Consensus MFE | -36.25 |
| Energy contribution | -35.53 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.84 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.10 |
| SVM RNA-class probability | 0.982464 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 20277919 120 - 27905053 ACAGGUGGUGUGCUGCUAUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCAUAAUCAACGCCAGCGACAACACCACCCGCAUCAUCGC ...((((((((((((((....))))))......((((...........(((((.((((((.........)))))).).))))...........))))....))))))))........... ( -43.75, z-score = -3.11, R) >droGri2.scaffold_14906 295462 120 - 14172833 GAAGGUUGCGUGCUGCUGUAAAGCGGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAACAUCAUCCACAUCAUCGC ....((((.((((((.(((...((((((....))))))..........(((((.((((((.........)))))).).)))).......))).))))))))))................. ( -40.50, z-score = -2.00, R) >droMoj3.scaffold_6540 20652663 120 - 34148556 GAAGGCUGCGUGCUGCUUUAAAGCGGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCAUCAUCCGCACCAUCGU ((.((.(((((((((((....))))))))...(((((...........(((((.((((((.........)))))).).))))........((....)).)))))......))))).)).. ( -42.50, z-score = -2.00, R) >droVir3.scaffold_13047 3639935 120 - 19223366 GAAGUUUGCGUGCUGCUUUUAAGCGGCACCAUUGCUUCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCAUCAUCCACAUCAUCGU (((((.((.((((((((....))))))))))..)))))..........(((((.((((((.........)))))).).)))).............((....))................. ( -37.10, z-score = -2.37, R) >droWil1.scaffold_181108 285409 120 + 4707319 GUAGAUUGAGUGCUGCUGUAAAGCAGCACAAUAGCUGCCAUUAAAAUAGACGCCGCAGUGCAACAACAACGCUGCCGUCGUCAUAAUCAACGCCAGCGCCAGCAUCAUCCACAUCAUCGC ((.(((.((((((((((....))))))))....((((...........(((((.((((((.........)))))).).))))........((....)).)))).))))).))........ ( -38.10, z-score = -3.04, R) >dp4.chr2 12270309 120 + 30794189 GCAGGUGGCGUGCUGCUAUACAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCACCAUCCACACCAUCGC ...(.((((((((((((....))))))))....((((...........(((((.((((((.........)))))).).))))...........)))))))).)................. ( -42.55, z-score = -2.60, R) >droAna3.scaffold_13088 319749 120 - 569066 GUAGGUGGUGUGCUGCUGUAAAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCACAAUCAGCGCCAGCGACAACACCAUCCGCACCAUCGC ...(((((((((.((((((...((((((....))))))..........(.(((....)))).)))))).(((((.(((.(.......).))).)))))...........))))))))).. ( -46.60, z-score = -2.54, R) >droEre2.scaffold_4820 2688638 120 + 10470090 ACAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGACAGCACCACCCGCAUCAUCGC ...((((((((((((((....))))))))....((((...........(((((.((((((.........)))))).).))))........((....)).))))))))))........... ( -46.10, z-score = -2.82, R) >droYak2.chr3R 2606656 120 + 28832112 ACAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUACAAUAGACGCCGCGGCGCAACAGCAACGCUGUCGUCGUCACAAUCAACGCCAGCGACAACACCACCCGCAUCAUCGC ...((((((((((((.(((((.(((((......)))))..)))))...(.(((....))))..))))..(((((.(((.(.......).))).)))))...))))))))........... ( -45.90, z-score = -3.14, R) >droSec1.super_0 20614460 120 - 21120651 ACAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGACAACACCACCCGCAUCAUCGC ...((((((((..((((((...(((((......)))))..........(.(((....)))).)))))).(((((.(((.(.......).))).)))))...))))))))........... ( -45.50, z-score = -3.07, R) >droSim1.chr3R 20118977 120 - 27517382 ACAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGACAGCACCACCCGCAUCAUCGC ...((((((((((((((....))))))))....((((...........(((((.((((((.........)))))).).))))........((....)).))))))))))........... ( -46.10, z-score = -2.82, R) >consensus GCAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGACAGCACCAUCCGCAUCAUCGC ...(((.((((((((((....))))))))....((((...........(((((.((((((.........)))))).).))))...........))))....))))).............. (-36.25 = -35.53 + -0.72)
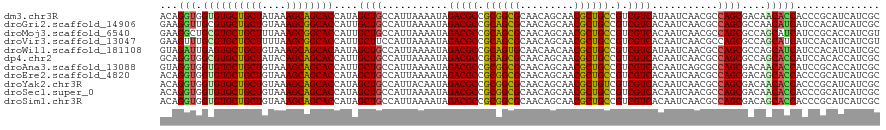
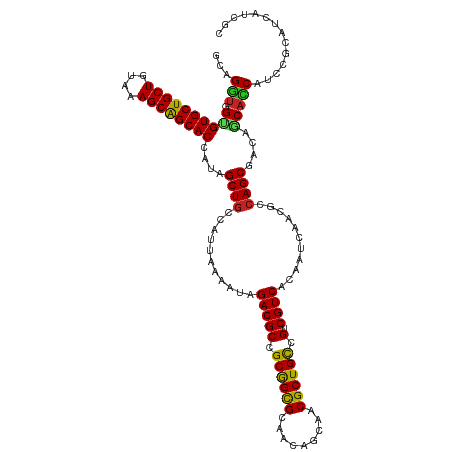
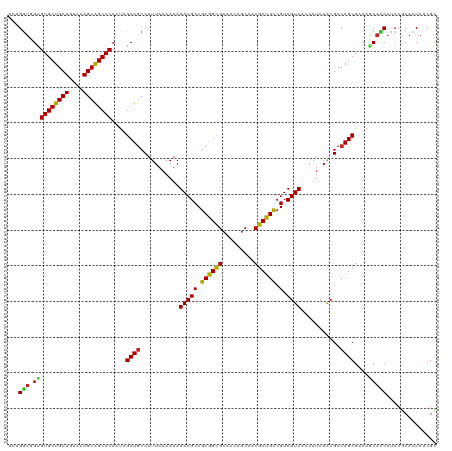
| Location | 20,277,959 – 20,278,062 |
|---|---|
| Length | 103 |
| Sequences | 12 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 80.37 |
| Shannon entropy | 0.42629 |
| G+C content | 0.55879 |
| Mean single sequence MFE | -38.49 |
| Consensus MFE | -32.41 |
| Energy contribution | -31.87 |
| Covariance contribution | -0.55 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.84 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.32 |
| SVM RNA-class probability | 0.988421 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
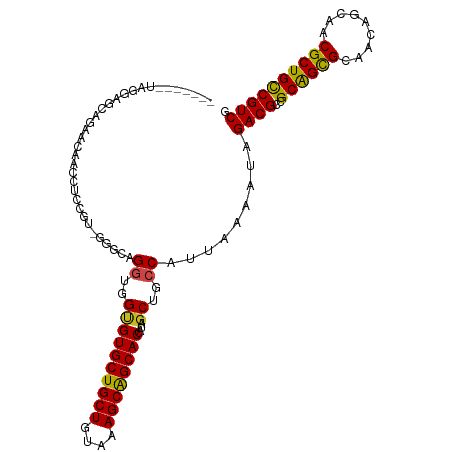
>dm3.chr3R 20277959 103 - 27905053 -------UAGGAGCAGGACAACCUCCGC-GGACAGGUGGUGUGCUGCUAUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCG -------.....((.(((.....)))))-((.(((.((..((((((((....))))))))..)).))))).........((((..((((((.........)))))))))). ( -39.30, z-score = -1.65, R) >droGri2.scaffold_14906 295502 107 - 14172833 ---UAGUUAACAGCACUUGUACCUCCAU-AAGAAGGUUGCGUGCUGCUGUAAAGCGGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCG ---.......(((((...((((((.(..-..).))).)))((((((((....))))))))...)))))...........((((..((((((.........)))))))))). ( -37.80, z-score = -1.95, R) >droMoj3.scaffold_6540 20652703 103 - 34148556 ------UAGUAGACCUACCUAAC-CCGU-GCGAAGGCUGCGUGCUGCUUUAAAGCGGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCG ------..((((.(((.((....-....-).).)))))))((((((((....))))))))...................((((..((((((.........)))))))))). ( -34.00, z-score = -0.96, R) >droVir3.scaffold_13047 3639975 101 - 19223366 ---UUGUAAAAAAC-----UACC-CCGU-GAGAAGUUUGCGUGCUGCUUUUAAGCGGCACCAUUGCUUCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCG ---...........-----....-..((-(.(((((.((.((((((((....))))))))))..)))))))).......((((..((((((.........)))))))))). ( -35.30, z-score = -3.26, R) >droWil1.scaffold_181108 285449 111 + 4707319 AUCGAGUAUAGUUCAAAUACAACUCCCUCGAGUAGAUUGAGUGCUGCUGUAAAGCAGCACAAUAGCUGCCAUUAAAAUAGACGCCGCAGUGCAACAACAACGCUGCCGUCG .(((((...((((.......))))..)))))((((.....((((((((....)))))))).....))))..........((((..((((((.........)))))))))). ( -37.00, z-score = -4.05, R) >droPer1.super_3 6120367 105 + 7375914 ------CAGAGCCCAGAGUAGAUACAAGUGAGCAGGUGGCGUGCUGCUAUACAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCG ------....((.((..((....))...)).)).((..((((((((((....))))))))....))..)).........((((..((((((.........)))))))))). ( -36.90, z-score = -1.67, R) >dp4.chr2 12270349 105 + 30794189 ------CAGAGCCCAGAGUAGAUACAAGUGAGCAGGUGGCGUGCUGCUAUACAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCG ------....((.((..((....))...)).)).((..((((((((((....))))))))....))..)).........((((..((((((.........)))))))))). ( -36.90, z-score = -1.67, R) >droAna3.scaffold_13088 319789 100 - 569066 ---------GGGGCA-ACAAGACUCUGU-GGGUAGGUGGUGUGCUGCUGUAAAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCG ---------..(((.-.....(((....-.))).((..((((((((((....))))))).)))..))))).........((((..((((((.........)))))))))). ( -39.40, z-score = -0.90, R) >droEre2.scaffold_4820 2688678 103 + 10470090 -------UAGGAGCAGGACAACCUCCGU-GGACAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCG -------..((((.........))))((-((.(((.((..((((((((....))))))))..)).))))))).......((((..((((((.........)))))))))). ( -41.40, z-score = -1.94, R) >droYak2.chr3R 2606696 103 + 28832112 -------UAGGAGCAGGACAACCUCCGU-GGACAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUACAAUAGACGCCGCGGCGCAACAGCAACGCUGUCGUCG -------..((((.........))))((-((.(((.((..((((((((....))))))))..)).))))))).......(((((.(((..((....))..))).).)))). ( -41.00, z-score = -1.78, R) >droSec1.super_0 20614500 103 - 21120651 -------UGGGAGCAGGACAACCUCCGC-GGACAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCG -------(((.(((......(((.((..-.....)).)))((((((((....))))))))....))).)))........((((..((((((.........)))))))))). ( -42.70, z-score = -1.74, R) >droSim1.chr3R 20119017 103 - 27517382 -------UAGGAGCAGGACAACCUCCGC-GGACAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCG -------.....((.(((.....)))))-((.(((.((..((((((((....))))))))..)).))))).........((((..((((((.........)))))))))). ( -40.20, z-score = -1.43, R) >consensus _______UAGGAGCAGAACAACCUCCGU_GGGCAGGUGGUGUGCUGCUGUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCG ..................................((..((((((((((....))))))))....))..)).........((((..((((((.........)))))))))). (-32.41 = -31.87 + -0.55)

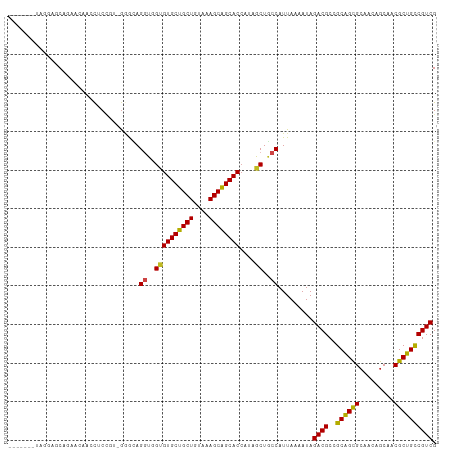
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:40:38 2011