
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 20,227,327 – 20,227,470 |
| Length | 143 |
| Max. P | 0.999253 |

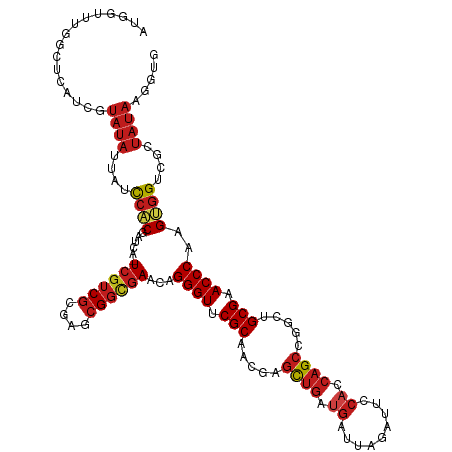
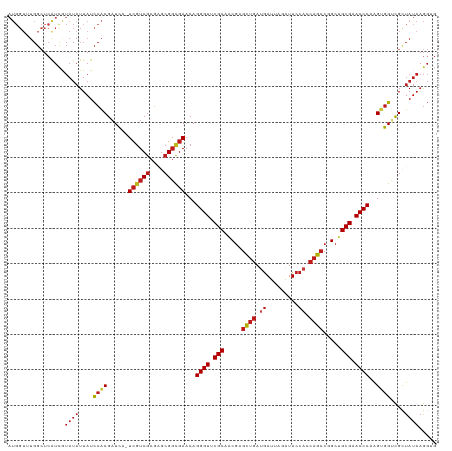
| Location | 20,227,327 – 20,227,447 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 121 |
| Reading direction | reverse |
| Mean pairwise identity | 81.59 |
| Shannon entropy | 0.42253 |
| G+C content | 0.53265 |
| Mean single sequence MFE | -41.13 |
| Consensus MFE | -28.00 |
| Energy contribution | -28.12 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.68 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.39 |
| SVM RNA-class probability | 0.935313 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
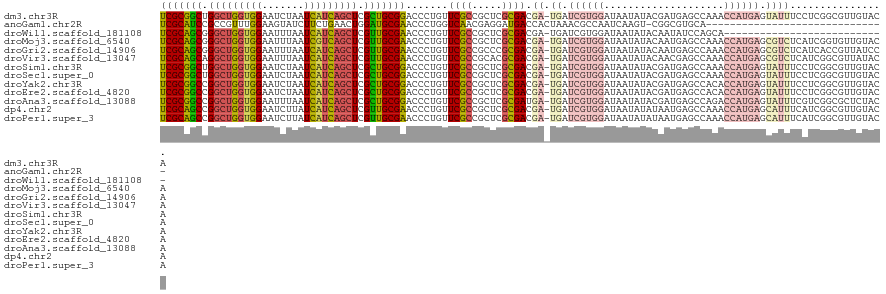
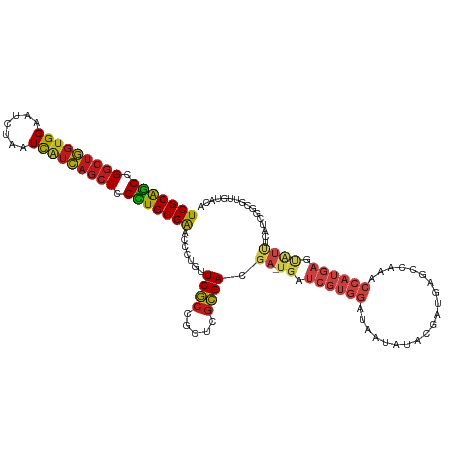
>dm3.chr3R 20227327 120 - 27905053 UCGCGGCUGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCAAACCAUGAGUAUUUCCUCGGCGUUGUACA .(((((..(((((((((.......))))))))).((.((((((...)))))).)))))))((((-((.(((.(((....((((.(((........)))..)))).))).)))))))))... ( -41.60, z-score = -1.04, R) >anoGam1.chr2R 32246982 90 + 62725911 UCGCAUCCGCCGUUUGGAAGUAUCUUCUGAACUGGAUGCGAACCCUGGUCAACGAGGAUGACCACUAAACGCCAAUCAAGU-CGGCGUGCA------------------------------ (((((((((..(((..((((...))))..))))))))))))....((((((.......))))))....(((((........-.)))))...------------------------------ ( -35.30, z-score = -3.58, R) >droWil1.scaffold_181108 229624 93 + 4707319 UCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACAAUAUCCAGCA--------------------------- (((((((((((((((((.......)))))))))))))))))............(((.((((...-...))))(((((........)))))))).--------------------------- ( -37.50, z-score = -3.34, R) >droMoj3.scaffold_6540 20581296 120 - 34148556 UCGCAGCGGGCUGGUGGAAUUUAAUCGUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACAAUGAGCCAAACCAUGAGCGUCUCAUCGGUGUUGUACA (((((((((((((..((.......))..))))))))))))).....((.(((((...(.((((.-...((((((....................)))))).)))).)..)))))....)). ( -43.35, z-score = -1.72, R) >droGri2.scaffold_14906 236051 120 - 14172833 UCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCCCGCGACGA-UGAUCGUGGAUAAUAUACAAUGAGCCAAACCAUGAGCGUCUCAUCACCGUUAUCCA (((((((((((((((((.......)))))))))))))))))........(((.....)))(((.-....)))(((((((.....(((((.(...........).)))))....))))))). ( -41.70, z-score = -2.32, R) >droVir3.scaffold_13047 3573704 120 - 19223366 UCGCAGCAGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCACGCGACGA-UGAUCGUGGAUAAUAUACAACGAGCCAAACCAUGAGCGUCUCAUCGGCGUUAUACA (((((((..((((((((.......))))))))..))))))).....((.(((((...(.((((.-...((((((....................)))))).)))).)..)))))....)). ( -42.95, z-score = -2.45, R) >droSim1.chr3R 20065650 120 - 27517382 UCGCGGCUGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCAAACCAUGAGUAUUUCCUCGGCGUUGUACA .(((((..(((((((((.......))))))))).((.((((((...)))))).)))))))((((-((.(((.(((....((((.(((........)))..)))).))).)))))))))... ( -41.60, z-score = -1.04, R) >droSec1.super_0 20565876 120 - 21120651 UCGCGGCUGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCAAACCAUGAGUAUUUCCUCGGCGUUGUACA .(((((..(((((((((.......))))))))).((.((((((...)))))).)))))))((((-((.(((.(((....((((.(((........)))..)))).))).)))))))))... ( -41.60, z-score = -1.04, R) >droYak2.chr3R 2554998 120 + 28832112 UCGCGGCCGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCACACCAUGAGUAUUUCCUCGGCGUUGUACA .(((((..(((((((((.......))))))))).((.((((((...)))))).)))))))((((-((.(((.(((....((((.(((........)))..)))).))).)))))))))... ( -41.70, z-score = -0.66, R) >droEre2.scaffold_4820 2637592 120 + 10470090 UCGCGGCCGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCACACCAUGAGUAUUUCCUCGGCGUUGUACA .(((((..(((((((((.......))))))))).((.((((((...)))))).)))))))((((-((.(((.(((....((((.(((........)))..)))).))).)))))))))... ( -41.70, z-score = -0.66, R) >droAna3.scaffold_13088 268315 120 - 569066 UCGCGGCCGGCUGGUGGAAUUUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGAUGA-UGAUCGUGGAUAAUAUACGAUGAGCCAGACCAUGAGUAUUUCGUCGGCGCUCUACA (((((((.(((((((((.......))))))))).))))))).....(..((((..(((((((..-..))))))).........(((((((((.....)).))...)))))))))..).... ( -42.70, z-score = -0.71, R) >dp4.chr2 12218517 120 + 30794189 UCGCAGCCGGCUGGUGGAAUCUUAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUAUAAUGAGCCAAACCAUGAGCAUUUCAUCGGCGUUGUACA ..(((((..((((((((((((((((.....(((((((((((((...))))))...((((((...-...)))))).........)))))))......))))).))))))))))))))))... ( -41.50, z-score = -1.83, R) >droPer1.super_3 6068580 120 + 7375914 UCGCAGCCGGCUGGUGGAAUCUUAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUAUAAUGAGCCAAACCAUGAGCAUUUCAUCGGCGUUGUACA ..(((((..((((((((((((((((.....(((((((((((((...))))))...((((((...-...)))))).........)))))))......))))).))))))))))))))))... ( -41.50, z-score = -1.83, R) >consensus UCGCAGCCGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGAACCCUGUUCGCCGCUCGCGACGA_UGAUCGUGGAUAAUAUACGAUGAGCCAAACCAUGAGUAUUUCAUCGGCGUUGUACA (((((((.(((((((((.......))))))))).))))))).......((((.....)))).......((((((....................))))))..................... (-28.00 = -28.12 + 0.11)
| Location | 20,227,350 – 20,227,470 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 121 |
| Reading direction | forward |
| Mean pairwise identity | 86.02 |
| Shannon entropy | 0.32658 |
| G+C content | 0.53562 |
| Mean single sequence MFE | -42.41 |
| Consensus MFE | -26.55 |
| Energy contribution | -27.53 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.63 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.815537 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
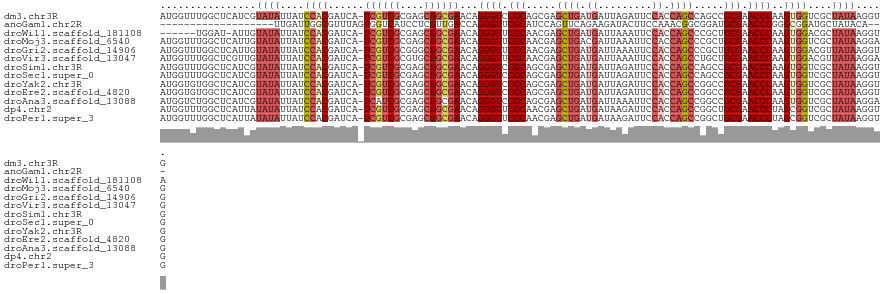
>dm3.chr3R 20227350 120 + 27905053 AUGGUUUGGCUCAUCGUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUCCGCAGCGAGCUGAUGAUUAGAUUCCACCAGCCAGCCGCGAACCCAAGUGGUCGCUAUAAGGUG ...........((((.((((....((((.....-((((((....))))))...((((.(((.((..((((.((.........)).))))..)).))).))))..))))....)))).)))) ( -40.80, z-score = -1.20, R) >anoGam1.chr2R 32246993 99 - 62725911 -------------------UUGAUUGGCGUUUAGUGGUCAUCCUCGUUGACCAGGGUUCGCAUCCAGUUCAGAAGAUACUUCCAAACGGCGGAUGCGAACCCGGGCGGAUGCUAUACA--- -------------------.....((((((((..((((((.......))))))((((((((((((.(((..((((...))))..)))...))))))))))))....))))))))....--- ( -43.50, z-score = -4.36, R) >droWil1.scaffold_181108 229627 113 - 4707319 ------UGGAU-AUUGUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGACGCUAUAAGGUA ------....(-((..((((.(.(((((.....-((((((....))))))...(((((((((.((.((((.((.........)).)))).)).)))))))))..))))).).))))..))) ( -44.80, z-score = -4.24, R) >droMoj3.scaffold_6540 20581319 120 + 34148556 AUGGUUUGGCUCAUUGUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGACGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGUCGCUAUAAGGAG .........(((.(((((......((((.....-((((((....))))))...(((((((((.((.((((..((......))...)))).)).)))))))))..))))....))))).))) ( -44.50, z-score = -2.94, R) >droGri2.scaffold_14906 236074 120 + 14172833 AUGGUUUGGCUCAUUGUAUAUUAUCCACGAUCA-UCGUCGCGGGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGACGUUAUAAGGUG ...........(((..((((...(((((.....-((((((....))))))...(((((((((.((.((((.((.........)).)))).)).)))))))))..)))))...))))..))) ( -46.60, z-score = -3.20, R) >droVir3.scaffold_13047 3573727 120 + 19223366 AUGGUUUGGCUCGUUGUAUAUUAUCCACGAUCA-UCGUCGCGUGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCUGCUGCGAACCCAAGUGGACGUUAUAAGGAG .........(((.(((((.....(((((.....-((((((....))))))...(((((((((.((.((((.((.........)).)))).)).)))))))))..)))))...))))).))) ( -47.00, z-score = -3.58, R) >droSim1.chr3R 20065673 120 + 27517382 AUGGUUUGGCUCAUCGUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUCCGCAGCGAGCUGAUGAUUAGAUUCCACCAGCCAGCCGCGAACCCAAGUGGUCGCUAUAAGGUG ...........((((.((((....((((.....-((((((....))))))...((((.(((.((..((((.((.........)).))))..)).))).))))..))))....)))).)))) ( -40.80, z-score = -1.20, R) >droSec1.super_0 20565899 120 + 21120651 AUGGUUUGGCUCAUCGUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUCCGCAGCGAGCUGAUGAUUAGAUUCCACCAGCCAGCCGCGAACCCAAGUGGUCGCUAUAAGGUG ...........((((.((((....((((.....-((((((....))))))...((((.(((.((..((((.((.........)).))))..)).))).))))..))))....)))).)))) ( -40.80, z-score = -1.20, R) >droYak2.chr3R 2555021 120 - 28832112 AUGGUGUGGCUCAUCGUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUCCGCAGCGAGCUGAUGAUUAGAUUCCACCAGCCGGCCGCGAACCCAAGUGGUCGCUAUAAGGUG (((((((((...............))))(((((-((((((....))))))...((((.(((.((..((((.((.........)).))))..)).))).))))...))))))))))...... ( -40.56, z-score = -0.33, R) >droEre2.scaffold_4820 2637615 120 - 10470090 AUGGUGUGGCUCAUCGUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUCCGCAGCGAGCUGAUGAUUAGAUUCCACCAGCCGGCCGCGAACCCAAGUGGUCGCUAUAAGGUG (((((((((...............))))(((((-((((((....))))))...((((.(((.((..((((.((.........)).))))..)).))).))))...))))))))))...... ( -40.56, z-score = -0.33, R) >droAna3.scaffold_13088 268338 120 + 569066 AUGGUCUGGCUCAUCGUAUAUUAUCCACGAUCA-UCAUCGCGAGCGGCGAACAGGGUCCGCAGCGAGCUGAUGAUUAAAUUCCACCAGCCGGCCGCGAACCCAAGUGGUCGCUAUAAGGAG ..((.((((((..((((.........))))(((-(((((((..(((((.......).)))).))))..))))))............))))))))((((.((.....))))))......... ( -38.40, z-score = -0.52, R) >dp4.chr2 12218540 120 - 30794189 AUGGUUUGGCUCAUUAUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUAAGAUUCCACCAGCCGGCUGCGAACCCUAGCGGUCGCUAUAAGGUG (((((.(((...((.....))...))).((((.-((((((....))))))..((((((((((.((.((((.((.........)).))))))..))))))))))...)))))))))...... ( -41.50, z-score = -1.62, R) >droPer1.super_3 6068603 120 - 7375914 AUGGUUUGGCUCAUUAUAUAUUAUCCACGAUCA-UCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUAAGAUUCCACCAGCCGGCUGCGAACCCUAGCGGUCGCUAUAAGGUG (((((.(((...((.....))...))).((((.-((((((....))))))..((((((((((.((.((((.((.........)).))))))..))))))))))...)))))))))...... ( -41.50, z-score = -1.62, R) >consensus AUGGUUUGGCUCAUCGUAUAUUAUCCACGAUCA_UCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAGAUUCCACCAGCCGGCUGCGAACCCAAGUGGUCGCUAUAAGGUG ................((((....((((......((((((....))))))...((((.(((.....((((.((.........)).)))).....))).))))..))))....))))..... (-26.55 = -27.53 + 0.98)
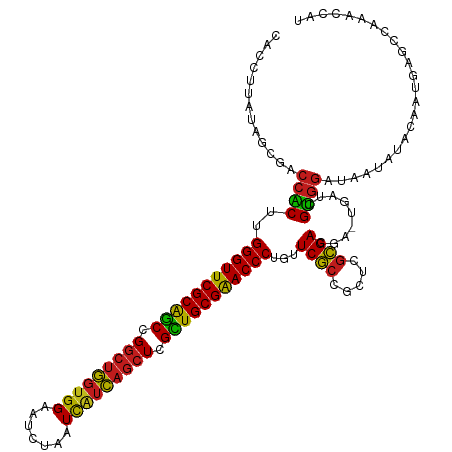
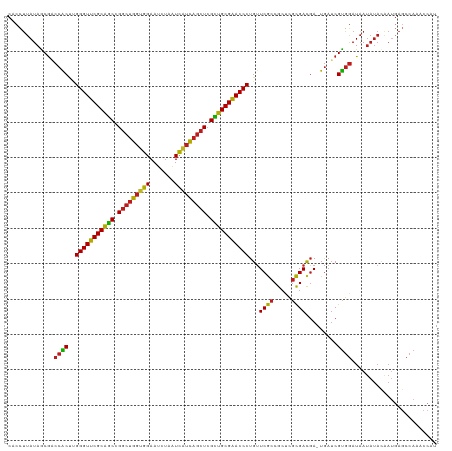
| Location | 20,227,350 – 20,227,470 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 121 |
| Reading direction | reverse |
| Mean pairwise identity | 86.02 |
| Shannon entropy | 0.32658 |
| G+C content | 0.53562 |
| Mean single sequence MFE | -48.12 |
| Consensus MFE | -42.21 |
| Energy contribution | -41.31 |
| Covariance contribution | -0.90 |
| Combinations/Pair | 1.36 |
| Mean z-score | -3.99 |
| Structure conservation index | 0.88 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.74 |
| SVM RNA-class probability | 0.999253 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 20227350 120 - 27905053 CACCUUAUAGCGACCACUUGGGUUCGCGGCUGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCAAACCAU .........((((.((...(((((((((((.(((((((((.......))))))))).))))))))))))).)))).((((.......-..((((((.......))))))))))........ ( -47.60, z-score = -3.24, R) >anoGam1.chr2R 32246993 99 + 62725911 ---UGUAUAGCAUCCGCCCGGGUUCGCAUCCGCCGUUUGGAAGUAUCUUCUGAACUGGAUGCGAACCCUGGUCAACGAGGAUGACCACUAAACGCCAAUCAA------------------- ---.((.(((((((((((.(((((((((((((..(((..((((...))))..)))))))))))))))).)))......)))))....))).)).........------------------- ( -42.60, z-score = -5.22, R) >droWil1.scaffold_181108 229627 113 + 4707319 UACCUUAUAGCGUCCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACAAU-AUCCA------ .....((((..((((((..(((((((((((((((((((((.......)))))))))))))))))))))...(((.((....)).)))-.....))))))..))))....-.....------ ( -52.90, z-score = -6.31, R) >droMoj3.scaffold_6540 20581319 120 - 34148556 CUCCUUAUAGCGACCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCGUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACAAUGAGCCAAACCAU ...(((((.((((.((...(((((((((((((((((..((.......))..))))))))))))))))))).))))...((((((...-...))))))..........)))))......... ( -48.40, z-score = -3.90, R) >droGri2.scaffold_14906 236074 120 - 14172833 CACCUUAUAACGUCCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCCCGCGACGA-UGAUCGUGGAUAAUAUACAAUGAGCCAAACCAU .....((((..((((((..(((((((((((((((((((((.......)))))))))))))))))))))...(((.((....)).)))-.....))))))..))))................ ( -51.60, z-score = -5.37, R) >droVir3.scaffold_13047 3573727 120 - 19223366 CUCCUUAUAACGUCCACUUGGGUUCGCAGCAGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCACGCGACGA-UGAUCGUGGAUAAUAUACAACGAGCCAAACCAU (((..((((..((((((..(((((((((((..((((((((.......))))))))..)))))))))))...(((.((....)).)))-.....))))))..))))....)))......... ( -47.70, z-score = -4.81, R) >droSim1.chr3R 20065673 120 - 27517382 CACCUUAUAGCGACCACUUGGGUUCGCGGCUGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCAAACCAU .........((((.((...(((((((((((.(((((((((.......))))))))).))))))))))))).)))).((((.......-..((((((.......))))))))))........ ( -47.60, z-score = -3.24, R) >droSec1.super_0 20565899 120 - 21120651 CACCUUAUAGCGACCACUUGGGUUCGCGGCUGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCAAACCAU .........((((.((...(((((((((((.(((((((((.......))))))))).))))))))))))).)))).((((.......-..((((((.......))))))))))........ ( -47.60, z-score = -3.24, R) >droYak2.chr3R 2555021 120 + 28832112 CACCUUAUAGCGACCACUUGGGUUCGCGGCCGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCACACCAU .........((((.((...(((((((((((.(((((((((.......))))))))).))))))))))))).)))).((((.......-..((((((.......))))))))))........ ( -47.60, z-score = -2.89, R) >droEre2.scaffold_4820 2637615 120 + 10470090 CACCUUAUAGCGACCACUUGGGUUCGCGGCCGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUACGAUGAGCCACACCAU .........((((.((...(((((((((((.(((((((((.......))))))))).))))))))))))).)))).((((.......-..((((((.......))))))))))........ ( -47.60, z-score = -2.89, R) >droAna3.scaffold_13088 268338 120 - 569066 CUCCUUAUAGCGACCACUUGGGUUCGCGGCCGGCUGGUGGAAUUUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGAUGA-UGAUCGUGGAUAAUAUACGAUGAGCCAGACCAU ...(((((.((((.((...(((((((((((.(((((((((.......))))))))).))))))))))))).))))...(((((((..-..)))))))..........)))))......... ( -48.80, z-score = -3.08, R) >dp4.chr2 12218540 120 + 30794189 CACCUUAUAGCGACCGCUAGGGUUCGCAGCCGGCUGGUGGAAUCUUAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUAUAAUGAGCCAAACCAU ...(((((.((((..((.((((((((((((.(((((((((.......))))))))).))))))))))))))))))...((((((...-...))))))..........)))))......... ( -47.80, z-score = -3.86, R) >droPer1.super_3 6068603 120 + 7375914 CACCUUAUAGCGACCGCUAGGGUUCGCAGCCGGCUGGUGGAAUCUUAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGA-UGAUCGUGGAUAAUAUAUAAUGAGCCAAACCAU ...(((((.((((..((.((((((((((((.(((((((((.......))))))))).))))))))))))))))))...((((((...-...))))))..........)))))......... ( -47.80, z-score = -3.86, R) >consensus CACCUUAUAGCGACCACUUGGGUUCGCAGCCGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGAACCCUGUUCGCCGCUCGCGACGA_UGAUCGUGGAUAAUAUACAAUGAGCCAAACCAU .............((((..(((((((((((.(((((((((.......))))))))).)))))))))))...((((.....)))).........))))........................ (-42.21 = -41.31 + -0.90)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:40:29 2011