
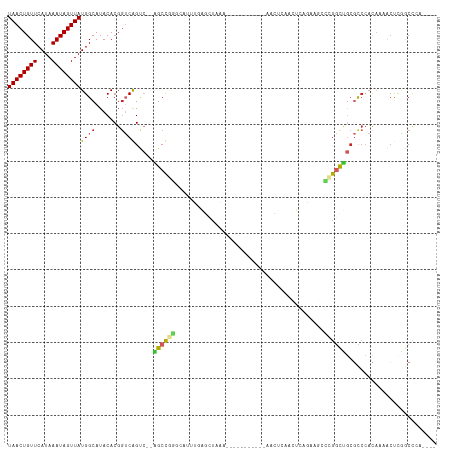
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 19,282,890 – 19,283,029 |
| Length | 139 |
| Max. P | 0.945829 |

| Location | 19,282,890 – 19,282,991 |
|---|---|
| Length | 101 |
| Sequences | 10 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 70.32 |
| Shannon entropy | 0.56734 |
| G+C content | 0.49448 |
| Mean single sequence MFE | -32.77 |
| Consensus MFE | -9.65 |
| Energy contribution | -9.52 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.83 |
| Structure conservation index | 0.29 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.51 |
| SVM RNA-class probability | 0.945829 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 19282890 101 - 27905053 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCGGUC--AGCCGGGCAUUUGAGCUAAA-----------AACUCAACUCAAAAGCCCGGCUGCGCCCACAAAACUUGGCUCA---- ((((((((....)))))))).(((.......((.((((--((((((((.((((((.....-----------.......)))))).))))))))).))).))........)))..---- ( -38.16, z-score = -5.05, R) >droSim1.chr3R 19102847 101 - 27517382 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCGGUC--AGCCGGGCAUUUGAGCUAAA-----------AACUCAACUCAAAAGCCCGGCUGCGCCCACAAAACUUGGCUCA---- ((((((((....)))))))).(((.......((.((((--((((((((.((((((.....-----------.......)))))).))))))))).))).))........)))..---- ( -38.16, z-score = -5.05, R) >droSec1.super_0 19646055 101 - 21120651 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCGGUC--AGCCGGGCAUUUGAGCUAAA-----------AACUCAACUCAGAAGCCCGGCUGCGCCCACAAAACUUGGCUCA---- ((((((((....)))))))).(((.......((.((((--((((((((.((((((.....-----------.......)))))).))))))))).))).))........)))..---- ( -38.26, z-score = -4.62, R) >droYak2.chr3R 20166008 114 - 28832112 UAACUGUUCAUAAAUAGUUAUGGCAUACACGCUCGGUC--AGCCGGGCAUUUGAGCUUAAAAACUCAGCUCAGCUCAACUCAAAAACCCGGCUGCGCCCACAAAACUUGGCUUAAG-- ((((((((....)))))))).(((..........((((--(((((((...(((((((..............)))))))........)))))))).)))...........)))....-- ( -30.94, z-score = -2.30, R) >droEre2.scaffold_4820 1703511 101 + 10470090 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCGGUC--AGCCGGGCAUUUGAGCUAAA-----------AACUCAACUCAAAAGCCCGGCUGCGCCCACAAAACUUGGCUCA---- ((((((((....)))))))).(((.......((.((((--((((((((.((((((.....-----------.......)))))).))))))))).))).))........)))..---- ( -38.16, z-score = -5.05, R) >droAna3.scaffold_13340 3426487 90 + 23697760 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCAG-CCGGGCUAAAAA-----------------------ACCCAGUUCAG-UGCUGGGCUGCGCCCACAAAACUCAGCCAAA--- ((((((((....))))))))((((......((....-))((((......-----------------------.((((((....-.))))))....))))..........))))..--- ( -29.50, z-score = -2.78, R) >dp4.chr2 2766286 104 - 30794189 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCAGUCCGAGCUCGGGA-UCGGGCU-GA-----------GGUUCUGUUCAC-UGCUGGGCUGCGCCCACAAAACUCAACCAAAGUU ((((((((....))))))))............(((((((((........-)))))))-))-----------((((..(((...-((.(((((...))))))).)))..))))...... ( -32.20, z-score = -1.53, R) >droPer1.super_7 2859516 104 + 4445127 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCAGUCCGAGCUCGGGA-UCGGGCU-GG-----------GGUUCUGUUCAG-UGCUGGGCUGCGCCCACAAAACUCAACCAAAGUU ((((((((....))))))))..........(((((((((((........-)))))))-)(-----------((((.(((...(-(((......))))..))).))))).)))...... ( -32.40, z-score = -1.16, R) >droVir3.scaffold_13047 8915534 98 - 19223366 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGACAGC---AGCUGCGCCCACAAAGUUG----------CCACUGCAACUCAGUU--CAGACCAGCCAGAUCCAAGCCGGCCC----- ((((((((....)))))))).(((......(((....---.((((.....((..(((((----------(....))))))..)).--.....))))....)))......))).----- ( -22.80, z-score = -0.52, R) >droMoj3.scaffold_6540 2397763 98 + 34148556 UAACUGUUCAUAAAUAGUUAUGGCAUACACGGACAGC---GGCUGCGCCCACAAAGUUG----------CCACUGCAGCUCGGCU--CGGCUCACCUCGACUAGGCCCUACCC----- ((((((((....)))))))).(((.....(((.....---(((((.(((.....(((((----------(....)))))).))).--)))))....))).....)))......----- ( -27.10, z-score = -0.22, R) >consensus UAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCAGUC__AGCCGGGCAUUUGAGCUAAA___________AACUCAACUCAGAAGCCCGGCUGCGCCCACAAAACUCGGCCCA____ ((((((((....))))))))((((.......)))).....((((((.........................................))))))......................... ( -9.65 = -9.52 + -0.13)


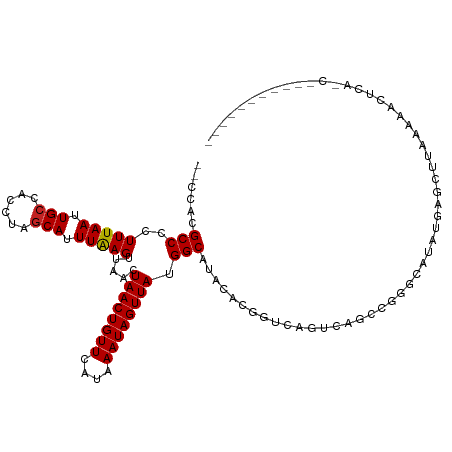
| Location | 19,282,924 – 19,283,029 |
|---|---|
| Length | 105 |
| Sequences | 11 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 78.65 |
| Shannon entropy | 0.42563 |
| G+C content | 0.44408 |
| Mean single sequence MFE | -26.05 |
| Consensus MFE | -13.11 |
| Energy contribution | -13.00 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.50 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.505862 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 19282924 105 - 27905053 --CCACGCCCCUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCGGUCAGCCGGGCAUUUGAGC-UAAAAACUCAACU---------- --....((((.......(((((..((((......))))..((((((((....))))))))))))).....(((......)))))))..(((((.-......)))))..---------- ( -26.40, z-score = -2.12, R) >droSim1.chr3R 19102881 105 - 27517382 --CCACGCCCCUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCGGUCAGCCGGGCAUUUGAGC-UAAAAACUCAACU---------- --....((((.......(((((..((((......))))..((((((((....))))))))))))).....(((......)))))))..(((((.-......)))))..---------- ( -26.40, z-score = -2.12, R) >droSec1.super_0 19646089 105 - 21120651 --CCACGCCCCUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCGGUCAGCCGGGCAUUUGAGC-UAAAAACUCAACU---------- --....((((.......(((((..((((......))))..((((((((....))))))))))))).....(((......)))))))..(((((.-......)))))..---------- ( -26.40, z-score = -2.12, R) >droYak2.chr3R 20166044 118 - 28832112 AGCCACGCCCCUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGCUCGGUCAGCCGGGCAUUUGAGCUUAAAAACUCAGCUCAGCUCAACU .((((......(((((.(((......))).))))).....((((((((....))))))))))))......((((((....))))))..(((((((.........)))))))....... ( -32.70, z-score = -3.00, R) >droEre2.scaffold_4820 1703545 105 + 10470090 --CCACGCCCCUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCGGUCAGCCGGGCAUUUGAGC-UAAAAACUCAACU---------- --....((((.......(((((..((((......))))..((((((((....))))))))))))).....(((......)))))))..(((((.-......)))))..---------- ( -26.40, z-score = -2.12, R) >droAna3.scaffold_13340 3426521 96 + 23697760 AGCCACGCCCAUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCAGCCGGGCUAAAAAACCCAGUU---------------------- ((((..(((.((((((.(((......))).))))))....((((((((....)))))))).))).....(((....))))))).............---------------------- ( -24.60, z-score = -2.42, R) >dp4.chr2 2766323 105 - 30794189 --CCACGCCCCUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCAGUCCGAGCUCGGGAUCGGGCUGAGGUUCUGUU----------- --....(((((......(((((..((((......))))..((((((((....))))))))))))).....))(((((((((........))))))))))))......----------- ( -30.00, z-score = -1.65, R) >droPer1.super_7 2859553 105 + 4445127 --CCCCGCCCCUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCAGUCCGAGCUCGGGAUCGGGCUGGGGUUCUGUU----------- --((((((((.......(((((..((((......))))..((((((((....))))))))))))).....((((...(((....))))))))))).)))).......----------- ( -34.30, z-score = -2.13, R) >droVir3.scaffold_13047 8915573 97 - 19223366 --CCACGCCCAUUUAAUUGCCACCUAACAUUUGAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGACAG-CAGCUGCGCCCACAAAGUUGCCA------------------ --....(.((.......(((((..((((......))))..((((((((....))))))))))))).....)).).(-(((((..........))))))..------------------ ( -19.90, z-score = -0.76, R) >droMoj3.scaffold_6540 2397802 97 + 34148556 --CCACGCCCAUUUAAUUGCCACCUAGCAUUUGAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGACAG-CGGCUGCGCCCACAAAGUUGCCA------------------ --....(((.((((((.(((......))).))))))....((((((((....)))))))).)))......((.(((-((((...)))......)))))).------------------ ( -21.20, z-score = -0.22, R) >droGri2.scaffold_15074 7025871 97 + 7742996 --CCACGCCCAUUUAAUUGCCACCUAGCAUUUGAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGACAG-CGGCUGCGCCCACAAAAUUGCCA------------------ --........((((((.(((......))).))))))....((((((((....))))))))(((((.....((...(-((....))))).......)))))------------------ ( -18.30, z-score = 0.44, R) >consensus __CCACGCCCCUUUAAUUGCCACCUAGCAUUUAAGUUAACUAACUGUUCAUAAAUAGUUAUGGCAUACACGGUCAGUCAGCCGGGCAUAUGAGCUUAAAAACUCA_C___________ ......(((..(((((.(((......))).))))).....((((((((....)))))))).)))...................................................... (-13.11 = -13.00 + -0.11)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:37:59 2011