
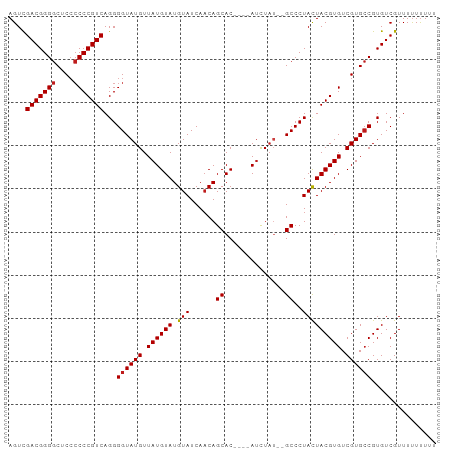
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 18,299,445 – 18,299,538 |
| Length | 93 |
| Max. P | 0.814515 |

| Location | 18,299,445 – 18,299,538 |
|---|---|
| Length | 93 |
| Sequences | 13 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 94.53 |
| Shannon entropy | 0.11862 |
| G+C content | 0.50065 |
| Mean single sequence MFE | -29.37 |
| Consensus MFE | -24.94 |
| Energy contribution | -25.02 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.85 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.814515 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 18299445 93 + 27905053 AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAU--GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUUU ...(((((.(((.(..(.(((..((((((((..((((.(((....)))..))----)).)))--)))))...))).)..).))).)))))......... ( -30.30, z-score = -2.31, R) >droPer1.super_0 6666260 93 + 11822988 AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAUU-GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUU- ...(((((.(((.(..(.(((..((((((....((((.(((....)))..))----))....)-)))))...))).)..).))).)))))........- ( -28.90, z-score = -1.84, R) >dp4.chr2 17013568 93 - 30794189 AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAUU-GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUU- ...(((((.(((.(..(.(((..((((((....((((.(((....)))..))----))....)-)))))...))).)..).))).)))))........- ( -28.90, z-score = -1.84, R) >droAna3.scaffold_12911 5340336 93 + 5364042 AGUUGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAA--GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUUU ..(((((((((....))))))))).((((((.((((((.(((......((..----......--))..))))))))).))))))............... ( -28.80, z-score = -1.96, R) >droEre2.scaffold_4820 691997 93 - 10470090 AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAU--GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUUU ...(((((.(((.(..(.(((..((((((((..((((.(((....)))..))----)).)))--)))))...))).)..).))).)))))......... ( -30.30, z-score = -2.31, R) >droYak2.chr3R 19144887 93 + 28832112 AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAU--GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUUU ...(((((.(((.(..(.(((..((((((((..((((.(((....)))..))----)).)))--)))))...))).)..).))).)))))......... ( -30.30, z-score = -2.31, R) >droSec1.super_0 18653857 93 + 21120651 AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAU--GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUUU ...(((((.(((.(..(.(((..((((((((..((((.(((....)))..))----)).)))--)))))...))).)..).))).)))))......... ( -30.30, z-score = -2.31, R) >droSim1.chr3R 18107955 93 + 27517382 AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAU--GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUUU ...(((((.(((.(..(.(((..((((((((..((((.(((....)))..))----)).)))--)))))...))).)..).))).)))))......... ( -30.30, z-score = -2.31, R) >droVir3.scaffold_12822 3443156 92 - 4096053 AGUUGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAUU-GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUU-- ..(((((((((....))))))))).((((((.((((((.(((.(((.((...----..)).))-)...))))))))).)))))).............-- ( -29.40, z-score = -1.94, R) >droGri2.scaffold_15074 3134420 92 + 7742996 AGUUGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC----AUCUAUU-GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUU-- ..(((((((((....))))))))).((((((.((((((.(((.(((.((...----..)).))-)...))))))))).)))))).............-- ( -29.40, z-score = -1.94, R) >droMoj3.scaffold_6540 840804 93 - 34148556 AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCACC---AUCUAAC-GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUU-- ....(((((((....)))))))...((((((.((((((.(((......((...---.......-))..))))))))).)))))).............-- ( -26.70, z-score = -0.74, R) >droWil1.scaffold_181089 2773527 95 + 12369635 AGUUGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCACCAGCAC----AUCUAUUUGCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUUU ..(((((((((....))))))))).((((((.((((((.(((......(((.----.......)))..))))))))).))))))............... ( -29.60, z-score = -2.14, R) >anoGam1.chr2R 11616804 99 - 62725911 AGUUGACGGGACACAGCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCACCAUUACUUUUUUGCCCUAUUACGUGUCGUGACGUGUCGUUUCUCUUU ..((((((((......)))))))).(..((((((((((((((......(((............))).....)))))).))))))))..).......... ( -28.60, z-score = -1.64, R) >consensus AGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC____AUCUAU__GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUUU ....(((((((....)))))))...((((((.((((((.(((..........................))))))))).))))))............... (-24.94 = -25.02 + 0.08)


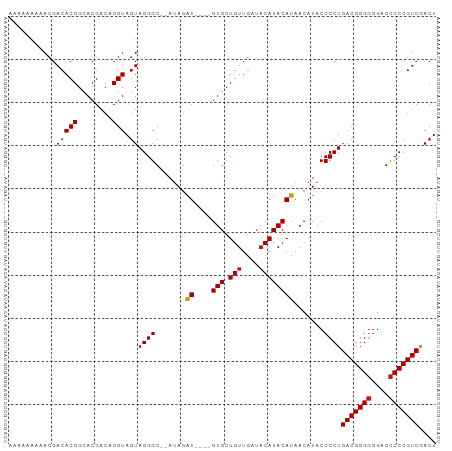
| Location | 18,299,445 – 18,299,538 |
|---|---|
| Length | 93 |
| Sequences | 13 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 94.53 |
| Shannon entropy | 0.11862 |
| G+C content | 0.50065 |
| Mean single sequence MFE | -27.94 |
| Consensus MFE | -22.19 |
| Energy contribution | -22.11 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.79 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.563242 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
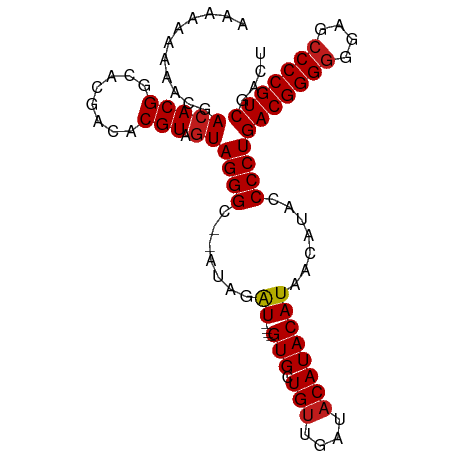
>dm3.chr3R 18299445 93 - 27905053 AAAAAAAAACGACACGGCACGACACGUAGUAGGGC--AUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU .........(((((((........)))....((((--....((----(((.(((....))))))))......(((((....))))).)))).))))... ( -28.60, z-score = -2.19, R) >droPer1.super_0 6666260 93 - 11822988 -AAAAAAAACGACACGGCACGACACGUAGUAGGGC-AAUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU -........(((((((........)))....((((-.....((----(((.(((....))))))))......(((((....))))).)))).))))... ( -28.60, z-score = -2.19, R) >dp4.chr2 17013568 93 + 30794189 -AAAAAAAACGACACGGCACGACACGUAGUAGGGC-AAUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU -........(((((((........)))....((((-.....((----(((.(((....))))))))......(((((....))))).)))).))))... ( -28.60, z-score = -2.19, R) >droAna3.scaffold_12911 5340336 93 - 5364042 AAAAAAAAACGACACGGCACGACACGUAGUAGGGC--UUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCAACU ..........((((((........)))....((((--((..((----(((.(((....))))))))......((((.....)))))))))).))).... ( -28.30, z-score = -2.19, R) >droEre2.scaffold_4820 691997 93 + 10470090 AAAAAAAAACGACACGGCACGACACGUAGUAGGGC--AUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU .........(((((((........)))....((((--....((----(((.(((....))))))))......(((((....))))).)))).))))... ( -28.60, z-score = -2.19, R) >droYak2.chr3R 19144887 93 - 28832112 AAAAAAAAACGACACGGCACGACACGUAGUAGGGC--AUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU .........(((((((........)))....((((--....((----(((.(((....))))))))......(((((....))))).)))).))))... ( -28.60, z-score = -2.19, R) >droSec1.super_0 18653857 93 - 21120651 AAAAAAAAACGACACGGCACGACACGUAGUAGGGC--AUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU .........(((((((........)))....((((--....((----(((.(((....))))))))......(((((....))))).)))).))))... ( -28.60, z-score = -2.19, R) >droSim1.chr3R 18107955 93 - 27517382 AAAAAAAAACGACACGGCACGACACGUAGUAGGGC--AUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU .........(((((((........)))....((((--....((----(((.(((....))))))))......(((((....))))).)))).))))... ( -28.60, z-score = -2.19, R) >droVir3.scaffold_12822 3443156 92 + 4096053 --AAAAAAACGACACGGCACGACACGUAGUAGGGC-AAUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCAACU --.........(((((........))).))((((.-.....((----(((.(((....)))))))).......))))(((((((....))))))).... ( -28.12, z-score = -2.14, R) >droGri2.scaffold_15074 3134420 92 - 7742996 --AAAAAAACGACACGGCACGACACGUAGUAGGGC-AAUAGAU----GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCAACU --.........(((((........))).))((((.-.....((----(((.(((....)))))))).......))))(((((((....))))))).... ( -28.12, z-score = -2.14, R) >droMoj3.scaffold_6540 840804 93 + 34148556 --AAAAAAACGACACGGCACGACACGUAGUAGGGC-GUUAGAU---GGUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU --.......(((((((........)))....((((-((((...---.(((.(((....)))))).))))...(((((....))))).)))).))))... ( -29.40, z-score = -1.48, R) >droWil1.scaffold_181089 2773527 95 - 12369635 AAAAAAAAACGACACGGCACGACACGUAGUAGGGCAAAUAGAU----GUGCUGGUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCAACU ...........(((((........))).))((((.......((----(((.((......))))))).......))))(((((((....))))))).... ( -28.14, z-score = -2.02, R) >anoGam1.chr2R 11616804 99 + 62725911 AAAGAGAAACGACACGUCACGACACGUAAUAGGGCAAAAAAGUAAUGGUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGCUGUGUCCCGUCAACU ..........(((((((......))))....((((((...((((....)))).)))...........(((((.(((....))).))))))))))).... ( -21.00, z-score = 0.19, R) >consensus AAAAAAAAACGACACGGCACGACACGUAGUAGGGC__AUAGAU____GUGCUGUUGAUACAUACAUAACAUACCCCUGACGGGGGGAGCCCCGUCGACU ............((((((((...........................)))))).))....................((((((((....))))))))... (-22.19 = -22.11 + -0.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:35:21 2011