| Sequence ID | dm3.chr2L |
|---|---|
| Location | 8,178,154 – 8,178,261 |
| Length | 107 |
| Max. P | 0.998248 |

| Location | 8,178,154 – 8,178,261 |
|---|---|
| Length | 107 |
| Sequences | 13 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 78.57 |
| Shannon entropy | 0.47111 |
| G+C content | 0.52976 |
| Mean single sequence MFE | -43.26 |
| Consensus MFE | -24.45 |
| Energy contribution | -25.14 |
| Covariance contribution | 0.69 |
| Combinations/Pair | 1.50 |
| Mean z-score | -2.99 |
| Structure conservation index | 0.57 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.30 |
| SVM RNA-class probability | 0.998248 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
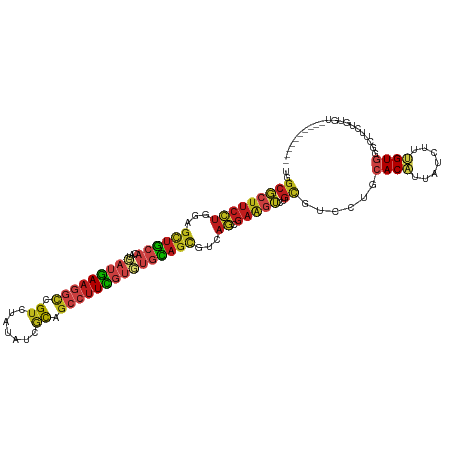
>dm3.chr2L 8178154 107 + 23011544 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCUGUGUAUAUCGCAGCCUUCGUGUGCAGUGUCAGCGAAGUCUGCGUACUGCACAUUAUCUUUGUGGGUUUCUGUGU---------- ..((((((((((..(((((((((...((((((((((.....)))))))))))))))))))..))).)))))..)).....(((((..((((....))))...)))))---------- ( -49.40, z-score = -4.58, R) >anoGam1.chr3R 22391166 107 - 53272125 UGGCUGUUCUUCGAAAUUCACUUGCUGAAGAUCGUGCUGAUCGUGGCGUUCUCGCUGAGCGUCAACUCGGUCAACUUCCUGCACCUGCUGUACGUGGUGCUCUCCGU---------- .(((((.((((((..(......)..)))))).)).)))(((((((((((((.....))))))))...)))))........(((((.((.....))))))).......---------- ( -32.10, z-score = -0.93, R) >droGri2.scaffold_15252 5074377 107 - 17193109 UGGCGCUUCCUCGAGCUGCACAUCAUGAAGGCCGUUUACAUCGCCGCCUUCGUCUGCAGCGUCAGCGAAGUUUGUGUCCUGCACAUUGUCUUUGUCGGCUUCUGUGU---------- ..((((........((((((....((((((((.((.......)).)))))))).))))))(((.((((((..((((.....))))....)))))).)))....))))---------- ( -37.70, z-score = -1.82, R) >droMoj3.scaffold_6500 30780113 107 - 32352404 UGGCGCUUCCUCGAGCUGCACAUCAUGAAGGCCGUCUACAUAGCCGCCUUCGUGUGCAGCGUGAGCGAGGUCUGUGUGCUGCACAUCAUCUUUGUGGGCUUCUGUGU---------- ..((((..(((((.(((((((((...((((((.((.......)).))))))))))))))).....)))))...))))(((.((((.......)))))))........---------- ( -44.10, z-score = -1.67, R) >droVir3.scaffold_12963 9686519 107 + 20206255 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCCGUUUAUAUAGCGGCCUUCGUGUGCAGCGUCAGCGAAGUAUGCGUCCUGCACAUUGUCUUUGUGGGCUUCUGUGU---------- .(((((((((((..(((((((((...((((((((((.....)))))))))))))))))))..))).)))....)))))..(((((..((((....))))...)))))---------- ( -53.60, z-score = -5.22, R) >droPer1.super_10 616029 107 + 3432795 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCCGUCUACAUCACCGCCUUUGUGUGCAGCGUGAGCGAGGUCUGCGUCCUGCACAUUGUCUUUGUGGGCUUCUGUGU---------- .(((((..(((.(.(((((((((...((((((.((.......)).))))))))))))))).....).)))...)))))..(((((..((((....))))...)))))---------- ( -43.50, z-score = -1.66, R) >dp4.chr4_group4 1612709 107 + 6586962 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCCGUCUAUAUCACCGCCUUUGUGUGCAGCGUGAGCGAGGUCUGCGUCCUGCACAUUGUCUUUGUGGGCUUCUGUGU---------- .(((((..(((.(.(((((((((...((((((.((.......)).))))))))))))))).....).)))...)))))..(((((..((((....))))...)))))---------- ( -43.50, z-score = -1.99, R) >droAna3.scaffold_12916 9523181 107 + 16180835 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCCGUCUACAUAGCCGCCUUUGUCUGCAGUGUCAGCGAGGUGUGUGUCCUGCACAUCGUCUUCGUGGGCUUCUGCGU---------- ..((((.(((((..((((((....(..(((((.((.......)).)))))..).))))))..))).))(((((((.....)))))))((((....))))....))))---------- ( -38.60, z-score = -0.26, R) >droEre2.scaffold_4929 17084165 107 + 26641161 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCUGUCUAUAUUGCAGCCUUCGUGUGCAGCGUCAGCGAAGUCUGCGUACUGCACAUUAUCUUUGUGGGUUUCUGUGU---------- ..((((((((((..(((((((((...(((((((((.......))))))))))))))))))..))).)))))..)).....(((((..((((....))))...)))))---------- ( -48.50, z-score = -4.33, R) >droYak2.chr2L 10810149 107 + 22324452 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCUGUGUAUAUCGCAGCCUUCGUGUGCAGUGUCAGCGAAGUCUGCGUACUGCACAUUAUCUUUGUGGGUUUCUGUGU---------- ..((((((((((..(((((((((...((((((((((.....)))))))))))))))))))..))).)))))..)).....(((((..((((....))))...)))))---------- ( -49.40, z-score = -4.58, R) >droSec1.super_3 3675212 107 + 7220098 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCUGUGUAUAUCGCAGCCUUCGUGUGCAGUGUCAGCGAAGUCUGCGUACUGCACAUUAUCUUUGUGGGUUUCUGUGU---------- ..((((((((((..(((((((((...((((((((((.....)))))))))))))))))))..))).)))))..)).....(((((..((((....))))...)))))---------- ( -49.40, z-score = -4.58, R) >droSim1.chr2L 7960835 107 + 22036055 UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCUGUGUAUAUCGCAGCCUUCGUGUGCAGUGUCAGUGAAGUCUGCGUACUGCACAUUAUCUUUGUGGGUUUCUGUGU---------- ..((((((((((..(((((((((...((((((((((.....)))))))))))))))))))..))).)))))..)).....(((((..((((....))))...)))))---------- ( -49.40, z-score = -4.89, R) >triCas2.ChLG5 7513326 117 - 18847211 UUCCUAUUCCUGGAGUUACACAUGCCCAAAUUCGUCGUUUUGUUUUUAAUGCUUGUUUGCAUUUACGACAAAUGCGCCUUGUACUUCAUCCUAGUCCUUCUCCUAGUACUUGCGUGU ((((.......))))....((((((........(.(((((((((...(((((......)))))...)))))).))).)..(((((...................)))))..)))))) ( -23.21, z-score = -2.38, R) >consensus UGGCGCUUCCUGGAGCUGCACAUCAUGAAGGCCGUCUAUAUCGCAGCCUUCGUGUGCAGCGUCAGCGAAGUCUGCGUCCUGCACAUUAUCUUUGUGGGCUUCUGUGU__________ ..(((((((((...((((((...(((((((((.((.......)).)))))))))))))))...)).)))))..))......((((.......))))..................... (-24.45 = -25.14 + 0.69)

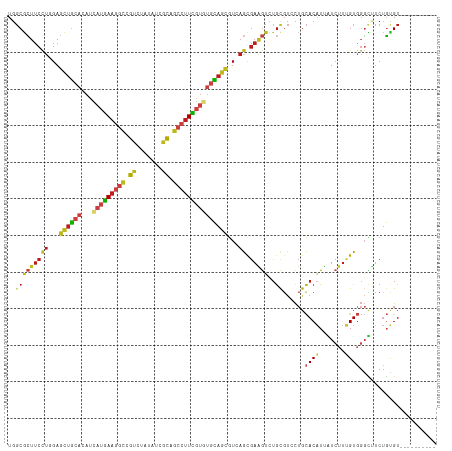
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:25:28 2011