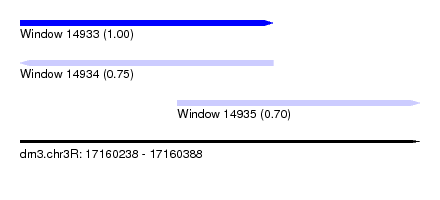
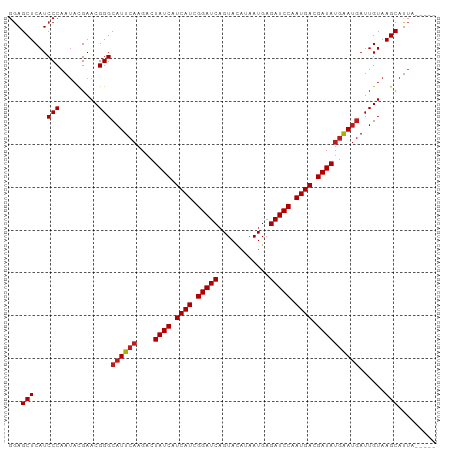
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 17,160,238 – 17,160,388 |
| Length | 150 |
| Max. P | 0.997903 |

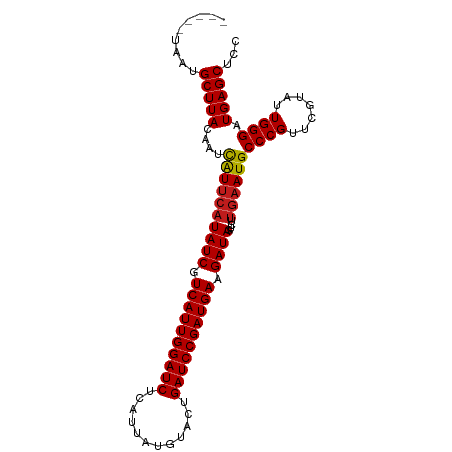
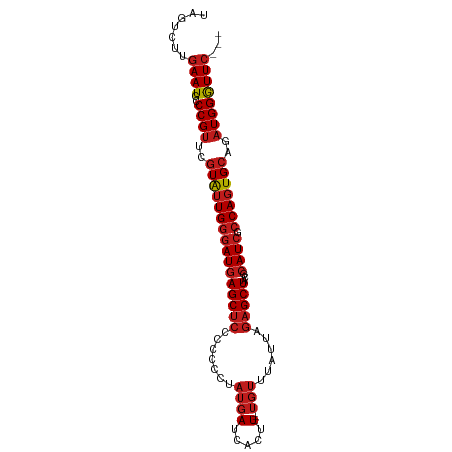
| Location | 17,160,238 – 17,160,333 |
|---|---|
| Length | 95 |
| Sequences | 13 |
| Columns | 100 |
| Reading direction | forward |
| Mean pairwise identity | 95.50 |
| Shannon entropy | 0.10704 |
| G+C content | 0.40095 |
| Mean single sequence MFE | -31.69 |
| Consensus MFE | -29.48 |
| Energy contribution | -29.57 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.07 |
| Mean z-score | -3.61 |
| Structure conservation index | 0.93 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.21 |
| SVM RNA-class probability | 0.997903 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 17160238 95 + 27905053 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))... ( -30.20, z-score = -3.20, R) >anoGam1.chr2L 35827589 97 - 48795086 UUUUCUGUUGCUUACAAUUGUCGCUAUCUUCAUUGGAUCGCAU---AUACGGAUCCGAUGACGAUAUUCUUGAAUGCCCGUUCAUGUUGGGAUGAGCUAA .........(((((..........((((.((((((((((.(..---....))))))))))).))))..........((((.......)))).)))))... ( -26.20, z-score = -1.68, R) >droGri2.scaffold_15074 2671045 95 + 7742996 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))... ( -34.10, z-score = -4.33, R) >droMoj3.scaffold_6540 351454 95 - 34148556 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))... ( -34.10, z-score = -4.33, R) >droVir3.scaffold_12822 2935471 95 - 4096053 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))... ( -34.10, z-score = -4.33, R) >droWil1.scaffold_181130 16154286 95 + 16660200 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))... ( -34.10, z-score = -4.33, R) >droPer1.super_19 1385422 95 + 1869541 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))... ( -34.10, z-score = -4.33, R) >dp4.chr2 5656885 95 + 30794189 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))... ( -34.10, z-score = -4.33, R) >droAna3.scaffold_13340 3812251 95 + 23697760 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))... ( -30.20, z-score = -3.20, R) >droEre2.scaffold_4770 13234240 95 - 17746568 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))... ( -30.20, z-score = -3.20, R) >droYak2.chr3R 5963948 95 + 28832112 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))... ( -30.20, z-score = -3.20, R) >droSec1.super_6 27801 95 - 4358794 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))... ( -30.20, z-score = -3.20, R) >droSim1.chr3R 23206378 95 - 27517382 -----UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC -----....(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))... ( -30.20, z-score = -3.20, R) >consensus _____UAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCC .........(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))... (-29.48 = -29.57 + 0.09)

| Location | 17,160,238 – 17,160,333 |
|---|---|
| Length | 95 |
| Sequences | 13 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 95.50 |
| Shannon entropy | 0.10704 |
| G+C content | 0.40095 |
| Mean single sequence MFE | -22.21 |
| Consensus MFE | -21.42 |
| Energy contribution | -21.65 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.22 |
| Structure conservation index | 0.96 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.752228 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 17160238 95 - 27905053 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.25, R) >anoGam1.chr2L 35827589 97 + 48795086 UUAGCUCAUCCCAACAUGAACGGGCAUUCAAGAAUAUCGUCAUCGGAUCCGUAU---AUGCGAUCCAAUGAAGAUAGCGACAAUUGUAAGCAACAGAAAA ...((((..............)))).(((.....((((.((((.(((((.((..---..))))))).)))).))))((.((....))..))....))).. ( -19.94, z-score = -0.95, R) >droGri2.scaffold_15074 2671045 95 - 7742996 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.24, R) >droMoj3.scaffold_6540 351454 95 + 34148556 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.24, R) >droVir3.scaffold_12822 2935471 95 + 4096053 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.24, R) >droWil1.scaffold_181130 16154286 95 - 16660200 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.24, R) >droPer1.super_19 1385422 95 - 1869541 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.24, R) >dp4.chr2 5656885 95 - 30794189 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.24, R) >droAna3.scaffold_13340 3812251 95 - 23697760 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.25, R) >droEre2.scaffold_4770 13234240 95 + 17746568 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.25, R) >droYak2.chr3R 5963948 95 - 28832112 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.25, R) >droSec1.super_6 27801 95 + 4358794 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.25, R) >droSim1.chr3R 23206378 95 + 27517382 GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA----- ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))....----- ( -22.40, z-score = -1.25, R) >consensus GGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUA_____ ...(((...(((.........)))((((((....((((.((((.(((((............))))).)))).))))))))))......)))......... (-21.42 = -21.65 + 0.24)

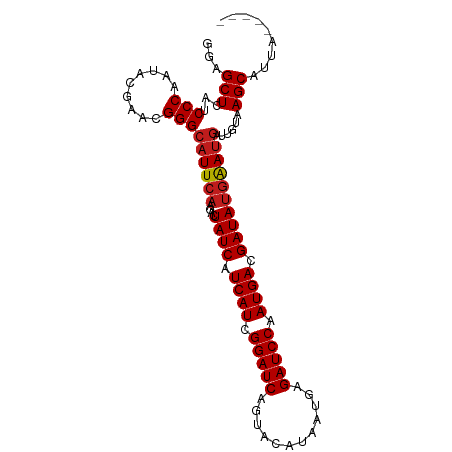
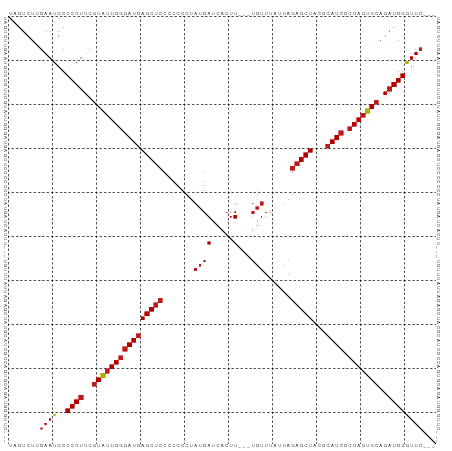
| Location | 17,160,297 – 17,160,388 |
|---|---|
| Length | 91 |
| Sequences | 13 |
| Columns | 97 |
| Reading direction | forward |
| Mean pairwise identity | 93.84 |
| Shannon entropy | 0.14006 |
| G+C content | 0.48583 |
| Mean single sequence MFE | -27.05 |
| Consensus MFE | -20.97 |
| Energy contribution | -21.51 |
| Covariance contribution | 0.54 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.78 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.697985 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 17160297 91 + 27905053 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC--- ...........((((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))))..--- ( -29.12, z-score = -2.54, R) >anoGam1.chr2L 35827650 85 - 48795086 UAUUCUUGAAUGCCCGUUCAUGUUGGGAUGAGCUAAGCCCC---GAUCACUU------UUUAAAACGCUAUGAAUAGGCAUUGCAGCUGGAUUC--- .(((((((((((((..(((((((((((...........)))---))).....------...........)))))..)))))).)))..))))..--- ( -18.69, z-score = -0.09, R) >droGri2.scaffold_15074 2671104 91 + 7742996 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUUAUAGAGCUACGCAUCGCCAGUGCAGAUGGAUUC--- .............((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))....--- ( -24.92, z-score = -1.65, R) >droMoj3.scaffold_6540 351513 94 - 34148556 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUUAUAGAGCUACGCAUCGCCAGUGCAGAUGGAUUGUUC .......((((..((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))...)))) ( -25.22, z-score = -1.57, R) >droVir3.scaffold_12822 2935530 91 - 4096053 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGAUUC--- .............((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))....--- ( -24.92, z-score = -1.65, R) >droWil1.scaffold_181130 16154345 94 + 16660200 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUUGUUUGUUUAAUAGAGCUACGCAUCGCCAGUGCAGAUGGAUUC--- .............((((..((((((((((((((((.......(..(........)..)......)))))...)))).)))))))..))))....--- ( -24.92, z-score = -1.47, R) >droPer1.super_19 1385481 91 + 1869541 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC--- ...........((((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))))..--- ( -29.12, z-score = -2.54, R) >dp4.chr2 5656944 91 + 30794189 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC--- ...........((((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))))..--- ( -29.12, z-score = -2.54, R) >droAna3.scaffold_13340 3812310 91 + 23697760 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC--- ...........((((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))))..--- ( -29.12, z-score = -2.54, R) >droEre2.scaffold_4770 13234299 91 - 17746568 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC--- ...........((((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))))..--- ( -29.12, z-score = -2.54, R) >droYak2.chr3R 5964007 91 + 28832112 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC--- ...........((((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))))..--- ( -29.12, z-score = -2.54, R) >droSec1.super_6 27860 91 - 4358794 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC--- ...........((((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))))..--- ( -29.12, z-score = -2.54, R) >droSim1.chr3R 23206437 91 - 27517382 UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU---UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC--- ...........((((((..((((((((((((((((.......(..(.....)---..)......)))))...)))).)))))))..))))))..--- ( -29.12, z-score = -2.54, R) >consensus UAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUU___UGUUUAUUAGAGCUACGCAUCGCCAGUGCAGAUGGGUUC___ .............((((..((((((((((((((((.............................)))))...)))).)))))))..))))....... (-20.97 = -21.51 + 0.54)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:32:01 2011