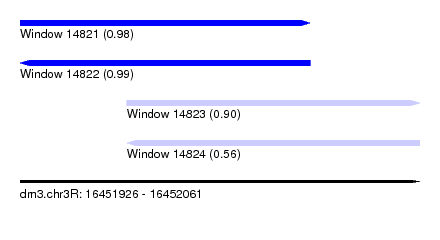
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 16,451,926 – 16,452,061 |
| Length | 135 |
| Max. P | 0.990889 |

| Location | 16,451,926 – 16,452,024 |
|---|---|
| Length | 98 |
| Sequences | 11 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 61.70 |
| Shannon entropy | 0.77893 |
| G+C content | 0.61039 |
| Mean single sequence MFE | -47.18 |
| Consensus MFE | -9.47 |
| Energy contribution | -9.54 |
| Covariance contribution | 0.07 |
| Combinations/Pair | 2.00 |
| Mean z-score | -2.87 |
| Structure conservation index | 0.20 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.01 |
| SVM RNA-class probability | 0.979102 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
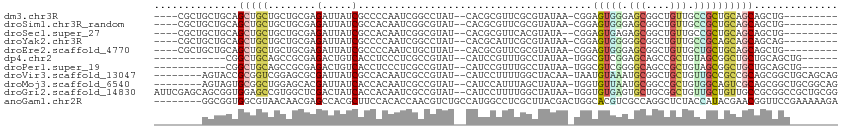
>dm3.chr3R 16451926 98 + 27905053 ----CGCUGCUGCAGCUGCUGCUGCGAGAUUAUCGCCCCAAUCGGCCUAU--CACGCGUUCGCGUAUAA-CGGAGUGGGAGCGGCUGUUGCCGCUGCAGCAGCUG--------- ----.(((((((((((....(((((((.....))))((((.((.(..(((--.((((....))))))).-).)).)))))))(((....))))))))))))))..--------- ( -51.40, z-score = -3.36, R) >droSim1.chr3R_random 820182 98 + 1307089 ----CGCUGCUGCAGCUGCUGCUGCGAGAUUAUCGCCACAAUCGGCGUAU--CACGCGUUCGCGUAUAA-CGGAGUGGGAGCGGCUGUUGCCGCUGCAGCAGCUG--------- ----.(((((((((((.(((.(..(..(((...((((......)))).))--)((((....))))....-....)..).)))(((....))))))))))))))..--------- ( -53.10, z-score = -3.58, R) >droSec1.super_27 514968 97 - 799419 ----CGCUGCUGCAGCUGCUGCUGCGAGAUUAUCGCCACAAUCGGCGUAU--CACGCGUUCACGUAUA--CGGAGUGAGAGCGGCUGUUGCCGCUGCAGCAGCUG--------- ----.(((((((((((.((.((.((..(((...((((......)))).))--).(((.(((((.....--....))))).))))).)).)).)))))))))))..--------- ( -50.40, z-score = -3.39, R) >droYak2.chr3R 5244665 98 + 28832112 ----CGCUGCUGCAGCUGCUGCUGCGAGAUUAUCGCCCCAAUCGGCCUAU--CACGCAUUCGCGUAUAA-CGGAGUGGGGGCGGCUGUUGCCGCAGCAGCAGCAG--------- ----.((....)).((((((((((((.(((..(((((((..((.(..(((--.((((....))))))).-).))...)))))))..)))..))))))))))))..--------- ( -53.40, z-score = -3.85, R) >droEre2.scaffold_4770 12530784 98 - 17746568 ----CGCUGCUGCAGCUGCUGCUGCGAGAUUAUCGCCCCAAUCUGCUUAU--CACGCGUUCGCGUAUAA-CGGAGUGGGAGCGGCUGUUGCUGCUGCAGCAGCUG--------- ----.(((((((((((.((((((((((.....))))((((.((((.((((--.((((....))))))))-)))).)))))))))).......)))))))))))..--------- ( -54.60, z-score = -4.86, R) >dp4.chr2 5786041 93 + 30794189 ------------CGGCUGCAGCCGCGAGACUGUCACCUCCCUCGCCGUAU--CAUCCGUUUGCCUAUAA-UGGCGUCGGAGCAGCCGCUGUAGCGGCUGCUGCAGCUG------ ------------((((((((((.(((((............))))).))..--..((((..((((.....-.)))).))))((((((((....))))))))))))))))------ ( -48.70, z-score = -4.47, R) >droPer1.super_19 1515937 93 + 1869541 ------------CGGCUGCAGCCGCGAGACUGUCACCUCCCUCGCCGUAU--CAUCCGUUUGCCUAUAA-UGGCGUCGGGGCAGCCGCUGUAGCGGCUGCUGCAGCUG------ ------------((((((((((.(((((............))))).))..--..((((..((((.....-.)))).))))((((((((....))))))))))))))))------ ( -47.60, z-score = -3.51, R) >droVir3.scaffold_13047 6940365 103 - 19223366 --------AGUACCGCGGUCGGAGCGCGAUUAUCGCCACAAUCGCCGUAU--CAUCCUUUUGGCUACAA-UAAUGUAAAUGCGGCUGCUGUUGCCGCCGCAGCGGCUGCAGCAG --------....((((((..((.((((((((........))))).))).)--)..)).......((((.-...))))...))))(((((((.(((((....))))).))))))) ( -39.70, z-score = -1.17, R) >droMoj3.scaffold_6540 29524188 103 + 34148556 --------AGUAGUGCGGCUGGAGCACGAUUAUCACCACAAUCGCCGUAU--CAUCCAUUUAGCUAUAA-UGGUGUUAAUGCGGCCGCUGUGGCAGUCGCAGCGGCUGCGGCAG --------....((((.......))))((((........))))(((.((.--((.(((((.......))-))))).))..((((((((((((.....))))))))))))))).. ( -41.50, z-score = -1.83, R) >droGri2.scaffold_14830 881039 111 + 6267026 AUUCGAGCAGCGGUGGAGCCGUGGCUCGACUAUCACCACAAUCGCCGUAU--CAUCCUUUUGGCUAUAA-UGGUGUGAGUGCUGCGGCUGUUGCUGUUGCCGCGGCCGCUGCGG ...(((((.((((.....)))).)))))(((..(((((..((.((((...--........)))).))..-)))))..)))((.((((((((.((....)).)))))))).)).. ( -49.90, z-score = -2.42, R) >anoGam1.chr2R 6040752 106 - 62725911 --------GGCGGUGGCGUAACAACGAGCCACGCUUCCACACCAACGUCUGCCAUGGCCUCGCUUACGACUGGCACGUCGCCAGGCUCUACCAUACGAACGGUUCCGAAAAAGA --------...((((((((((...(((((((.((....((......))..))..))).)))).))))).(((((.....)))))...))))).......((....))....... ( -28.70, z-score = 0.83, R) >consensus ________GCUGCAGCUGCUGCUGCGAGAUUAUCGCCACAAUCGCCGUAU__CAUGCGUUCGCCUAUAA_UGGAGUGGGAGCGGCUGCUGCCGCUGCAGCAGCUGC________ ..............(((((........(.....).......................................(((((.(((....))).)))))))))).............. ( -9.47 = -9.54 + 0.07)
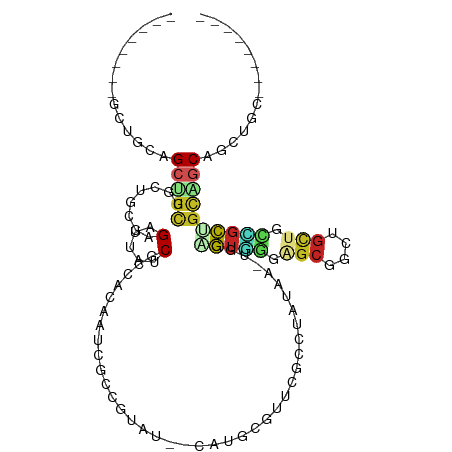
| Location | 16,451,926 – 16,452,024 |
|---|---|
| Length | 98 |
| Sequences | 11 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 61.70 |
| Shannon entropy | 0.77893 |
| G+C content | 0.61039 |
| Mean single sequence MFE | -45.27 |
| Consensus MFE | -9.81 |
| Energy contribution | -9.87 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.64 |
| Mean z-score | -2.97 |
| Structure conservation index | 0.22 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.44 |
| SVM RNA-class probability | 0.990889 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 16451926 98 - 27905053 ---------CAGCUGCUGCAGCGGCAACAGCCGCUCCCACUCCG-UUAUACGCGAACGCGUG--AUAGGCCGAUUGGGGCGAUAAUCUCGCAGCAGCAGCUGCAGCAGCG---- ---------..((((((((((((((....)))(((((((.((.(-((.(((((....)))))--...))).)).))))((((.....)))))))....))))))))))).---- ( -52.10, z-score = -4.53, R) >droSim1.chr3R_random 820182 98 - 1307089 ---------CAGCUGCUGCAGCGGCAACAGCCGCUCCCACUCCG-UUAUACGCGAACGCGUG--AUACGCCGAUUGUGGCGAUAAUCUCGCAGCAGCAGCUGCAGCAGCG---- ---------..((((((((((((((....)))(((.....((((-.....)).))..(((.(--((.(((((....)))))...))).))))))....))))))))))).---- ( -47.20, z-score = -3.54, R) >droSec1.super_27 514968 97 + 799419 ---------CAGCUGCUGCAGCGGCAACAGCCGCUCUCACUCCG--UAUACGUGAACGCGUG--AUACGCCGAUUGUGGCGAUAAUCUCGCAGCAGCAGCUGCAGCAGCG---- ---------..((((((((((((((....)))((((((((....--.....))))..(((.(--((.(((((....)))))...))).))).).))).))))))))))).---- ( -48.40, z-score = -3.74, R) >droYak2.chr3R 5244665 98 - 28832112 ---------CUGCUGCUGCUGCGGCAACAGCCGCCCCCACUCCG-UUAUACGCGAAUGCGUG--AUAGGCCGAUUGGGGCGAUAAUCUCGCAGCAGCAGCUGCAGCAGCG---- ---------(((((((.((((((((....)))((.((((.((.(-((.(((((....)))))--...))).)).))))((((.....)))).)).))))).)))))))..---- ( -48.50, z-score = -3.09, R) >droEre2.scaffold_4770 12530784 98 + 17746568 ---------CAGCUGCUGCAGCAGCAACAGCCGCUCCCACUCCG-UUAUACGCGAACGCGUG--AUAAGCAGAUUGGGGCGAUAAUCUCGCAGCAGCAGCUGCAGCAGCG---- ---------..(((((((((((.((....)).(((((((.((.(-((.(((((....)))))--...))).)).))))((((.....)))))))....))))))))))).---- ( -47.90, z-score = -4.14, R) >dp4.chr2 5786041 93 - 30794189 ------CAGCUGCAGCAGCCGCUACAGCGGCUGCUCCGACGCCA-UUAUAGGCAAACGGAUG--AUACGGCGAGGGAGGUGACAGUCUCGCGGCUGCAGCCG------------ ------..((((((((((((((....))))))))((((..(((.-.....)))...))))..--...(.((((((..(....)..)))))).).))))))..------------ ( -48.50, z-score = -4.11, R) >droPer1.super_19 1515937 93 - 1869541 ------CAGCUGCAGCAGCCGCUACAGCGGCUGCCCCGACGCCA-UUAUAGGCAAACGGAUG--AUACGGCGAGGGAGGUGACAGUCUCGCGGCUGCAGCCG------------ ------..((((((((((((((....))))))...(((..(((.-.....)))...)))...--.....((((((..(....)..)))))).))))))))..------------ ( -47.10, z-score = -3.57, R) >droVir3.scaffold_13047 6940365 103 + 19223366 CUGCUGCAGCCGCUGCGGCGGCAACAGCAGCCGCAUUUACAUUA-UUGUAGCCAAAAGGAUG--AUACGGCGAUUGUGGCGAUAAUCGCGCUCCGACCGCGGUACU-------- .(((((..(((((....)))))..)))))(((((..(((((...-.)))))......(((.(--.....((((((((....)))))))).))))....)))))...-------- ( -39.80, z-score = -1.17, R) >droMoj3.scaffold_6540 29524188 103 - 34148556 CUGCCGCAGCCGCUGCGACUGCCACAGCGGCCGCAUUAACACCA-UUAUAGCUAAAUGGAUG--AUACGGCGAUUGUGGUGAUAAUCGUGCUCCAGCCGCACUACU-------- ..(((((.(((((((.(.....).))))))).)).....(((((-((.......))))).))--....)))((((((....))))))((((.......))))....-------- ( -36.50, z-score = -1.77, R) >droGri2.scaffold_14830 881039 111 - 6267026 CCGCAGCGGCCGCGGCAACAGCAACAGCCGCAGCACUCACACCA-UUAUAGCCAAAAGGAUG--AUACGGCGAUUGUGGUGAUAGUCGAGCCACGGCUCCACCGCUGCUCGAAU ..((((((((.(((((..........))))).)).....(((((-(....(((.........--....)))....))))))...((.((((....)))).))))))))...... ( -43.92, z-score = -2.86, R) >anoGam1.chr2R 6040752 106 + 62725911 UCUUUUUCGGAACCGUUCGUAUGGUAGAGCCUGGCGACGUGCCAGUCGUAAGCGAGGCCAUGGCAGACGUUGGUGUGGAAGCGUGGCUCGUUGUUACGCCACCGCC-------- .......(((...(((..(((((((...((...(((((......)))))..))...))))).))..))).((((((((.((((.....)))).)))))))))))..-------- ( -38.00, z-score = -0.16, R) >consensus ________GCAGCUGCUGCAGCGGCAGCAGCCGCUCCCACUCCA_UUAUACGCGAACGCAUG__AUACGCCGAUUGUGGCGAUAAUCUCGCAGCAGCAGCUGCAGC________ ...........(((((.((.((.......)).))........................................((..(((.......)))..))))))).............. ( -9.81 = -9.87 + 0.06)
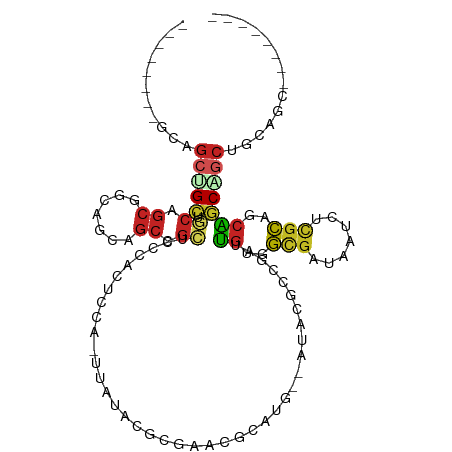
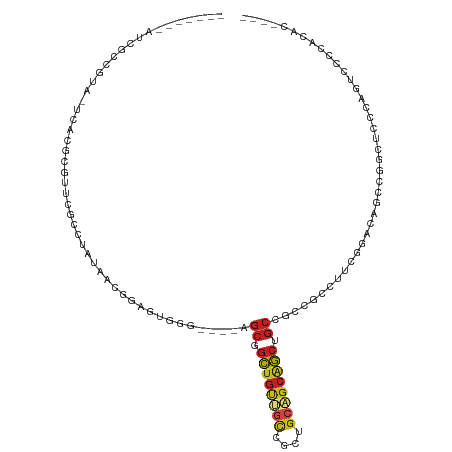
| Location | 16,451,962 – 16,452,061 |
|---|---|
| Length | 99 |
| Sequences | 12 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 62.86 |
| Shannon entropy | 0.76608 |
| G+C content | 0.62948 |
| Mean single sequence MFE | -41.30 |
| Consensus MFE | -12.86 |
| Energy contribution | -13.13 |
| Covariance contribution | 0.27 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.31 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.895821 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
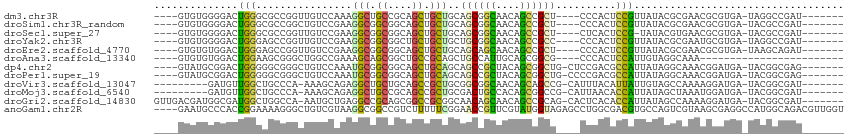
>dm3.chr3R 16451962 99 + 27905053 -------AUCGGCCUA-UCACGCGUUCGCGUAUAACGGAGUGGG----AGCGGCUGUUGCCGCUGCAGCAGCUGCGGCAGCCUUUGGACAACCGGCGCCCAGUCCCCACAC---- -------...((....-..((((....)))).....(((.((((----.(((((((((((.((((...)))).)))))))))..(((....))))).)))).)))))....---- ( -45.20, z-score = -1.75, R) >droSim1.chr3R_random 820218 99 + 1307089 -------AUCGGCGUA-UCACGCGUUCGCGUAUAACGGAGUGGG----AGCGGCUGUUGCCGCUGCAGCAGCUGCCGCCGCCUUCGGACAGCCGGCGCCCAGUCCCCACAC---- -------...((.((.-..((((....))))...))(((.((((----.(((((((((((....)))))))))))(((((.((......)).))))))))).)))))....---- ( -47.10, z-score = -1.79, R) >droSec1.super_27 515004 98 - 799419 -------AUCGGCGUA-UCACGCGUUCACGUAUA-CGGAGUGAG----AGCGGCUGUUGCCGCUGCAGCAGCUGCCGCCGCCUUCGGACAACCGGCGCCCAGUCCCCACAC---- -------...((.(((-(.(((......))))))-)(((.((..----.(((((((((((....)))))))))))(((((.(....).....)))))..)).)))))....---- ( -38.60, z-score = -0.81, R) >droYak2.chr3R 5244701 99 + 28832112 -------AUCGGCCUA-UCACGCAUUCGCGUAUAACGGAGUGGG----GGCGGCUGUUGCCGCAGCAGCAGCAGCCGCCGCCUUCGGACAACCGGCUCCCAGUCCCCACAC---- -------...((....-..((((....)))).....(((.((((----((((((((((((.......))))))))))))(((...((....))))).)))).)))))....---- ( -47.70, z-score = -3.41, R) >droEre2.scaffold_4770 12530820 99 - 17746568 -------AUCUGCUUA-UCACGCGUUCGCGUAUAACGGAGUGGG----AGCGGCUGUUGCUGCUGCAGCAGCUGCCGCCGCCUUCGGACAACCGGCUCCCAGUCCACACAC---- -------......(((-(.((((....)))))))).(((.((((----((((((((((((....))))))))))).((((.(....).....))))))))).)))......---- ( -46.60, z-score = -3.43, R) >droAna3.scaffold_13340 10566200 83 - 23697760 ------------------------UUUGCCUACAAUGGAGUGGG----CGCCGCUGCAAUGGCAGCUGCGGCAGCCGCUGCUUUCGGCCAGCCCGCUUCCAGUCCACACAC---- ------------------------...........(((((((((----(((((((((....))))).(((((....)))))....)))..)))))).))))..........---- ( -37.60, z-score = -1.71, R) >dp4.chr2 5786069 102 + 30794189 -------CUCGCCGUA-UCAUCCGUUUGCCUAUAAUGGCGUCGGAG-CAGCCGCUGUAGCGGCUGCUGCAGCUGCCGCCGCAUUUGGACAGCCCGCCCCCAGUCCGCAUAC---- -------...((....-...((((..((((......)))).))))(-(((((((....)))))))).((.(((((((.......))).))))..)).........))....---- ( -39.20, z-score = -1.18, R) >droPer1.super_19 1515965 102 + 1869541 -------CUCGCCGUA-UCAUCCGUUUGCCUAUAAUGGCGUCGGGG-CAGCCGCUGUAGCGGCUGCUGCAGCUGCCGCCGCAUUUGGACAGCCCGCCCCCAGUCCGCAUAC---- -------...((.((.-....(((..((((......)))).)))((-(.((.((((((((....)))))))).)).)))))....((((............))))))....---- ( -39.60, z-score = -0.43, R) >droVir3.scaffold_13047 6940397 96 - 19223366 -------AUCGCCGUA-UCAUCCUUUUGGCUACAAUAAUGUAAAUG-CGGCUGCUGUUGCCGCCGCAGCGGCUGCAGCAGCCUCUGCUUU-UGGGCAGCCAACAUC--------- -------...((((..-.........)))).......((((.....-.(((((((((.(((((....))))).))))))))).(((((..-..)))))...)))).--------- ( -39.80, z-score = -2.69, R) >droMoj3.scaffold_6540 29524220 96 + 34148556 -------AUCGCCGUA-UCAUCCAUUUAGCUAUAAUGGUGUUAAUG-CGGCCGCUGUGGCAGUCGCAGCGGCUGCGGCAGCCUCUGCUUU-UGGGCAGCCAACAUC--------- -------.........-....(((((.......)))))((((...(-(((((((((((.....))))))))))))(((.((((.......-.)))).)))))))..--------- ( -40.00, z-score = -2.12, R) >droGri2.scaffold_14830 881079 105 + 6267026 -------AUCGCCGUA-UCAUCCUUUUGGCUAUAAUGGUGUGAGUG-CUGCGGCUGUUGCUGUUGCCGCGGCCGCUGCGGCCUCAGCAUU-UGGCCAGCCAUCGCCAUCGUCAAC -------...((((..-.........))))....((((((((.(((-(.((((((((.((....)).)))))))).))((((........-.)))).)))).))))))....... ( -42.30, z-score = -1.48, R) >anoGam1.chr2R 6040784 111 - 62725911 ACCAACGUCUGCCAUGGCCUCGCUUACGACUGGCACGUCGCCAGGCUCUACCAUACGAACGGUUCCGAAAAAGACGGCCGCCUUACGACAGCCCUUUUCCGGUGGGCAUUC---- ......(((..((...((((((....)))..)))..((((..((((...(((........))).(((.......)))..))))..))))...........))..)))....---- ( -31.90, z-score = 0.16, R) >consensus _______AUCGCCGUA_UCACGCGUUCGCCUAUAACGGAGUGGG____AGCGGCUGUUGCCGCUGCAGCAGCUGCCGCCGCCUUCGGACAGCCGGCUCCCAGUCCCCACAC____ .................................................((.((((((((....)))))))).))........................................ (-12.86 = -13.13 + 0.27)
| Location | 16,451,962 – 16,452,061 |
|---|---|
| Length | 99 |
| Sequences | 12 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 62.86 |
| Shannon entropy | 0.76608 |
| G+C content | 0.62948 |
| Mean single sequence MFE | -42.87 |
| Consensus MFE | -12.66 |
| Energy contribution | -11.94 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 2.14 |
| Mean z-score | -1.31 |
| Structure conservation index | 0.30 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.558336 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 16451962 99 - 27905053 ----GUGUGGGGACUGGGCGCCGGUUGUCCAAAGGCUGCCGCAGCUGCUGCAGCGGCAACAGCCGCU----CCCACUCCGUUAUACGCGAACGCGUGA-UAGGCCGAU------- ----...((((((.((((.((.((((((........((((((.((....)).)))))))))))))).----)))).)))....(((((....))))).-....)))..------- ( -42.80, z-score = -0.43, R) >droSim1.chr3R_random 820218 99 - 1307089 ----GUGUGGGGACUGGGCGCCGGCUGUCCGAAGGCGGCGGCAGCUGCUGCAGCGGCAACAGCCGCU----CCCACUCCGUUAUACGCGAACGCGUGA-UACGCCGAU------- ----(((((.(((.((((.((.(((((((((...(((((((...)))))))..)))..)))))))).----)))).)))....(((((....))))).-)))))....------- ( -49.80, z-score = -1.72, R) >droSec1.super_27 515004 98 + 799419 ----GUGUGGGGACUGGGCGCCGGUUGUCCGAAGGCGGCGGCAGCUGCUGCAGCGGCAACAGCCGCU----CUCACUCCG-UAUACGUGAACGCGUGA-UACGCCGAU------- ----(((((.(((.((((.((.(((((((((...(((((((...)))))))..)))..)))))))).----)))).))).-)))))......(((...-..)))....------- ( -44.30, z-score = -0.75, R) >droYak2.chr3R 5244701 99 - 28832112 ----GUGUGGGGACUGGGAGCCGGUUGUCCGAAGGCGGCGGCUGCUGCUGCUGCGGCAACAGCCGCC----CCCACUCCGUUAUACGCGAAUGCGUGA-UAGGCCGAU------- ----...((((((.((((.(((((....))...)))((((((((.(((((...))))).))))))))----)))).)))....(((((....))))).-....)))..------- ( -46.30, z-score = -1.24, R) >droEre2.scaffold_4770 12530820 99 + 17746568 ----GUGUGUGGACUGGGAGCCGGUUGUCCGAAGGCGGCGGCAGCUGCUGCAGCAGCAACAGCCGCU----CCCACUCCGUUAUACGCGAACGCGUGA-UAAGCAGAU------- ----((.((((((.(((((((.((((((..(...((.(((((....))))).))..).)))))))))----)))).)))....(((((....))))).-...))).))------- ( -46.30, z-score = -2.45, R) >droAna3.scaffold_13340 10566200 83 + 23697760 ----GUGUGUGGACUGGAAGCGGGCUGGCCGAAAGCAGCGGCUGCCGCAGCUGCCAUUGCAGCGGCG----CCCACUCCAUUGUAGGCAAA------------------------ ----.(((.((.(.((((.(.((((.(((((.......)))))(((((.((.......)).))))))----))).))))).).)).)))..------------------------ ( -38.20, z-score = -0.57, R) >dp4.chr2 5786069 102 - 30794189 ----GUAUGCGGACUGGGGGCGGGCUGUCCAAAUGCGGCGGCAGCUGCAGCAGCCGCUACAGCGGCUG-CUCCGACGCCAUUAUAGGCAAACGGAUGA-UACGGCGAG------- ----.....((((...(.(((..(((((((....).)))))).))).).((((((((....)))))))-))))).(((((((((.(.....)..))))-)..))))..------- ( -44.40, z-score = -1.31, R) >droPer1.super_19 1515965 102 - 1869541 ----GUAUGCGGACUGGGGGCGGGCUGUCCAAAUGCGGCGGCAGCUGCAGCAGCCGCUACAGCGGCUG-CCCCGACGCCAUUAUAGGCAAACGGAUGA-UACGGCGAG------- ----((((.((..(((((((((((((((((....).))))))..)))).((((((((....)))))))-))))...(((......)))...))).)))-)))......------- ( -44.90, z-score = -1.01, R) >droVir3.scaffold_13047 6940397 96 + 19223366 ---------GAUGUUGGCUGCCCA-AAAGCAGAGGCUGCUGCAGCCGCUGCGGCGGCAACAGCAGCCG-CAUUUACAUUAUUGUAGCCAAAAGGAUGA-UACGGCGAU------- ---------(((((...((((...-...)))).((((((((..(((((....)))))..)))))))))-))))((((....))))(((..........-...)))...------- ( -39.32, z-score = -2.07, R) >droMoj3.scaffold_6540 29524220 96 - 34148556 ---------GAUGUUGGCUGCCCA-AAAGCAGAGGCUGCCGCAGCCGCUGCGACUGCCACAGCGGCCG-CAUUAACACCAUUAUAGCUAAAUGGAUGA-UACGGCGAU------- ---------...(((..((((...-...)))).)))((((((.(((((((.(.....).))))))).)-).....(((((((.......))))).)).-...))))..------- ( -34.20, z-score = -1.32, R) >droGri2.scaffold_14830 881079 105 - 6267026 GUUGACGAUGGCGAUGGCUGGCCA-AAUGCUGAGGCCGCAGCGGCCGCGGCAACAGCAACAGCCGCAG-CACUCACACCAUUAUAGCCAAAAGGAUGA-UACGGCGAU------- ((((..((..((..((((((....-..(((((..(((((.......)))))..))))).))))))..)-)..)).......(((..((....))...)-))))))...------- ( -35.20, z-score = -0.21, R) >anoGam1.chr2R 6040784 111 + 62725911 ----GAAUGCCCACCGGAAAAGGGCUGUCGUAAGGCGGCCGUCUUUUUCGGAACCGUUCGUAUGGUAGAGCCUGGCGACGUGCCAGUCGUAAGCGAGGCCAUGGCAGACGUUGGU ----......(((((((((((((((((((....)))))))..)))))))))...(((..(((((((...((...(((((......)))))..))...))))).))..))).))). ( -48.70, z-score = -2.65, R) >consensus ____GUGUGGGGACUGGGAGCCGGCUGUCCAAAGGCGGCGGCAGCUGCUGCAGCGGCAACAGCCGCU____CCCACUCCAUUAUAGGCAAACGCGUGA_UACGGCGAU_______ ..............(((................(((.((....)).)))..((((((....))))))..........)))................................... (-12.66 = -11.94 + -0.72)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:30:27 2011