| Sequence ID | dm3.chr3R |
|---|---|
| Location | 16,400,694 – 16,400,828 |
| Length | 134 |
| Max. P | 0.996815 |

| Location | 16,400,694 – 16,400,828 |
|---|---|
| Length | 134 |
| Sequences | 11 |
| Columns | 137 |
| Reading direction | forward |
| Mean pairwise identity | 74.01 |
| Shannon entropy | 0.55614 |
| G+C content | 0.44624 |
| Mean single sequence MFE | -33.96 |
| Consensus MFE | -14.47 |
| Energy contribution | -14.51 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.92 |
| Structure conservation index | 0.43 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.99 |
| SVM RNA-class probability | 0.996815 |
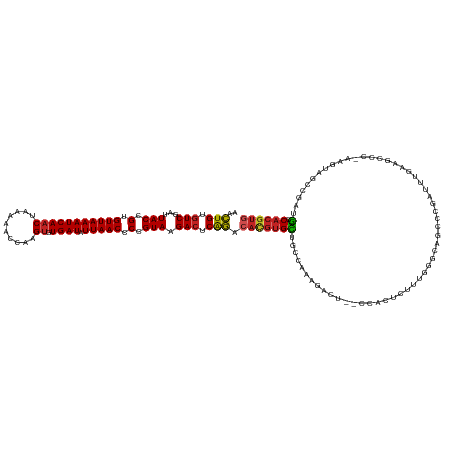
| Prediction | RNA |
Download alignment: ClustalW | MAF
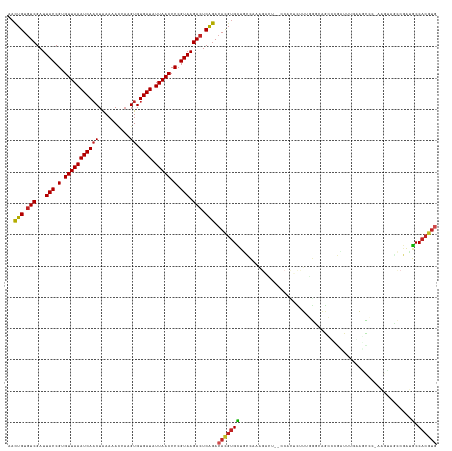
>dm3.chr3R 16400694 134 + 27905053 AACUGUGUCGAUUACCGUGUUAAAUCAUCUAAAAACCA-GUGUGAUAUUAACCCCGUAAGACUCAGACAUGUGCUGCCAAAGACU--ACACUUUUUGGCCAGCUCGCUUUGAAGCCCCAAGUAGCCGAUGCCACGUG ..(((.(((...(((.(.((((((((((((.......)-).))))).))))).).))).))).))).((((((..((((((((..--....))))))))..((((((((((......)))..)).))).)))))))) ( -33.00, z-score = -2.25, R) >droSim1.chr3R 22449020 134 - 27517382 AACUGUGUCGAUUACCGUGUUAAAUCAACUAAAAACCA-GUGUGAUAUUAACCCCGUAAGACUCAGACACGUGCUGCCAAAGACU--CCGCUCUUUGGCCAGCUCGCUUUGGAGCCCCAAGUAGCCGAUGCCACGUG ..(((.(((...(((.(.((((((((((((.......)-)).)))).))))).).))).))).))).((((((..((((((((..--....))))))))..(((((((((((....))))..)).))).)))))))) ( -40.70, z-score = -3.71, R) >droSec1.super_27 463620 134 - 799419 AACUGUGUCGAUUACCGUGUUAAAUCAACUAAAAACCA-GUGUGAUAUUAACCCCGUAAGACUCAGACACGUGCUGCCAAAGACU--ACGCUCUUUGGCCAGCUCGCUUUGGAGCCCCAAGUAGCCGAUGCCACGUG ..(((.(((...(((.(.((((((((((((.......)-)).)))).))))).).))).))).))).((((((..((((((((..--....))))))))..(((((((((((....))))..)).))).)))))))) ( -40.70, z-score = -3.60, R) >droYak2.chr3R 5186418 134 + 28832112 AACUGUGUCGAUUACCGUGUUAAAUCAACUAAAAACCA-GUGUGAUAUUAACCCCGUAAGACUCAGACAUGUGCUGCCAAAGACU--CCACUCUUUGGGCAGCUCGUUUUAAAGCCGAAAGUAGCCGAUGCCACGUG ..(((.(((...(((.(.((((((((((((.......)-)).)))).))))).).))).))).))).((((((((((((((((..--....))))).))))))..........(((....)..)).......))))) ( -34.40, z-score = -2.11, R) >droEre2.scaffold_4770 12474356 134 - 17746568 AACUGUGUCGAUUACCGUGUUAAAUCAACUAAAAACCA-GUGUGAUAUUAACCCCGUAAGACUCAGACACGUGCUGCCAAAGACU--CCACUUUUUGGCCAGCUCGCUUUGAACCCAAAAGAAGCCGAUGCCACGUG ..(((.(((...(((.(.((((((((((((.......)-)).)))).))))).).))).))).))).((((((..((((((((..--....))))))))..(((((((((..........)))).))).)))))))) ( -36.20, z-score = -3.68, R) >droAna3.scaffold_13340 10521363 131 - 23697760 AACUGUUUCGAUUACCGUGUUAAAUCAACUAACAA-CAAGUGUGAUUUUAACCCCGUAAGACUCAGACACGUGUUGCCAAAGAUGA-AAAUAUCUUGGUCAACCACACAACUACU----AUACCCCAAUGCCACGUG ..(((..((...(((.(.((((((((((((.....-..))).))).)))))).).))).))..))).((((((..((..((((((.-...))))))((....))...........----..........)))))))) ( -23.50, z-score = -0.73, R) >dp4.chr2 28181924 137 - 30794189 AACUGUGUCGAUUACCGUGUUAAAUCAACUGAAUAUCAAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGCUACUAACGGAUUCCCAUUCCGUUAGUCGCCCGAUUCUAUAUGCAAUCUGUUCGAUGCCACGUG ..(((.(((...(((.(.((((((((((((........))).)))).))))).).))).))).))).(((((((.(((((((((.......))))))))).))............(((.((.....))))).))))) ( -38.40, z-score = -3.91, R) >droPer1.super_7 1662108 137 - 4445127 AACUGUGUCGAUUACCGUGUUAAAUCAACUGAAUAUCAAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGCUACUAACGGAUUCCCAUUCCGUUAGUCGCCCGAUUCUAUAUGCAAUCUCUUCGAUGCCACGUG ..(((.(((...(((.(.((((((((((((........))).)))).))))).).))).))).))).(((((((.(((((((((.......))))))))).))............(((.((.....))))).))))) ( -38.40, z-score = -4.22, R) >droVir3.scaffold_13047 8808111 116 - 19223366 AACUGUGUCGAUUACCGUGUUAAAUCAACUAAAUAUCAAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGUUUGC--------------U-UUUAGCAGACGCAUUUCGAUU----UGGCC--AUUGCCACGUG ..(((.(((...(((.(.((((((((((((........))).)))).))))).).))).))).))).(((((((((((--------------.-....)))))))).........----((((.--...)))).))) ( -33.30, z-score = -3.30, R) >droMoj3.scaffold_6540 5598709 113 - 34148556 AAUUGUGUCGAUUACCGUGUUAAAUCAACUGAAAAUCAAGUGUGAUCUUAACCCCGUAAGACUCGAACACGUGUUUGC--------------U-UU-AGCUGGCAAAUA--GAUU----AGUUU--UAUGCCACGUU ....(((((((((((.(.((((((((((((........))).)))).))))).).))))...))).)))).(((((((--------------(-..-....))))))))--....----.....--........... ( -23.80, z-score = -1.02, R) >droGri2.scaffold_14624 4094245 112 - 4233967 ACCUGUGUCGAUUACCGUGUUAAAUCAACUAAAAAUCCAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGUUUGC--------------CAUUUGGCAAACGCAUUAGAAUC----UGGCC--UGAACC----- ....(((((..((((.(.((((((((((((........))).)))).))))).).))))......)))))((((((((--------------(....))))))))).((((....----....)--)))...----- ( -31.20, z-score = -3.55, R) >consensus AACUGUGUCGAUUACCGUGUUAAAUCAACUAAAAACCAAGUGUGAUAUUAACCCCGUAAGACUCAGACACGUGCUGCCAAAGACU__CCACUCUUUGGGCAGCCCGAUUUGAAGCCC_AAGUAGCCGAUGCCACGUG ..(((.(((...(((.(.(((((((((((..........)).)))).))))).).))).))).))).(((((((.......................................................).)))))) (-14.47 = -14.51 + 0.04)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:30:18 2011