
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 16,350,236 – 16,350,355 |
| Length | 119 |
| Max. P | 0.870208 |

| Location | 16,350,236 – 16,350,355 |
|---|---|
| Length | 119 |
| Sequences | 12 |
| Columns | 139 |
| Reading direction | forward |
| Mean pairwise identity | 74.38 |
| Shannon entropy | 0.51659 |
| G+C content | 0.31138 |
| Mean single sequence MFE | -27.06 |
| Consensus MFE | -7.71 |
| Energy contribution | -7.64 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.84 |
| Structure conservation index | 0.28 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.870208 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 16350236 119 + 27905053 CCUAAUUAAUUGCCAUUUGCGG---UCUCCCCUAAUUGCAAUUAGUU----ACCUUUAAUUAUAAUUAGUUUGAUUUCACUAUUUAGAUUAUUCAGCUGCGGUU-----ACCGCAAUGGUUAACUAAUUAA-------- ..((((((...((((((.((((---(..((.(((((((.((((((..----....)))))).))))))).((((..((........))....))))....))..-----)))))))))))....)))))).-------- ( -28.60, z-score = -3.72, R) >droSim1.chr3R 22398062 119 - 27517382 CCUAAUUAAUUGCCAUUUGCGG---UCGCCCCUAAUUGCAAUUAGUU----ACCUUUAAUUAUAAUUAGUUUGAUUUCACUAUUUAGAUUAUUCAGCUGCGGUC-----ACCGCAAUGGUUAACUAAUUAA-------- ..((((((...((((((.((((---(.(((.(((((((.((((((..----....)))))).))))))).((((..((........))....))))....))).-----)))))))))))....)))))).-------- ( -30.20, z-score = -3.90, R) >droSec1.super_27 413865 119 - 799419 CCUAAUUAAUUGCCAUUUGCGG---UCGCUCCUAAUUGCAAUUAGUU----ACCUUUAAUUAUAAUUAGUUUGAUUUCACUAUUUAGAUUAUUCAGCUGCGGUC-----ACCGCAAUGGUUAACUAAUUAA-------- ..((((((...((((((.((((---((((..(((((((.((((((..----....)))))).))))))).((((..((........))....))))..)))...-----)))))))))))....)))))).-------- ( -28.30, z-score = -3.30, R) >droYak2.chr3R 5132766 118 + 28832112 CCUAAUUAAUUGCCAUUUGCGG---UCGCCUCUAAUUGCAAUUAGUU----ACCUUUAAUUAUAAUUAGUUUGAUU-CACUAUUUAGAUUAUUCAGCUGCGGUU-----ACCGCAACGGUUAACUAAUUAA-------- ..((((((...(((..((((((---(.(((.(((((((.((((((..----....)))))).))))))).((((.(-(........))....))))....))).-----))))))).)))....)))))).-------- ( -27.70, z-score = -3.00, R) >droEre2.scaffold_4770 12422470 119 - 17746568 CCUAAUUAAUUGCCAUUUGCGG---UCGCCUCUAAUUGCAAUUAGUU----ACCUUUAAUUAUAAUUAGUUUGAUUUCACUAUUUAGAUUAUUCAGCUGUGGUU-----ACCGCAAUGGUUAACUAAUUAA-------- ..((((((...((((((.((((---(.(((.(((((((.((((((..----....)))))).))))))).((((..((........))....))))....))).-----)))))))))))....)))))).-------- ( -30.10, z-score = -3.89, R) >droAna3.scaffold_12911 2656290 123 - 5364042 CCAAAUUAAUUGCCAUUUGCGGCCCUCCGGCUUGAAUGCAAUUAGUUU---ACCUUUAAUUAUAAUUAGUUUGAUUUCACUAUUUAGAUAAUUUAACUGCGGUU-----ACCGCAAUGAUUAACUAAUUAA-------- ..(((((((((((.(((((.((((....))))))))))))))))))))---...........(((((((((.....(((((....))..........(((((..-----.))))).)))..))))))))).-------- ( -28.40, z-score = -3.20, R) >dp4.chr2 15503859 119 + 30794189 CCUAAUUAAUUGCCAUUUGCGG---UCGGCUUUGAAUGCAAUUAGUU----ACCUUUAAUUGUAAUUAGUUUGCUUUCACUAUUUAGAUUAUUUAGCUGCGGUA-----ACCGCAAUGAUUUACUAAUUAA-------- ..((((((........((((((---((((((.((((.((((((((((----((........)))))))).)))).))))((....)).......))))).....-----)))))))........)))))).-------- ( -26.79, z-score = -2.03, R) >droPer1.super_0 6913861 119 - 11822988 CCUAAUUAAUUGCCAUUUGCGG---UCGGCUUUGAAUGCAAUUAGUU----ACCUUUAAUUGUAAUUAGUUUGCUUUCACUAUUUAGAUUAUUUAGAUGCGGUA-----ACCGCAAUGAUUUACUAAUUAA-------- ..((((((........((((((---((.((..((((.((((((((((----((........)))))))).)))).))))..(((((((...))))))))).)..-----)))))))........)))))).-------- ( -26.99, z-score = -2.34, R) >droWil1.scaffold_181108 2151176 128 - 4707319 CUUAAUUAAUUGCCAUUUGCGG--UUUGGUUUUCAAUGCAAUUAGCUGCUUACCUUUAAUUAUAAUUAGUCUGAUGUCAUUAGAUAGACUAUUGAAAUGUGGUUUUU-UGCUAUAAUCAUUAACUAAUUAA-------- ..(((((((.........((((--((..((.......))....))))))......)))))))((((((((.(((((....(((..(((((((......)))))))..-..)))....))))))))))))).-------- ( -25.36, z-score = -1.41, R) >droVir3.scaffold_13047 8746629 113 - 19223366 UCUAAUUAAUUGGCAUUUACGU---UUUGGCUUUAAUACAAUUAUUU----GACCAUAAUUAUAAUUAGUGGCAUUUCAC--ACUUGCUUAUUUAAUUGUGGUUU---ACCUAA--CUAGCUAUUAA------------ ..........((((........---.(((.........)))......----(((((((((((.....((((........)--)))........))))))))))).---......--...))))....------------ ( -17.62, z-score = -0.63, R) >droMoj3.scaffold_6540 5541833 129 - 34148556 UCUAAUUAAUUGGCAUUUACGU---UCCGCCAUUAAUACAAUUAUUG----CACCACAAUUAUAAUUAGUGGCGUUUCAC--GUUUGCUUAUUUAAUUGUGGUUUCCAUGUCCCCGCAAUCGAUUAACUAGUCAUUAA- .(((.((((((((((...((((---..((((((((((......((((----.....))))....))))))))))....))--)).))).......(((((((...........)))))))))))))).))).......- ( -30.70, z-score = -3.73, R) >droGri2.scaffold_14624 4038741 130 - 4233967 UCUAAUUAAUUGGCAUUUUCGG---UUUAGCUUUAACACAAUUAGUU----GACCGCAAUUAUAAUUAGUGGUAUUUCAC--ACUUACAUAUUUACUUGUGGUCUCUAAUUCAAUGCAAUUCAUUAACUACACAAUUAU ..((((.(((((.((((....(---((.......)))..((((((..----((((((((.((.....((((........)--)))........)).)))))))).)))))).))))))))).))))............. ( -24.02, z-score = -2.93, R) >consensus CCUAAUUAAUUGCCAUUUGCGG___UCGGCCUUAAAUGCAAUUAGUU____ACCUUUAAUUAUAAUUAGUUUGAUUUCACUAUUUAGAUUAUUUAGCUGCGGUU_____ACCGCAAUGAUUAACUAAUUAA________ ...(((((((((..........................)))))))))...................................................(((((......)))))......................... ( -7.71 = -7.64 + -0.06)

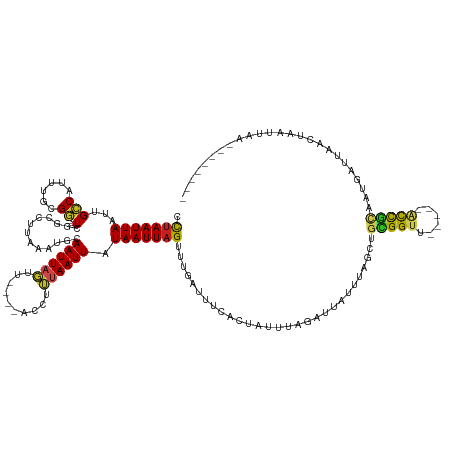
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:30:11 2011