
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 8,075,067 – 8,075,220 |
| Length | 153 |
| Max. P | 0.514054 |

| Location | 8,075,067 – 8,075,220 |
|---|---|
| Length | 153 |
| Sequences | 12 |
| Columns | 156 |
| Reading direction | reverse |
| Mean pairwise identity | 72.45 |
| Shannon entropy | 0.59486 |
| G+C content | 0.40502 |
| Mean single sequence MFE | -27.82 |
| Consensus MFE | -18.86 |
| Energy contribution | -17.91 |
| Covariance contribution | -0.95 |
| Combinations/Pair | 1.43 |
| Mean z-score | -0.16 |
| Structure conservation index | 0.68 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.514054 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 8075067 153 - 23011544 AUAGCUAAUCCAAAUAAAUAUUAAUUAAAUUCCAUU-UUCUUUUAUGAU--UACAGAACGAUUCAACGAAUCCAUAUUCGAUGCCCUGCCCACAUAUCUGCUGAACAUUUGUGAGGUGUGCGGCAGCUCUCAGUGUCCGUACUGCUCCAUCUACAA ...(.(((((...(((((....(((........)))-....))))))))--))).(((.(((((...)))))....)))((((..((((((((((.((.((.........)))).))))).)))))....(((((....)))))...))))..... ( -24.20, z-score = 0.61, R) >droMoj3.scaffold_6500 21213683 149 - 32352404 -------CUAUAACUACAUAUAUUUAAUACUAUACCUUCCUGUUACUCCCAUACAGAGCGCUUCAACGAGUCCAUCUUUGAUGCUCUGCCCACAUAUCUGCUAAAUAUUUGCGAGGUGUGCGGCAGCUCGCAGUGCCCCUACUGUUCCAUCUAUAA -------................................................((((((.((((.((.....)).)))).))..(((((((((.((.((.........)))).))))).))))))))((((((....))))))........... ( -32.30, z-score = -1.56, R) >droVir3.scaffold_12963 16272318 144 - 20206255 ------CUAAAUUGCAUUUUA---UAAUCGCUUUUU-UGUUUUUCCUCC--CACAGAGCGCUUCAACGAGUCCAUUUUCGAUGCACUGCCCACAUAUCUGCUGAACAUUUGCGAGGUGUGCGGCAGCUCGCAGUGUCCAUACUGUUCCAUCUAUAA ------......(((((....---....(((((...-(((.........--.))))))))......((((......)))))))))((((((((((.((.((.........)))).))))).)))))...((((((....))))))........... ( -32.30, z-score = -0.73, R) >droWil1.scaffold_180708 6318415 142 - 12563649 -----------UGACAAUUGACACUAAUUUCGUUUC-UAUUUUCAAAUU--UGCAGAGCGCUUCAAUGAGUCAAUAUUCGAUGCUUUACCCACAUAUUUGCUUAACAUUUGUGAAGUGUGCGGCAGUUCGCAAUGUCCAUAUUGCUCCAUCUACAA -----------(((.(((.((.((.......)).))-.))).)))...(--(((...(((((((..(((((.(((((................))))).)))))((....)))))))))...))))...((((((....))))))........... ( -24.09, z-score = 1.05, R) >droAna3.scaffold_12916 11798526 137 + 16180835 -------------UUAAUCGAAAUAAGU--AAUUAA-U-CUAUGUAUCU--UGCAGAGCGAUUCAACGAAUCAAUAUUUGAUGCCCUGCCCACAUAUCUGCUGAACAUUUGCGAGGUGUGCGGCAGUUCGCAGUGUCCCUAUUGUUCCAUAUACAA -------------...............--......-.-...(((((..--((..(((((((((...))))).........(((.((((((((((.((.((.........)))).))))).)))))...)))...........)))))).))))). ( -29.40, z-score = -0.20, R) >droPer1.super_46 57830 140 - 590583 -----------AAAUAAUGCCAGAUAAU--AAUGUA-UGAUGUUUAUCU--UUUAGAACGCUUCAAUGAAUCAAUUUUCGAUGCUUUGCCCACAUAUUUGCUAAACAUAUGCGAGGUAUGCGGCAGCUCUCAGUGCCCGUACUGUUCCAUCUACAA -----------.................--..((((-.((((.......--........((..((.((((......)))).))...((((..(((((((((.........)))).))))).))))))...(((((....)))))...)))))))). ( -24.90, z-score = 0.87, R) >dp4.chr4_group4 5587252 140 + 6586962 -----------AAAUAAUGCCAGUUAAU--AAUGUA-UGUUGUUUAUCU--UUUAGAACGCUUCAAUGAAUCAAUUUUCGAUGCUUUGCCCACAUAUUUGCUAAACAUAUGCGAGGUAUGCGGCAGCUCUCAGUGCCCGUACUGUUCCAUCUACAA -----------.................--..((((-(((.(((((.(.--..((....((..((.((((......)))).))....)).....))...).)))))))))))).((((((.((((........))))))))))............. ( -22.70, z-score = 1.52, R) >droSim1.chr2L 7855835 143 - 22036055 -----------CUAUAAACCAAAUAAAUCUAAUAUUCUUCUAUGUUAAU--UGCAGAACGAUUCAACGAAUCAAUAUUCGAUGCCCUGCCCACAUAUCUGCUGAACAUUUGCGAGGUGUGCGGCAGCUCUCAGUGUCCGUACUGCUCCAUCUACAA -----------..................((((((......))))))..--.((((.(((((((...))))).......(((((.((((((((((.((.((.........)))).))))).)))))......))))).)).))))........... ( -30.80, z-score = -1.69, R) >droSec1.super_3 3571960 151 - 7220098 --AUCUAAUCCAAAUAAAUGUUAAAUACAUCCUACU-UUCUUCUAUGCU--UACAGAACGAUUCAACGAAUCGAUAUUCGAUGCCCUGCCCACAUAUCUGCUGAACAUUUGUGAGGUGUGCGGCAGCUCUCAGUGUCCGUACUGCUCCAUCUACAA --...............((((.....))))......-............--..(((.(((((((...)))))((((((.((.(..((((((((((.((.((.........)))).))))).)))))..))))))))).)).)))............ ( -27.70, z-score = -0.29, R) >droYak2.chr2L 10705133 149 - 22324452 --AACUAAAUUAAAUAAUUGUUUAAUAAAUCUUAUU-UCCCUCU--GCU--UUCAGAACGGUUCAACGAAUCAAUAUUCGAUGCACUGCCUACAUAUCUGCUGAACAUUUGUGAGGUGUGCGGUAGCUCUCAGUGUCCGUACUGCUCCAUCUACAA --((((..(((((((....)))))))..........-....(((--(..--..))))..))))...(((((....))))).(((((.((((.((((..((.....))..)))))))))))))((((....(((((....)))))......)))).. ( -27.80, z-score = -0.57, R) >droEre2.scaffold_4929 16980673 151 - 26641161 --AACUAAAUAAAGUAAAUUGUAUAUAAAUCUUAUU-UUCUUCUAUGAU--UACAGAACGGUUCAACGAAUCAAUAUUUGAUGCCCUGCCCACAUAUCUGCUGAACAUUUGUGAGGUAUGCGGCAGCUCUCAGUGUCCGUACUGCUCCAUCUACAA --................(((((....((((.....-.........)))--).(((.((((..((..((((((.....))))...(((((..(((((((((.........)).))))))).)))))...))..)).)))).))).......))))) ( -26.84, z-score = 0.13, R) >anoGam1.chr3R 10368131 142 - 53272125 -----------AAAUGAUCUCCCUUUAUCUAACAACCGUUUAUCUCAU---UCCAGAACGCUUCAAUGAGACCAUCUUCGAUUCGCUGCAAACCUACCUGCUGAACAUCUGCGAAGUGUGCGGCAGCUCGCAGUGCCCGUACUGCUCGAUCUACAA -----------....((((.......................((((((---(..((....))..)))))))........((...(((((..((((...(((.(.....).))).)).))...)))))..((((((....))))))))))))..... ( -30.80, z-score = -1.09, R) >consensus ___________AAAUAAUUGAAAUUAAUAUAAUAUU_UUCUUUUAAUCU__UACAGAACGCUUCAACGAAUCAAUAUUCGAUGCCCUGCCCACAUAUCUGCUGAACAUUUGCGAGGUGUGCGGCAGCUCUCAGUGUCCGUACUGCUCCAUCUACAA ...............................................................................((((..(((((..(((((((((.........)).))))))).)))))....(((((....)))))...))))..... (-18.86 = -17.91 + -0.95)

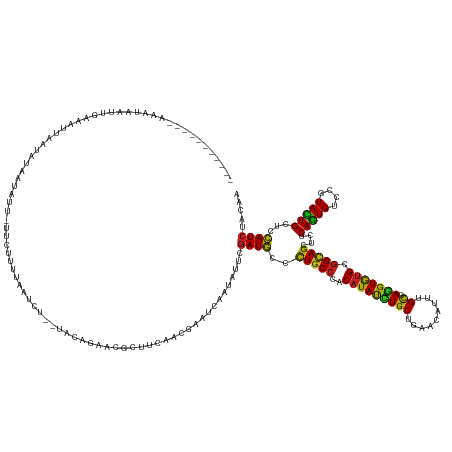
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:25:00 2011