| Sequence ID | dm3.chr3R |
|---|---|
| Location | 15,797,880 – 15,797,974 |
| Length | 94 |
| Max. P | 0.928463 |

| Location | 15,797,880 – 15,797,974 |
|---|---|
| Length | 94 |
| Sequences | 12 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 73.03 |
| Shannon entropy | 0.54697 |
| G+C content | 0.43107 |
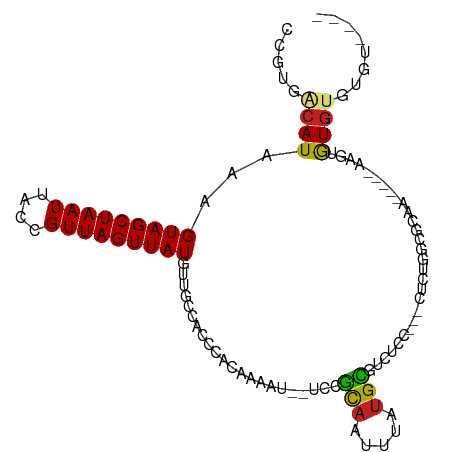
| Mean single sequence MFE | -26.79 |
| Consensus MFE | -8.38 |
| Energy contribution | -8.13 |
| Covariance contribution | -0.25 |
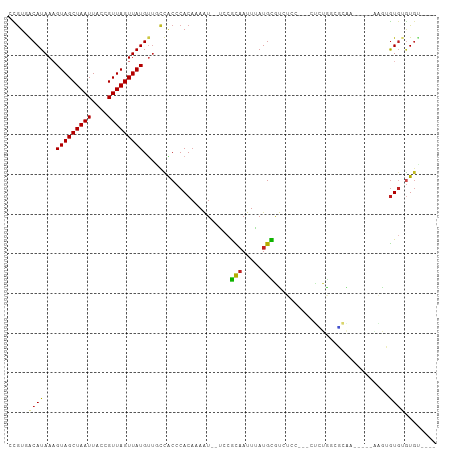
| Combinations/Pair | 1.31 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.31 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.928463 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
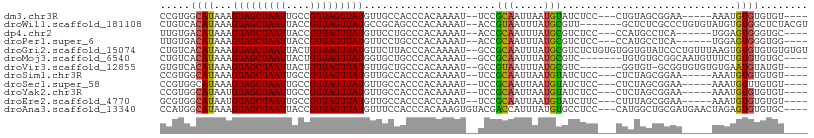
>dm3.chr3R 15797880 94 + 27905053 CCGUGGCAUAAAGUAGCUAAUUGCCGUUAGUUAUGUUGCCACCCACAAAAU--UCCGCAAUUAAUGUAUCUCC---CUGUAGCGGAA-----AAAUGUGUGUGU---- ..((((((....(((((((((....)))))))))..)))))).((((...(--(((((...............---.....))))))-----...)))).....---- ( -28.15, z-score = -4.01, R) >droWil1.scaffold_181108 4009129 99 + 4707319 CUGUCACAUAAAGUAGCUAAUUACCGUUAGUUAUGCCGCAGCCCACAAAAU--ACCGUAAUUUAUGCGUU-------GCUCUCGCCCUGUGUAUGUGUGGCUCUACGU ..((((((((..(((((((((....)))))))))..(((((..........--..((((.....))))..-------((....)).)))))..))))))))....... ( -23.10, z-score = -0.85, R) >dp4.chr2 2910267 93 - 30794189 UUGUGACAUAAAGUAGCUAAUUACCGUUAGUUAUGUUCCUGCCCACAAAAU--ACCGCAAUUUAUGCGUCUCC---CCAUGCCUCA------UGGAGUGGGUGC---- ..(..((((((..((((........)))).))))))..).((((((.....--..((((.....)))).....---(((((...))------))).))))))..---- ( -26.50, z-score = -2.54, R) >droPer1.super_6 2928336 93 - 6141320 UUGUGACAUAAAGUAGCUAAUUACCGUUAGUUAUGUUCCUGCCCACAAAAU--ACCGCAAUUUAUGCGUCUCC---CCAUGCCUCA------UGGAGUGGGUGG---- ....(((((((..((((........)))).))))))).(..(((((.....--..((((.....)))).....---(((((...))------))).)))))..)---- ( -27.30, z-score = -2.62, R) >droGri2.scaffold_15074 2395282 106 - 7742996 CUGUCACAUAAAGUAGCUAAUUACUGUUAGUUAUGUUCUUACCCACAAAAU--GCCGCAAUUUAUGCGUCUCUGUGUGGUGUAUCCCUGUUUAAGUGUGUGUGUGUGU ....((((((..(((((((((....))))))))).....((((((((....--(.((((.....)))).)....))))).)))............))))))....... ( -26.60, z-score = -2.74, R) >droMoj3.scaffold_6540 9018401 95 - 34148556 CUGUCACAUAAAGUAGCUAAUUACUGUUAGUUAUGGUGCUGCCCACAAAAU--GCCGCAAUUUAUGCGUC-------UGUGUGCGGCAAUGUUUCUGUGUGUGC---- ....((((((..(((((((((....)))))))))..((((((.((((....--(.((((.....)))).)-------)))).)))))).......))))))...---- ( -29.40, z-score = -1.81, R) >droVir3.scaffold_12855 7523013 94 + 10161210 GUGUCACAUAAAGUAGCUAAUUACUGUUAGUUAUGUUGCUGCCCACAAAAU--GCCGUAAUUUAUGCGUC-------GGUGU-GCGGUGUGUGUGAAUGUAUGU---- ...(((((((..(((((((((....)))))))))...(((((.(((...((--((..........)))).-------.))).-))))).)))))))........---- ( -27.00, z-score = -1.50, R) >droSim1.chr3R 21841089 94 - 27517382 CCGUGGCAUAAAGUAGCUAAUUGCCGUUAGUUAUGUUGCCACCCACAAAAU--UCCGCAAUUAAUGUAUCUCC---CUCUAGCGGAA-----AAAUGUGUGUGU---- ..((((((....(((((((((....)))))))))..)))))).((((...(--(((((...............---.....))))))-----...)))).....---- ( -28.15, z-score = -4.38, R) >droSec1.super_58 39082 94 - 188520 CCGUGGCAUAAAGUAGCUAAUUGCCGUUAGUUAUGUUGCCACCCACAAAAU--UCCGCAAUUAAUGUAUCUCC---CUCUAGCGGAA-----AAAUGUUUGUGU---- ..((((((....(((((((((....)))))))))..)))))).((((((.(--(((((...............---.....))))))-----.....)))))).---- ( -29.05, z-score = -4.91, R) >droYak2.chr3R 4578648 94 + 28832112 CCGUGGCAUAAUGUAGCUAAUUGCCGUUAGUUAUGUUGCCACCCACAAAAU--UCCGCAAUUAAUGUAUCUCC---CUCUAGCGGAA-----AAAUGUGUGUGU---- ..((((((..(((((((((((....))))))))))))))))).((((...(--(((((...............---.....))))))-----...)))).....---- ( -28.05, z-score = -4.14, R) >droEre2.scaffold_4770 11883247 94 - 17746568 GCGUGGCAUAAUGUAGCUAAUUGCCGUUAGUUAUGUUGCCACCCACCAAAU--UCCGCAAUUAAUGUAUCUUC---CUUUAGCGGAA-----AAAUGUGUGUGU---- ..((((((..(((((((((((....))))))))))))))))).(((((..(--(((((...............---.....))))))-----...)).)))...---- ( -27.25, z-score = -3.11, R) >droAna3.scaffold_13340 2377168 101 + 23697760 CCAUGGCAUAAAGUAGCUAAUUACCGUUAGUUAUGUUUCCACCCACAAAGUGUACGACCAUUUAUGUGCCUCC---CAUGGCUGCGAUGAACUAGAGUGUGUGC---- ((((((......(((((((((....))))))))).........(((((((((......))))).))))....)---)))))..(((....((....))...)))---- ( -20.90, z-score = 0.27, R) >consensus CCGUGACAUAAAGUAGCUAAUUACCGUUAGUUAUGUUGCCACCCACAAAAU__UCCGCAAUUUAUGCGUCUCC___CUCUGGCGCAA_____AAGUGUGUGUGU____ .....((((...(((((((((....)))))))))......................(((.....))).............................))))........ ( -8.38 = -8.13 + -0.25)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:28:47 2011