| Sequence ID | dm3.chr2L |
|---|---|
| Location | 8,052,511 – 8,052,631 |
| Length | 120 |
| Max. P | 0.994139 |

| Location | 8,052,511 – 8,052,631 |
|---|---|
| Length | 120 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.85 |
| Shannon entropy | 0.37997 |
| G+C content | 0.49792 |
| Mean single sequence MFE | -43.66 |
| Consensus MFE | -31.88 |
| Energy contribution | -31.63 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.54 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.73 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.67 |
| SVM RNA-class probability | 0.994139 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
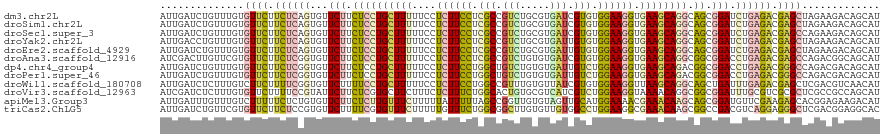
>dm3.chr2L 8052511 120 + 23011544 AUUGAUCUGUUUGUGUUCUUCUCAGUGUUCUUCUCCUGCUUUUUCCUCUUCCUCGCCGUCUGCGUGAUCGUGUGGAAGGUGAAGCAGGCAGCGGAUCUGAGACGAGCUAGAAGACAGCAU ......((((((..((((.((((((...(((.((((((((((....((((((.(((.(((.....))).))).)))))).)))))))).)).))).)))))).))))....))))))... ( -49.20, z-score = -3.81, R) >droSim1.chr2L 7831583 120 + 22036055 AUUGAUCUGUUUGUGUUCUUCUCAGUGUUCUUCUCCUGCUUUUUCCUCUUCCUCGCCGUCUGCGUGAUCGUGUGGAAGGUGAAGCAGGCAGCGGAUCUGAGACGAGCUAGAAGACAGCAU ......((((((..((((.((((((...(((.((((((((((....((((((.(((.(((.....))).))).)))))).)))))))).)).))).)))))).))))....))))))... ( -49.20, z-score = -3.81, R) >droSec1.super_3 3549243 120 + 7220098 AUUGAUCUGUUUGUGUUCUUCUCAGUGUUCUUCUCCUGCUUUUUCCUCUUCCUCGCCGUCUGCGUGAUCGUGUGGAAGGUGAAGCAGGCAGCGGAUCUGAGACGAGCCAGAAGACAGCAU .(((.(((.((((.((((.((((((...(((.((((((((((....((((((.(((.(((.....))).))).)))))).)))))))).)).))).)))))).))))))))))))))... ( -48.80, z-score = -3.57, R) >droYak2.chr2L 10682433 120 + 22324452 AUUGACCUGUUUGUGUUCUUCUCAGUGUUCUUCUCCUGCUUUUUCCUCUUCCUCGCCGUCUGCGUGAUUGUGUGGAAGGUGAAGCAGGCAGCGGAUCUGAGACGAGCUAGAAGACAGCAU ......((((((..((((.((((((...(((.((((((((((....((((((.(((.(((.....))).))).)))))).)))))))).)).))).)))))).))))....))))))... ( -49.40, z-score = -3.98, R) >droEre2.scaffold_4929 16957940 120 + 26641161 AUUGAUCUGUUUGUGUUCUUCUCAGUGUUCUUCUCCUGCUUUUUCCUCUUCCUCGCCGUCUGCGUGAUUGUGUGGAAGGUGAAGCAGGCAGCGGAUCUGAGACGAGCUAGAAGACAGCAU ......((((((..((((.((((((...(((.((((((((((....((((((.(((.(((.....))).))).)))))).)))))))).)).))).)))))).))))....))))))... ( -49.20, z-score = -3.93, R) >droAna3.scaffold_12916 11775276 120 - 16180835 AUCGACUUGUUCGUGUUCUUCUCGGUGUUCUUCUCCUGCUUUUUCCUCUUCCUCGCCGUCUGUGUGAUCGUGUGGAAGGUGAAGCAGGCGGCGGACCUGAGACGAGCCAGACGGCAGCAU ......((((..(.((((.((((((.((((.((.((((((((....((((((.(((.(((.....))).))).)))))).)))))))).)).)))))))))).)))))..))))...... ( -48.50, z-score = -2.41, R) >dp4.chr4_group4 5561627 120 - 6586962 AUUGAUCUGUUUGUGUUCUUCUCGGUGUUCUUCUCCUGCUUUUUCCUCUUCCUGGCUGUCUGUGUGAUUGUCUGGAAGGUGAAGCAGACGGCGGACCUGAGACGGGCCAGACGACAGCAU .(((.((.(((((.((((.((((((.((((.((..(((((((....((((((.(((.(((.....))).))).)))))).)))))))..)).)))))))))).))))))))))))))... ( -46.50, z-score = -2.86, R) >droPer1.super_46 32238 120 + 590583 AUUGAUCUGUUUGUGUUCUUCUCGGUGUUCUUCUCCUGCUUUUUCCUCUUCCUGGCUGUCUGUGUGAUUGUCUGGAAGGUGAAGCAGACGGCGGACCUGAGACGGGCCAGACGACAGCAU .(((.((.(((((.((((.((((((.((((.((..(((((((....((((((.(((.(((.....))).))).)))))).)))))))..)).)))))))))).))))))))))))))... ( -46.50, z-score = -2.86, R) >droWil1.scaffold_180708 6294774 120 + 12563649 AUUGAUCUCUUUGUCUUCUUUUCGGUGUUCUUUUCCUGCUUUUUCCUCUUCCUGGCCGUUUGUGUUAUCGUGUGGAAGGUUAAGCAGGCAGCUGAUUUGAGACGAGCUCGACGUCAACAU .(((((.((((((((((....(((((........(((((((.....((((((..((.((.......)).))..))))))..)))))))..)))))...))))))))...)).)))))... ( -31.20, z-score = -0.85, R) >droVir3.scaffold_12963 16249553 120 + 20206255 AUCGAUCUCUUUGUGUUCUUUUCCGUAUUCUUCUCGUGCUUCUUUCUCUUUCUGGCACUGUGCGUCAUCGUCUGGAAGGUAAAACAGGCGGCGGAUUUGCGUCGCGCUCGCCGCCAGCAU ...........(((.((.((((((...........(((((.............)))))...(((....)))..)))))).)).)))((((((((....(((...)))))))))))..... ( -30.62, z-score = -0.05, R) >apiMel3.Group3 9351374 120 - 12341916 AUUGAUUUGUUUGUCUUUUUCUCUGUGUUCUUCUCUUGUUUCUUUUUAUUUUUAGCCGGUUGUGUAGUUGCAUGGAAAACGAAACAAGCAGCGGAUGUUCGAAGAGCACGGAGAAGACAU ...........(((((...((((((((((((((.(((((((((((((.....((((..........))))....))))).)))))))).(((....))).))))))))))))))))))). ( -35.60, z-score = -2.50, R) >triCas2.ChLG5 16752665 120 + 18847211 AUUGAUCUGUUCGUGUUCUUCUCCGUGUUCUUUUCGUGUUUCUUUUUGUUUCUGGCGGCUUGUGUUGUGGCCUGGAAGGCGAAACAAGCGGCCGACGUCAGGAGGGCUCGACGGAGGCAC ..((.(((((.((.((((((((.(((.........(.....)..........((((.((((((....(.(((.....))).).)))))).)))))))..)))))))).)))))))..)). ( -39.20, z-score = -0.46, R) >consensus AUUGAUCUGUUUGUGUUCUUCUCAGUGUUCUUCUCCUGCUUUUUCCUCUUCCUCGCCGUCUGCGUGAUCGUGUGGAAGGUGAAGCAGGCAGCGGAUCUGAGACGAGCUAGAAGACAGCAU ..............((((.((((((...(((.((((((((((....((((((.(((.(((.....))).))).)))))).)))))))).)).))).)))))).))))............. (-31.88 = -31.63 + -0.25)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:24:58 2011