
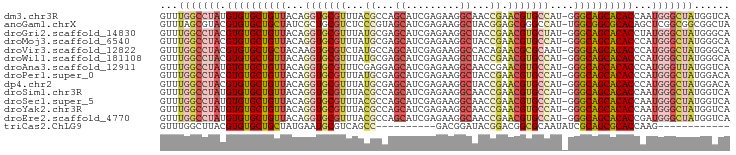
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 14,631,777 – 14,631,871 |
| Length | 94 |
| Max. P | 0.986802 |

| Location | 14,631,777 – 14,631,871 |
|---|---|
| Length | 94 |
| Sequences | 14 |
| Columns | 95 |
| Reading direction | reverse |
| Mean pairwise identity | 84.98 |
| Shannon entropy | 0.36505 |
| G+C content | 0.56921 |
| Mean single sequence MFE | -40.16 |
| Consensus MFE | -29.68 |
| Energy contribution | -30.20 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.74 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.25 |
| SVM RNA-class probability | 0.986802 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
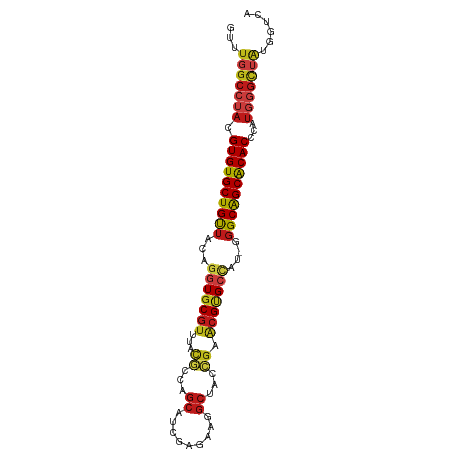
>dm3.chr3R 14631777 94 - 27905053 GUUUGGCCUAUGUGUGCUGUUACAGGUGCGUUUACGCCAGCAUCGAGAAGGCAACCGAACGUGCCAU-GGGCAGCACACCAAUGGGCUAUGGUCA ...((((((((((((((((((...((..(((((..(((..(.....)..)))....)))))..))..-.))))))))))..))))))))...... ( -42.10, z-score = -2.94, R) >anoGam1.chrX 11043653 94 + 22145176 GUUUAGCGUACGUGUGCUGCUAUCGCUGCGUCUCCCGUAGCAUCGAGAAGGCUACGGAGCGGGCCAU-UGGCGGCGCACAGCUCGGCGGCGGCUA ...((((....(((((((((((..(((.((.((((.(((((.........))))))))))))))...-))))))))))).(((....))).)))) ( -42.40, z-score = -0.55, R) >droGri2.scaffold_14830 2580293 94 + 6267026 GUUUGGCCUACGUGUGCUGUUACAGGUGCGUUUAUGCGAGCAUCGAGAAGGCUACCGAACGUGCUAU-GGGCAGCACACCUAUGGGCUAUGGGCA ((((((((((.((((((((((...((..(((((.....(((.........)))...)))))..))..-.))))))))))...)))))))..))). ( -38.00, z-score = -1.81, R) >droMoj3.scaffold_6540 19075189 94 + 34148556 GUUUGGCCUACGUGUGCUGUUACAGGUGCGUUUAUGCGAGCAUCGAGAAGGCUACCGAACGUGCCAU-GGGCAGCACACCCAUGGGCUAUGGGCA ((((((((((.((((((((((...((..(((((.....(((.........)))...)))))..))..-.))))))))))...)))))))..))). ( -39.90, z-score = -1.90, R) >droVir3.scaffold_12822 3167596 94 - 4096053 GUUUGGCCUACGUGUGCUGCUACAAGUGCGUCUAUGCCAGCAUCGAGAAGGCCACAGAACGCGCAAU-GGGCAGCACACCCAUGGGCUAUGGGCA ((((((((((.((((((((((....((((((((..(((..(.....)..)))...)).))))))...-.))))))))))...)))))))..))). ( -42.00, z-score = -2.29, R) >droWil1.scaffold_181108 2863190 94 - 4707319 GUUUGGCCUACGUGUGCUGUUACAGGUGCGUUUAUGCGAGCAUCGAGAAGGCUACCGAACGUGCCAU-GGGCAGCACACCCAUGGGCUAUGGGCA ((((((((((.((((((((((...((..(((((.....(((.........)))...)))))..))..-.))))))))))...)))))))..))). ( -39.90, z-score = -1.90, R) >droAna3.scaffold_12911 2985528 94 - 5364042 GUUUGGCCUAUGUGUGCUGUUACAGGUGCGUUUCGAGGAGCAUCGAGAAGGCAACCGAACGUGCCAU-GGGCAGCACACCCAUGGGUUAUGGUCA ...((((((((((((((((((...((..((((.((.(..((.........))..)))))))..))..-.))))))))))..))))))))...... ( -39.00, z-score = -2.39, R) >droPer1.super_0 6169830 94 + 11822988 GUUUGGCCUACGUGUGCUGUUACAGGUGCGUUUAUGCGAGCAUCGAGAAGGCUACCGAACGUGCCAU-GGGCAGCACACCCAUGGGCUAUGGACA ((((((((((.((((((((((...((..(((((.....(((.........)))...)))))..))..-.))))))))))...)))))))..))). ( -40.40, z-score = -2.57, R) >dp4.chr2 14758327 94 - 30794189 GUUUGGCCUACGUGUGCUGUUACAGGUGCGUUUAUGCGAGCAUCGAGAAGGCUACCGAACGUGCCAU-GGGCAGCACACCCAUGGGCUAUGGACA ((((((((((.((((((((((...((..(((((.....(((.........)))...)))))..))..-.))))))))))...)))))))..))). ( -40.40, z-score = -2.57, R) >droSim1.chr3R 20673149 94 + 27517382 GUUUGGCCUAUGUGUGCUGUUACAGGUGCGUUUACGCCAGCAUCGAGAAGGCAACCGAACGUGCCAU-GGGCAGCACACCAAUGGGCUAUGGUCA ...((((((((((((((((((...((..(((((..(((..(.....)..)))....)))))..))..-.))))))))))..))))))))...... ( -42.10, z-score = -2.94, R) >droSec1.super_5 598516 94 + 5866729 GUUUGGCCUAUGUGUGCUGUUACAGGUGCGUUUACGCCAGCAUCGAGAAGGCAACCGAACGUGCCAU-GGGCAGCACACCAAUGGGCUAUGGUCA ...((((((((((((((((((...((..(((((..(((..(.....)..)))....)))))..))..-.))))))))))..))))))))...... ( -42.10, z-score = -2.94, R) >droYak2.chr3R 3290727 94 - 28832112 GUUUGGCCUAUGUGUGCUGUUACAGGUGCGUUUACGCCAGCAUCGAGAAGGCAACCGAACGUGCCAU-GGGCAGCACACCAAUGGGCUAUGGUCA ...((((((((((((((((((...((..(((((..(((..(.....)..)))....)))))..))..-.))))))))))..))))))))...... ( -42.10, z-score = -2.94, R) >droEre2.scaffold_4770 10743362 94 + 17746568 GUUUGGCCUAUGUGUGCUGUUACAGGUGCGUUUACGCCAGCAUCGAGAAGGCAACCGAACGUGCCAU-GGGCAGCACACCGAUGGGCUAUGGUCA ...((((((((((((((((((...((..(((((..(((..(.....)..)))....)))))..))..-.))))))))))..))))))))...... ( -42.10, z-score = -2.86, R) >triCas2.ChLG9 14498349 73 - 15222296 GUUUGGCUUACGUGUGCUGCUAUGAAUGCGUCAGCC----------GACGGAUACGGACGGCGCAAUAUCGCAGCGCACCAAG------------ ...........(((((((((.(((..((((((..((----------(.......)))..)))))).))).)))))))))....------------ ( -29.70, z-score = -2.18, R) >consensus GUUUGGCCUACGUGUGCUGUUACAGGUGCGUUUACGCCAGCAUCGAGAAGGCUACCGAACGUGCCAU_GGGCAGCACACCCAUGGGCUAUGGUCA ...(((((((.((((((((((...(((((((...((...((.........))...)).)))))))....))))))))))...)))))))...... (-29.68 = -30.20 + 0.52)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:26:01 2011