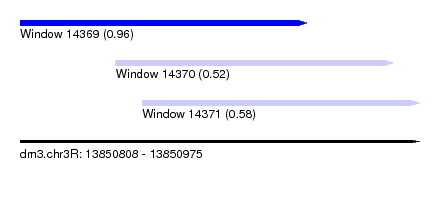
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 13,850,808 – 13,850,975 |
| Length | 167 |
| Max. P | 0.956957 |

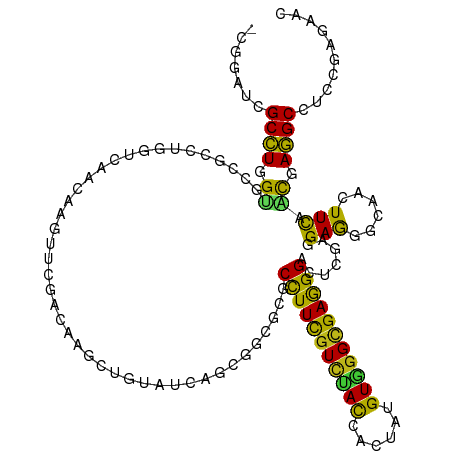
| Location | 13,850,808 – 13,850,928 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 121 |
| Reading direction | forward |
| Mean pairwise identity | 75.53 |
| Shannon entropy | 0.53134 |
| G+C content | 0.56795 |
| Mean single sequence MFE | -47.47 |
| Consensus MFE | -22.14 |
| Energy contribution | -20.24 |
| Covariance contribution | -1.90 |
| Combinations/Pair | 1.70 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.47 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.64 |
| SVM RNA-class probability | 0.956957 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
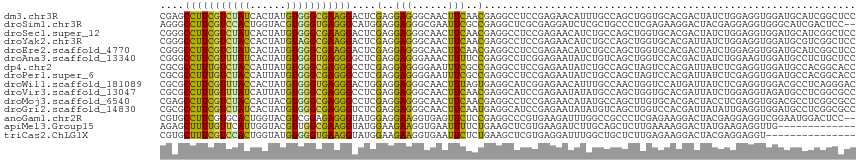
>dm3.chr3R 13850808 120 + 27905053 -CGGAUCGCCUGGUGCCGCCUGGUCAACAAGUUCGACAAGCUCUAUCAGCGGCGAGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAAC -((((..((((.(((((((.((((.....((((.....))))..)))))))))((((((((((((((......))))))))))).)))...((((....)))).)).)))).))))..... ( -52.60, z-score = -3.21, R) >droSim1.chr3R 15784460 118 + 27517382 --CGA-GGCCUGGGCCCGUCUGGAUCACAAGUUCGAUCUGAUGUACGCCAAGAGGGCCUUCGUCCACUGGUACGUGGGUGAGGGCAUGGAGGAGGGCGAAUUCGCCGAGGCUCGCGAGGAU --(((-(.(((.(((.((((.(((((........)))))))))..((((......((((((..((((......))))..)))))).........)))).....))).)))))))....... ( -50.36, z-score = -1.26, R) >droSec1.super_12 1817160 120 - 2123299 -CGGAUCGCCUGGUGCCGCCUGGUCAACAAGUUCGACAAGCUCUAUCAGCGGCGGGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAAC -((((..((((.(((((((.((((.....((((.....))))..)))))))))((((((((((((((......))))))))))).)))...((((....)))).)).)))).))))..... ( -51.70, z-score = -2.75, R) >droYak2.chr3R 2446404 120 + 28832112 -CGGAUCGCCUGGUGCCGCCUGGUCAACAAGUUCGACAAGCUUUACCAGCGGCGGGCCUUCGUCUACCACUAUGUAGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAAC -((((..((((.(((((((.((((....(((((.....))))).)))))))))((((((((((((((......))))))))))).)))...((((....)))).)).)))).))))..... ( -57.50, z-score = -4.56, R) >droEre2.scaffold_4770 9970765 120 - 17746568 -CGGAUCGCCUGGUGCCGCCUGGUCAACAAGUUCGACAAGCUCUACCAGCGGCGGGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAAC -((((..((((.(((((((.((((.....((((.....))))..)))))))))((((((((((((((......))))))))))).)))...((((....)))).)).)))).))))..... ( -54.30, z-score = -3.22, R) >droAna3.scaffold_13340 11836523 120 - 23697760 -CGGAUAGCCUGGUGCCGGCUGGUGAACAAGUUCGACAAACUGUAUCAGAAGCGGGCCUUCGUUUAUCACUAUGUGGGUGAGGGGCUCGAGGAGGGAAACUUUUCCGAGGCCUCGGAGAAU -(((.....)))..(((.(((..(((...((((.....))))...)))..))).)))(((((....(((((.....)))))(((.((((..((((....))))..)))).))))))))... ( -44.40, z-score = -1.54, R) >dp4.chr2 28905906 120 + 30794189 -CGGAUCGCCUGGUGCCGGCUGGUCAAUAAGUUCGAUAAGCUGUAUCAGAGGCGCGCCUUUGUCUACCAUUAUGUGGGCGAGGGCCUCGAGGAGGGGAAUUUCGCCGAGGCCUCCGAGAAU -((((....(((((((.((((.(((.........))).))))))))))).(((..((((((((((((......))))))))))))((((.((((.....))))..)))))))))))..... ( -48.90, z-score = -1.31, R) >droPer1.super_6 4232528 120 + 6141320 -CGGAUCGCCUGGUGCCGGCUGGUCAAUAAGUUCGAUAAGCUGUAUCAGAGGCGCGCCUUUGUCUACCAUUAUGUGGGCGAGGGCCUCGAGGAGGGGAAUUUCGCCGAGGCCUCCGAGAAU -((((....(((((((.((((.(((.........))).))))))))))).(((..((((((((((((......))))))))))))((((.((((.....))))..)))))))))))..... ( -48.90, z-score = -1.31, R) >droWil1.scaffold_181089 7532804 120 - 12369635 -CGUAUCGCCUGGUGUCGCCUGGUGAAUAAGUUCGACAAGCUCUAUCAGCGGCGCGCCUUCGUUUACCACUAUGUGGGUGAGGGACUGGAGGAGGGCAACUUUAGUGAGGCAUCGGAGAAC -....((..(((((((((((((.(((...((((.....))))...))).))).)).((((((..(((......)))..))))))(((((((........)))))))..)))))))).)).. ( -39.50, z-score = -0.01, R) >droVir3.scaffold_13047 12397668 120 - 19223366 -CGGAUAGCCUGGUGCCGCCUGGUCAACAAAUUCGAUAAGCUUUAUCAGCGGCGCGCCUUUGUUUAUCAUUAUGUGGGCGAGGGCCUCGAGGAGGGCAACUUCAACGAGGCAUCCGAGAAU -(((((.(((..(((((((.((.....)).....((((.....)))).)))))))((((((((((((......)))))))))))).(((..((((....))))..)))))))))))..... ( -50.30, z-score = -3.25, R) >droMoj3.scaffold_6540 31464892 120 - 34148556 -CGGAUUGCCUGGUGCCGCCUGGUCAACAAGUUCGACAAGCUCUACCAGCGACGAGCCUUCGUCUACCACUACGUGGGCGAGGGCCUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAAC -((((..((((.((..(((.((((.....((((.....))))..))))))).(((((((((((((((......))))))))))).))))..((((....)))).)).)))).))))..... ( -51.30, z-score = -2.38, R) >droGri2.scaffold_14830 3565118 120 - 6267026 -CGGAUGGCCUGGUGCCGAUUAGUCAAUAAGUUCGAUAAGCUCUAUCAACGGCGCGCCUUCGUCUAUCACUAUGUGGGCGAGGGUCUCGAGGAGGGCAACUUCAAUGAGGCAUCCGAGAAU -((((((.....(((((((....)).....(((.((((.....))))))))))))((((((((((((......))))))))))))((((..((((....))))..)))).))))))..... ( -48.00, z-score = -2.93, R) >anoGam1.chr2R 1623925 118 + 62725911 GCCGA-GGCCUGGGCCCGUCUGGACCACAAGUUCGAUCUGAUGUACGCCAAGCGUGCCUUCGUGCACUGGUACGUCGGAGAGGGUAUGGAGGAAGGUGAGUUCUCCGAGGCCCGUGAAG-- ..((.-(((((..((((.(((((((.....))))).((((((((((((...))((((......))))..)))))))))))))))).(((((((.......)))))))))))))).....-- ( -46.10, z-score = -0.68, R) >apiMel3.Group15 6377691 118 - 7856270 GCUGA-AGCUUGGGCUCGACUUGAUCAUAAAUUUGAUCUUAUGUAUGCUAAAAGAGCUUUUGUUCAUUGGUACGUUGGCGAAGGUAUGGAAGAAGGUGAAUUUUCUGAAGCUCGUGAAG-- ((...-.))...(((...((..(((((......)))))....))..)))....(((((((.(((((((.........((....)).........))))))).....)))))))......-- ( -27.37, z-score = -0.08, R) >triCas2.ChLG1X 2817431 118 - 8109244 GCUGA-AGCUUGGGCCAGGUUGGACCAUAAAUUCGACUUGAUGUACGCCAAACGUGCUUUCGUCCACUGGUAUGUGGGUGAAGGUAUGGAAGAAGGUGAAUUCUCUGAAGCUCGUGAGG-- .....-(((((.((.(((((((((.......))))))))).....((((...(((((((((..((((......))))..)))))))))......))))......)).)))))(....).-- ( -40.80, z-score = -2.35, R) >consensus _CGGAUCGCCUGGUGCCGCCUGGUCAACAAGUUCGACAAGCUGUAUCAGCGGCGCGCCUUCGUCUACCACUAUGUGGGCGAGGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAAC .......((((.((..........................................(((((((((((......))))))))))).......(((......))).)).)))).......... (-22.14 = -20.24 + -1.90)

| Location | 13,850,848 – 13,850,964 |
|---|---|
| Length | 116 |
| Sequences | 15 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 73.76 |
| Shannon entropy | 0.57424 |
| G+C content | 0.55076 |
| Mean single sequence MFE | -44.11 |
| Consensus MFE | -13.85 |
| Energy contribution | -11.51 |
| Covariance contribution | -2.33 |
| Combinations/Pair | 1.76 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.31 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.522092 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 13850848 116 + 27905053 UCUAUCAGCGGCGAGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUUUGCCAGCUGGUGCACGACUAUCUGGAGGUGGAU ..((((((((((((((((((((((((......))))))))))).((((..((((....))))..)))).............)))))).))))))).....(((((....))))).. ( -48.20, z-score = -2.87, R) >droSim1.chr3R 15784498 114 + 27517382 UGUACGCCAAGAGGGCCUUCGUCCACUGGUACGUGGGUGAGGGCAUGGAGGAGGGCGAAUUCGCCGAGGCUCGCGAGGAUCUCGCUGCCCUCGAGAAGGACUACGAGGAGGUGG-- ....((((..(((.((((((..((((......))))..)))))).........((((....))))....)))(((((...)))))...(((((((.....)).))))).)))).-- ( -49.90, z-score = -1.61, R) >droSec1.super_12 1817200 116 - 2123299 UCUAUCAGCGGCGGGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUCUGCCAGCUGGUGCACGACUAUCUGGAGGUGGAU ...(((..(((.((.(((((((((((......))))))))))).((((..((((....))))..)))).)).)))....(((((.((((.((((.....)))).)))))))))))) ( -50.40, z-score = -3.00, R) >droYak2.chr3R 2446444 116 + 28832112 UUUACCAGCGGCGGGCCUUCGUCUACCACUAUGUAGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUCUGCCAGCUGGUGCACGAUUAUCUGGAGGUGGAU ..((((((((((((((((((((((((......))))))))))).((((..((((....))))..)))).............)))))).))))))).....(((((....))))).. ( -51.90, z-score = -3.89, R) >droEre2.scaffold_4770 9970805 116 - 17746568 UCUACCAGCGGCGGGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUCUGCCAGCUGGUGCACGACUAUCUGGAGGUGGAU ..((((((((((((((((((((((((......))))))))))).((((..((((....))))..)))).............)))))).))))))).....(((((....))))).. ( -52.50, z-score = -3.45, R) >droAna3.scaffold_13340 11836563 116 - 23697760 UGUAUCAGAAGCGGGCCUUCGUUUAUCACUAUGUGGGUGAGGGGCUCGAGGAGGGAAACUUUUCCGAGGCCUCGGAGAAUAUCUGUCAGCUGGUCCACGACUAUCUGGAAGUGGAU .....(((..(((((.(((((....(((((.....)))))(((.((((..((((....))))..)))).))))))))....)))))...)))((((((..((....))..)))))) ( -42.60, z-score = -1.49, R) >dp4.chr2 28905946 116 + 30794189 UGUAUCAGAGGCGCGCCUUUGUCUACCAUUAUGUGGGCGAGGGCCUCGAGGAGGGGAAUUUCGCCGAGGCCUCCGAGAAUAUCUGCCAGCUAGUCCACGAUUAUCUCGAGGUGGAU ....((.(((((..((((((((((((......))))))))))))((((.((((.....))))..))))))))).))....(((..((((.(((((...))))).))...))..))) ( -43.60, z-score = -0.79, R) >droPer1.super_6 4232568 116 + 6141320 UGUAUCAGAGGCGCGCCUUUGUCUACCAUUAUGUGGGCGAGGGCCUCGAGGAGGGGAAUUUCGCCGAGGCCUCCGAGAAUAUCUGCCAGCUAGUCCACGAUUAUCUCGAGGUGGAU ....((.(((((..((((((((((((......))))))))))))((((.((((.....))))..))))))))).))....(((..((((.(((((...))))).))...))..))) ( -43.60, z-score = -0.79, R) >droWil1.scaffold_181089 7532844 116 - 12369635 UCUAUCAGCGGCGCGCCUUCGUUUACCACUAUGUGGGUGAGGGACUGGAGGAGGGCAACUUUAGUGAGGCAUCGGAGAACAUUUGCCAACUGGUCCAUGAUUAUCUCGAGGUGGAC ((((((..((..((.((((((..(((......)))..)))))).((.(..((((....))))..).))))..))((((.(((..(((....)))..)))....))))..)))))). ( -35.90, z-score = -0.15, R) >droVir3.scaffold_13047 12397708 116 - 19223366 UUUAUCAGCGGCGCGCCUUUGUUUAUCAUUAUGUGGGCGAGGGCCUCGAGGAGGGCAACUUCAACGAGGCAUCCGAGAAUAUAUGCCAGCUGGUGCACGAUUAUCUGGAGGUAGAU ..((((((((((((((((.(((........))).))))).((((((((..((((....))))..))))))..)).........)))).))))))).....(((((....))))).. ( -43.10, z-score = -2.31, R) >droMoj3.scaffold_6540 31464932 116 - 34148556 UCUACCAGCGACGAGCCUUCGUCUACCACUACGUGGGCGAGGGCCUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUAUGCCAGCUUGUGCACGACUACCUCGAGGUGGAC ((((((.((.((((((..((((((((......))))))))((((((((..((((....))))..))))))))................)))))))).(((.....))).)))))). ( -53.10, z-score = -4.08, R) >droGri2.scaffold_14830 3565158 116 - 6267026 UCUAUCAACGGCGCGCCUUCGUCUAUCACUAUGUGGGCGAGGGUCUCGAGGAGGGCAACUUCAAUGAGGCAUCCGAGAAUAUAUGUCAGCUGGUCCACGAUUAUAUUGAGGUGGAU ........((((((((((((((((((......))))))))))))((((..((((....))))..))))))....((.(.....).)).))))((((((............)))))) ( -41.20, z-score = -1.94, R) >anoGam1.chr2R 1623965 114 + 62725911 UGUACGCCAAGCGUGCCUUCGUGCACUGGUACGUCGGAGAGGGUAUGGAGGAAGGUGAGUUCUCCGAGGCCCGUGAAGAUUUGGCCGCCCUCGAGAAGGACUACGAGGAGGUCG-- .((((((...))))))((((((((....))))........((((.(((((((.......)))))))..))))..))))....((((..(((((((.....)).))))).)))).-- ( -43.10, z-score = -0.80, R) >apiMel3.Group15 6377731 114 - 7856270 UGUAUGCUAAAAGAGCUUUUGUUCAUUGGUACGUUGGCGAAGGUAUGGAAGAAGGUGAAUUUUCUGAAGCUCGUGAAGAUCUUGCAGCUCUUGAAAAGGACUAUGAAGAGGUUG-- ............(((((((.(((((((.........((....)).........))))))).....))))))).....((((((.((..((((....))))...))..)))))).-- ( -24.97, z-score = 0.29, R) >triCas2.ChLG1X 2817471 112 - 8109244 UGUACGCCAAACGUGCUUUCGUCCACUGGUAUGUGGGUGAAGGUAUGGAAGAAGGUGAAUUCUCUGAAGCUCGUGAGGAUUUGGCUGCUCUUGAGAAGGACUACGAGGAGGU---- .(((.(((...(((((((((..((((......))))..))))))))).........((((((((((.....)).))))))))))))))(((((((.....)).)))))....---- ( -37.60, z-score = -2.12, R) >consensus UGUAUCAGCGGCGCGCCUUCGUCUACCACUAUGUGGGCGAGGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAAUAUCUGCCAGCUGGUGCACGACUAUCUGGAGGUGGAU .....((........(((((((((((......)))))))))))....(..(((......)))..)..................))............................... (-13.85 = -11.51 + -2.33)

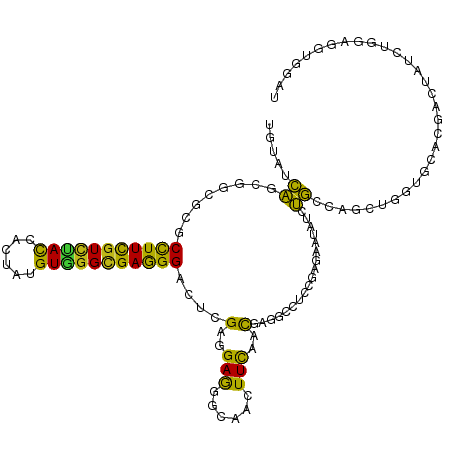
| Location | 13,850,859 – 13,850,975 |
|---|---|
| Length | 116 |
| Sequences | 15 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 71.95 |
| Shannon entropy | 0.63439 |
| G+C content | 0.57335 |
| Mean single sequence MFE | -44.68 |
| Consensus MFE | -13.79 |
| Energy contribution | -12.24 |
| Covariance contribution | -1.55 |
| Combinations/Pair | 1.73 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.31 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.582056 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
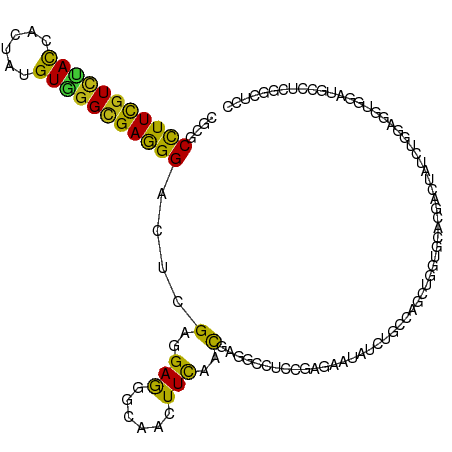
>dm3.chr3R 13850859 116 + 27905053 CGAGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUUUGCCAGCUGGUGCACGACUAUCUGGAGGUGGAUGCAUCGGCUCC (((((((((((((((......))))))))))).)))).(((((.(...........).))))).............((((((((((...(((((....))))).)))))))))).. ( -49.90, z-score = -3.11, R) >droSim1.chr3R 15784509 114 + 27517382 AGGGCCUUCGUCCACUGGUACGUGGGUGAGGGCAUGGAGGAGGGCGAAUUCGCCGAGGCUCGCGAGGAUCUCGCUGCCCUCGAGAAGGACUACGAGGAGGUGGGCAUCGACUCC-- ...((((((..((((......))))..)))))).....((((((((....))))((.(((((((((...)))))...(((((((.....)).)))))....)))).))..))))-- ( -53.30, z-score = -1.80, R) >droSec1.super_12 1817211 116 - 2123299 CGGGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUCUGCCAGCUGGUGCACGACUAUCUGGAGGUGGAUGCAUCGGCUCC (((((((((((((((......))))))))))).((((..((((....))))..))))...))))............((((((((((...(((((....))))).)))))))))).. ( -50.50, z-score = -2.80, R) >droYak2.chr3R 2446455 116 + 28832112 CGGGCCUUCGUCUACCACUAUGUAGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUCUGCCAGCUGGUGCACGAUUAUCUGGAGGUGGAUGCGUCGGCUCC .((((((((((((((......))))))))))).((((..((((....))))..))))(((..((....(((((.((((.((((.....)))).)))))))))....))..)))))) ( -50.80, z-score = -2.82, R) >droEre2.scaffold_4770 9970816 116 - 17746568 CGGGCCUUCGUCUAUCACUAUGUGGGCGAAGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUCUGCCAGCUGGUGCACGACUAUCUGGAGGUGGAUGCAUCGGCUCC (((((((((((((((......))))))))))).((((..((((....))))..))))...))))............((((((((((...(((((....))))).)))))))))).. ( -50.50, z-score = -2.80, R) >droAna3.scaffold_13340 11836574 116 - 23697760 CGGGCCUUCGUUUAUCACUAUGUGGGUGAGGGGCUCGAGGAGGGAAACUUUUCCGAGGCCUCGGAGAAUAUCUGUCAGCUGGUCCACGACUAUCUGGAAGUGGAUGCCUCUGCUCC ((((((((......(((((.....))))))))))))).((((((((....))))(((((..((((.....)))).......((((((..((....))..)))))))))))..)))) ( -42.90, z-score = -0.72, R) >dp4.chr2 28905957 116 + 30794189 CGCGCCUUUGUCUACCAUUAUGUGGGCGAGGGCCUCGAGGAGGGGAAUUUCGCCGAGGCCUCCGAGAAUAUCUGCCAGCUAGUCCACGAUUAUCUCGAGGUGGAUGCCACGGCACC ...((((((((((((......))))))))))))(((((((.((.(.....).))....))).))))......((((.....((((((............)))))).....)))).. ( -43.70, z-score = -0.63, R) >droPer1.super_6 4232579 116 + 6141320 CGCGCCUUUGUCUACCAUUAUGUGGGCGAGGGCCUCGAGGAGGGGAAUUUCGCCGAGGCCUCCGAGAAUAUCUGCCAGCUAGUCCACGAUUAUCUCGAGGUGGAUGCCACGGCACC ...((((((((((((......))))))))))))(((((((.((.(.....).))....))).))))......((((.....((((((............)))))).....)))).. ( -43.70, z-score = -0.63, R) >droWil1.scaffold_181089 7532855 116 - 12369635 CGCGCCUUCGUUUACCACUAUGUGGGUGAGGGACUGGAGGAGGGCAACUUUAGUGAGGCAUCGGAGAACAUUUGCCAACUGGUCCAUGAUUAUCUCGAGGUGGACGCCUCAGGGAC (.(((((.(((........))).))))).)((((..(..((((....)))).....((((...(....)...))))..)..)))).......(((((((((....))))).)))). ( -38.40, z-score = -0.62, R) >droVir3.scaffold_13047 12397719 116 - 19223366 CGCGCCUUUGUUUAUCAUUAUGUGGGCGAGGGCCUCGAGGAGGGCAACUUCAACGAGGCAUCCGAGAAUAUAUGCCAGCUGGUGCACGAUUAUCUGGAGGUAGAUGCCUCGGCGCC .(((((.((((.(((((.((((((..(..((((((((..((((....))))..))))))..))..)..)))))).....))))).)))).......(((((....)))))))))). ( -46.70, z-score = -2.29, R) >droMoj3.scaffold_6540 31464943 116 - 34148556 CGAGCCUUCGUCUACCACUACGUGGGCGAGGGCCUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAACAUAUGCCAGCUUGUGCACGACUACCUCGAGGUGGACGCCUCGGCGCC ...((((((((((((......))))))))))))((((..((((....))))..))))((..(((((...........((....)).((.(((((....))))).)).))))).)). ( -50.80, z-score = -2.25, R) >droGri2.scaffold_14830 3565169 116 - 6267026 CGCGCCUUCGUCUAUCACUAUGUGGGCGAGGGUCUCGAGGAGGGCAACUUCAAUGAGGCAUCCGAGAAUAUAUGUCAGCUGGUCCACGAUUAUAUUGAGGUGGAUGCCUCGGCGCC (((((((((((((((......))))))))))))......((((....))))..(((((((((((..((((((.(((...........)))))))))....)))))))))))))).. ( -46.70, z-score = -2.39, R) >anoGam1.chr2R 1623976 114 + 62725911 CGUGCCUUCGUGCACUGGUACGUCGGAGAGGGUAUGGAGGAAGGUGAGUUCUCCGAGGCCCGUGAAGAUUUGGCCGCCCUCGAGAAGGACUACGAGGAGGUCGGAAUGGACUCC-- ((((((..........))))))..((((.((((.(((((((.......)))))))..)))).......(((((((..(((((((.....)).))))).))))))).....))))-- ( -43.80, z-score = -0.85, R) >apiMel3.Group15 6377742 103 - 7856270 AGAGCUUUUGUUCAUUGGUACGUUGGCGAAGGUAUGGAAGAAGGUGAAUUUUCUGAAGCUCGUGAAGAUCUUGCAGCUCUUGAAAAGGACUAUGAAGAGGUUG------------- .(((((((.(((((((.........((....)).........))))))).....))))))).....((((((.((..((((....))))...))..)))))).------------- ( -24.97, z-score = -0.22, R) >triCas2.ChLG1X 2817482 101 - 8109244 CGUGCUUUCGUCCACUGGUAUGUGGGUGAAGGUAUGGAAGAAGGUGAAUUCUCUGAAGCUCGUGAGGAUUUGGCUGCUCUUGAGAAGGACUACGAGGAGGU--------------- (((((((((..((((......))))..))))))))).....((.(((((((((((.....)).))))))))).)).(((((.((.....))...)))))..--------------- ( -33.60, z-score = -2.24, R) >consensus CGCGCCUUCGUCUACCACUAUGUGGGCGAGGGACUCGAGGAGGGCAACUUCAACGAGGCCUCCGAGAAUAUCUGCCAGCUGGUGCACGACUAUCUGGAGGUGGAUGCCUCGGCUCC ....(((((((((((......)))))))))))....(..(((......)))..).............................................................. (-13.79 = -12.24 + -1.55)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:24:10 2011