| Sequence ID | dm3.chr3R |
|---|---|
| Location | 13,708,402 – 13,708,544 |
| Length | 142 |
| Max. P | 0.995827 |

| Location | 13,708,402 – 13,708,544 |
|---|---|
| Length | 142 |
| Sequences | 11 |
| Columns | 157 |
| Reading direction | reverse |
| Mean pairwise identity | 77.90 |
| Shannon entropy | 0.45132 |
| G+C content | 0.51617 |
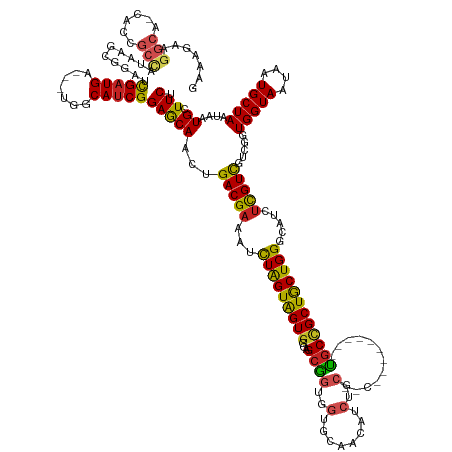
| Mean single sequence MFE | -56.04 |
| Consensus MFE | -25.62 |
| Energy contribution | -25.17 |
| Covariance contribution | -0.46 |
| Combinations/Pair | 1.30 |
| Mean z-score | -3.24 |
| Structure conservation index | 0.46 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.85 |
| SVM RNA-class probability | 0.995827 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 13708402 142 - 27905053 GAAAGAAGGCACCACCGCCAUUAGCGGAUCGAUGA---UGGCAUCGGAGCAGCUGACGAAAUCUAGUAGUGGGAGCGGUGGUGCAACAUCUGC------------UGCCGCUGCUGGGCAUCUUGUCGUCGGUGGUACUAAUGCUAAUAAUGCUUCU ...(((((.((...((((.....))))........---((((((..(.((.((((((((..(((((((((((.(((((((......)).))))------------).))))))))))).......)))))))).)).)..))))))....))))))) ( -59.70, z-score = -3.34, R) >droSec1.super_12 1677059 142 + 2123299 GAAAGAAGGCACCACCGCCAUUAGCGGAUCGAUGA---UGGCAUCGGAGCAGCUGACGAAAUCUAGUAGUGGGAGCGGUGGUGCAACAUCUGC------------UGCCGCUGCUGGGCAUCUUGUCGUCGGUGGUACUAAUGCUAAUAAUGCUUCU ...(((((.((...((((.....))))........---((((((..(.((.((((((((..(((((((((((.(((((((......)).))))------------).))))))))))).......)))))))).)).)..))))))....))))))) ( -59.70, z-score = -3.34, R) >droYak2.chr3R 2300652 142 - 28832112 GAAAGAAGGCACCACCGCCAUUAGCGGAUCGAUGA---UGGCAUCGGAGCAGCUGACGAAAUCUAGUAGUGGGAGCGGUGGUGCAACAUCUGC------------UGCCGCUGCUGGGCAUCUUGUCGUCGGUGGUACUAAUGCUAAUAAUGCUUCU ...(((((.((...((((.....))))........---((((((..(.((.((((((((..(((((((((((.(((((((......)).))))------------).))))))))))).......)))))))).)).)..))))))....))))))) ( -59.70, z-score = -3.34, R) >droEre2.scaffold_4770 9829730 142 + 17746568 GAAAGAAGGCACCACCGCCAUGAGCGGAUCGAUGA---UGGCAUCGGAGCAGCUGACGAAAUCUAGUAGUGGGAGCGGUGGUGCAACAUCUGC------------UGCCGCUGCUGGGCAUCUUGUCGUCGGUGGUACUAAUGCUAAUAAUGCUUCU ...(((((.((...((((.....))))........---((((((..(.((.((((((((..(((((((((((.(((((((......)).))))------------).))))))))))).......)))))))).)).)..))))))....))))))) ( -59.70, z-score = -3.18, R) >droAna3.scaffold_13340 11693787 130 + 23697760 ---------------GAAAGAAGGCGGAUCGAUGAUCGGAGCAUCGGAGCAGCUGACGAAAUCUAGUAGUGGGAGCGGUGGUGCA---------ACAUC---UGCUGCCGCUGCUGGGCAUCAUGUCGUCGGUGGUACUAAUGCUAAUAAUGCUUCU ---------------...(((((.(.((((...)))).)(((((..(.((.((((((((..(((((((((((.(((((((.....---------.)).)---)))).))))))))))).......)))))))).)).)..))))).......))))) ( -55.00, z-score = -3.89, R) >dp4.chr2 28707038 145 - 30794189 GAAAGAAGGCAUCAACGCUAUCGGCGGAACGAUGG---CAUCAUCGGAGCAACAGACGAAAUCUAGUAGUGGGAGCGGUGGUGCAACAUCUGCUGC---------CGCCGCUGCUGGCAAUGUCGUCCUCGGUGGUAAUAAUGCUAAUAAUGCUUCU ...(((((.(((....(((((((......))))))---)(((((((.((.....(((((...((((((((((..((((..(........)..))))---------..)))))))))).....)))))))))))))).............)))))))) ( -55.10, z-score = -2.65, R) >droPer1.super_6 4038987 145 - 6141320 GAAAGAAGGCAUCAACGCUAUCGGCGGAACGAUGG---CAUCAUCGGAGCAACAGACGAAAUCUAGUAGUGGGAGCGGUGGUGCAACAUCUGCUGC---------CGCCGCUGCUGGCAAUGUCGUCCUCGGUGGUAAUAAUGCUAAUAAUGCUUCU ...(((((.(((....(((((((......))))))---)(((((((.((.....(((((...((((((((((..((((..(........)..))))---------..)))))))))).....)))))))))))))).............)))))))) ( -55.10, z-score = -2.65, R) >droWil1.scaffold_181089 7403884 133 + 12369635 ---------------GAAAGAAGGCGGAACGAUG------GCAUCGGAACAACAGACGAAAAUUGGUAGUGGGAGCGGUGGUGCAGCAGCAACUGCAUC---CGCUGCUGCUGCUGGUCAUGUCGUCGUCGGUGGUAAUAAUGCUAAUAAUGCUUCU ---------------...(((((((.((.(((((------((((((..........)))..((..((((..(.(((((..((((((......)))))))---)))).)..))))..))..)))))))))).))((((....)))).......))))) ( -51.50, z-score = -3.22, R) >droMoj3.scaffold_6540 31257909 157 + 34148556 GAAAGAAGGCAUCAUCGCCGUAAACGGAUUGUUGGCAUCGGCAUCGGAACAAUUGACGAAAUCUAGUGGUGGGAGCGGUGGUGCAACAUCUGCCGCAGCUACCGCUGCCGCUACUGGUAAUAUCGUCUGCGAUGGUAAUAAUGCUAAUAAUGCUUCU ...(((((((......)))......(.(((((((((((.(.(((((.(......(((((...((((((((((.(((((((((((..........))).)))))))).)))))))))).....)))))).))))).)....))))))))))).))))) ( -63.80, z-score = -4.38, R) >droVir3.scaffold_13047 12218926 148 + 19223366 GAAAGAAGGC---AUCGCCAUGAACGGAUUGAUGGCAUCGGCAUCGGAGCAACUGACGAAAUCUAGUAGUGGGAGCGGUGGUGCAACAACUGCCGCAGC------UGCCGCUGCUGGUAAUAUCGUCUGCGAUGGUAAUAAUGCUAAUAAUGCUUCU ...(((((.(---((.(((((.((....)).)))))((..((((((..(((...(((((...((((((((((.(((.(((((.........))))).))------).)))))))))).....))))))))..))......))))..)).)))))))) ( -55.10, z-score = -2.60, R) >droGri2.scaffold_14830 3388239 133 + 6267026 GAAAGAAGGC---AUCGCUAUGAACGGAUUGAUGA---UGACAUCGGAACAUCUGACGAAAUCUAGUAGUGGGAGCAAUAUUGGUGC------------------UCCGGCUGCUGGUAAUAUCGUUGUCGAUGGUAAUAAUGCUAAUAAUGCUUCU ...(((((.(---(((((.((((.((((((((((.---...)))))....))))).......((((((((.((((((.......)))------------------))).)))))))).....)))).).)))))(((....)))........))))) ( -42.00, z-score = -3.03, R) >consensus GAAAGAAGGCA_CACCGCCAUAAGCGGAUCGAUGA___UGGCAUCGGAGCAACUGACGAAAUCUAGUAGUGGGAGCGGUGGUGCAACAUCUGC__C_________UGCCGCUGCUGGGCAUCUCGUCGUCGGUGGUAAUAAUGCUAAUAAUGCUUCU .......(((......)))..........(((((.......)))))(((((...(((((...((((((((((...................................)))))))))).....))))).....(((((....)))))....)).))). (-25.62 = -25.17 + -0.46)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:23:40 2011