| Sequence ID | dm3.chr3R |
|---|---|
| Location | 12,758,832 – 12,758,946 |
| Length | 114 |
| Max. P | 0.935835 |

| Location | 12,758,832 – 12,758,946 |
|---|---|
| Length | 114 |
| Sequences | 13 |
| Columns | 131 |
| Reading direction | forward |
| Mean pairwise identity | 92.68 |
| Shannon entropy | 0.16158 |
| G+C content | 0.67065 |
| Mean single sequence MFE | -57.04 |
| Consensus MFE | -47.86 |
| Energy contribution | -48.71 |
| Covariance contribution | 0.85 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.84 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.40 |
| SVM RNA-class probability | 0.935835 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
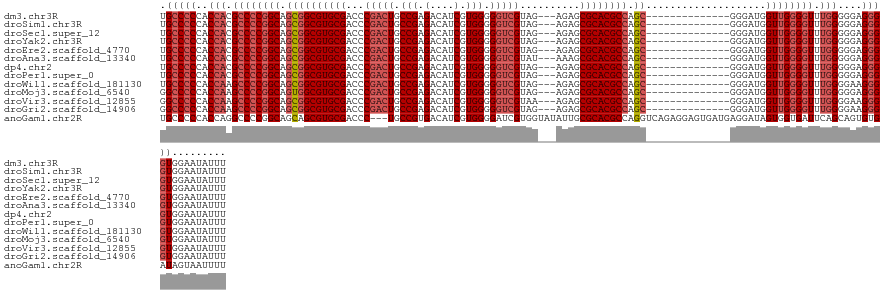
>dm3.chr3R 12758832 114 + 27905053 UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......)))))))).)))..).)))))......... ( -57.90, z-score = -2.22, R) >droSim1.chr3R 18803053 114 - 27517382 UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......)))))))).)))..).)))))......... ( -57.90, z-score = -2.22, R) >droSec1.super_12 726860 114 - 2123299 UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......)))))))).)))..).)))))......... ( -57.90, z-score = -2.22, R) >droYak2.chr3R 17202584 114 - 28832112 UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......)))))))).)))..).)))))......... ( -57.90, z-score = -2.22, R) >droEre2.scaffold_4770 8872595 114 - 17746568 UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......)))))))).)))..).)))))......... ( -57.90, z-score = -2.22, R) >droAna3.scaffold_13340 16557665 114 - 23697760 UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAU---AAAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.(((.((((((((.((((((((((...(((((.(((.(....).))).)))))...---....)))))))).))--------------......)))))))).)))..).)))))......... ( -57.90, z-score = -2.23, R) >dp4.chr2 17755912 114 + 30794189 UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......)))))))).)))..).)))))......... ( -57.90, z-score = -2.22, R) >droPer1.super_0 11611187 114 + 11822988 UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......)))))))).)))..).)))))......... ( -57.90, z-score = -2.22, R) >droWil1.scaffold_181130 11851604 114 - 16660200 UGCCCCCACCAAGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGAAGGGGUGGAAUAUUU .(((((..((((((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......))))))))))))....)))))......... ( -60.10, z-score = -3.37, R) >droMoj3.scaffold_6540 2041136 114 + 34148556 GGCCCCCACCAAGCCCCGGCAGUGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .((((((.((((((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..))))))))).)--------------......))))))))))))..).)))))......... ( -59.80, z-score = -3.12, R) >droVir3.scaffold_12855 9847157 114 - 10161210 GGCCCCCACCAAGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAA---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGAAGGGGUGGAAUAUUU .(((((..((((((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......))))))))))))....)))))......... ( -60.30, z-score = -3.26, R) >droGri2.scaffold_14906 12933550 114 + 14172833 GGCCCCCACCAAGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG---AGAGCGCACGCCAGC--------------GGGAUGGUUGGGGUUUGGGGAAGGGGUGGAAUAUUU .(((((..((((((((((((.((((((((((..((((((.(((.(....).))).)))))...---.)..)))))))).))--------------......))))))))))))....)))))......... ( -60.30, z-score = -3.30, R) >anoGam1.chr2R 62218031 128 + 62725911 UGCCCCCACCAGGCCCCGGCAGCAGCGUGCGACCC---UGCCGUGACAUCGUGGGGAUCGUGGUAUAUUGCGCACGCCAGGUCAGAGGAGUGAUGAGGAUAGUGGUGAUUCAGCAGUGUGAUAGUAAUUUU (((((((((...(.(.((((((...(....)...)---))))).).)...)))))).......((((((((...(((((.(((...(......)...)))..))))).....))))))))...)))..... ( -37.80, z-score = 1.52, R) >consensus UGCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAG___AGAGCGCACGCCAGC______________GGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAUAUUU .(((((..(((.((((((((.((((((((((...(((((.(((.(....).))).)))))..........)))))))).))....................)))))))).)))....)))))......... (-47.86 = -48.71 + 0.85)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:21:11 2011