
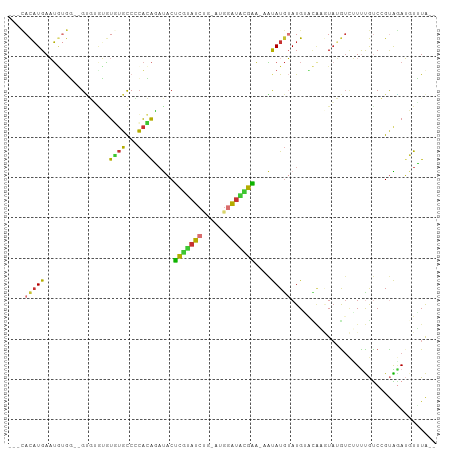
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 12,697,947 – 12,698,038 |
| Length | 91 |
| Max. P | 0.965810 |

| Location | 12,697,947 – 12,698,038 |
|---|---|
| Length | 91 |
| Sequences | 8 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 62.70 |
| Shannon entropy | 0.73269 |
| G+C content | 0.39504 |
| Mean single sequence MFE | -27.61 |
| Consensus MFE | -9.21 |
| Energy contribution | -8.10 |
| Covariance contribution | -1.11 |
| Combinations/Pair | 1.88 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.33 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.917172 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 12697947 91 + 27905053 ---CACAUGAAUAUGG--GUGGGUGUGUGCCCCACAGAUACUCGUAUCUG-AUGGAUACGAA-AAUAUGUAUG------UUUGUCUUUUGUCCGUAAAUGUUUA-- ---.((((...(((((--(((((.(....))))))((((..(((((((..-...)))))))(-((((....))------))))))).....))))).))))...-- ( -24.60, z-score = -1.44, R) >droSim1.chr3R 18742331 99 - 27517382 ---CACAUGAAUGUGGCGGUGGGUGUGUGCCCCACAGAUACUCGUAUCUG-AUGGAUACGAA-AAUAUGUAUGAACAAGUAUGUCUUUUGUCCGUAGAUGUUUA-- ---((((....))))...(((((.(....))))))......(((((((..-...)))))))(-(((((.((((.(((((.......))))).)))).)))))).-- ( -29.70, z-score = -1.85, R) >droSec1.super_12 666756 97 - 2123299 ---CACAUGAAUGUGG--GUGGGUGUGUGCCCCACAGAUACUCGUAUCUG-AUGGAUACGAA-AAUAUGUAUGAACAAGUAUGUCUUUUGUCCGUAGAUGUUUA-- ---((((....)))).--(((((.(....))))))......(((((((..-...)))))))(-(((((.((((.(((((.......))))).)))).)))))).-- ( -29.70, z-score = -2.20, R) >droYak2.chr3R 17139642 97 - 28832112 ---CACAUGAAUGCGG--GCGUGUGUUUGCCCCACAGAUACUCGUAUCUG-AUGGAUACGAA-AAUAUGUAUGUGCAAGUAUGUCUUUUGUCCGUAGAUGUUUA-- ---........((.((--(((......))))))).......(((((((..-...)))))))(-(((((.((((..((((.......))))..)))).)))))).-- ( -28.40, z-score = -1.84, R) >droEre2.scaffold_4770 8810642 97 - 17746568 ---CACAUGAAUGUGG--GUGUUUGUGUGCCGCACAGCUACUCGUAUCUG-AUGGAUACGAA-AAUAUGUAUGAGCAAGUAUGUCUUUUGUCCGUAGAUGUUUA-- ---((((....)))).--(((.(((((.....))))).)))(((((((..-...)))))))(-(((((.((((..((((.......))))..)))).)))))).-- ( -28.20, z-score = -1.74, R) >dp4.chr2 17688494 99 + 30794189 ---UAUAUGAUUAUACAUGCUUAGAUUUGCCACAGAUACCCAUGUAUC----UGUGUGGAUAUAGCAUGAAUGUAUGCAUAAGUUAUCCACCAGCGGAGCUGUGGA ---..((((..(((.((((((...(((..(.((((((((....)))))----))))..)))..)))))).)))....)))).....(((((.(((...)))))))) ( -34.20, z-score = -3.35, R) >droPer1.super_0 11543450 106 + 11822988 AGAUAUAUGAUUAUACAUGCUUGGAUUUGCCACAGAUACACAUCCAUCCACAUGGAUGGAUAUAGCAUGAAUGUAUGCAUAAGUUAUCCACCAGCGGAGCUGUGGA .(((.((((..(((.(((((((((.....))).........((((((((....))))))))..)))))).)))....)))).))).(((((.(((...)))))))) ( -32.70, z-score = -2.20, R) >droMoj3.scaffold_6540 1963352 92 + 34148556 ---AACUUGAAAUCGU--GAAUAAUUUACUCUGGAGUAUACUCUCAUCUG-----ACAAGGUUCUCAUAUAUGUAUAUUUAAA--GUUUGUCGGUGUAUAUAUA-- ---..((((.....((--((.....))))...((((....))))......-----.)))).......((((((((((((....--.......))))))))))))-- ( -13.40, z-score = 0.43, R) >consensus ___CACAUGAAUGUGG__GUGUGUGUGUGCCCCACAGAUACUCGUAUCUG_AUGGAUACGAA_AAUAUGUAUGUACAAGUAUGUCUUUUGUCCGUAGAUGUUUA__ ....(((((................((((...)))).....((((((((....))))))))....))))).................................... ( -9.21 = -8.10 + -1.11)


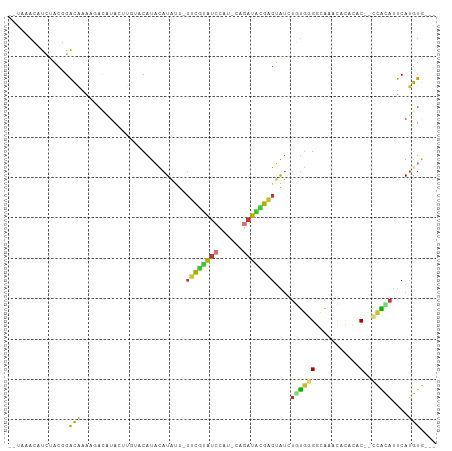
| Location | 12,697,947 – 12,698,038 |
|---|---|
| Length | 91 |
| Sequences | 8 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 62.70 |
| Shannon entropy | 0.73269 |
| G+C content | 0.39504 |
| Mean single sequence MFE | -23.26 |
| Consensus MFE | -6.75 |
| Energy contribution | -5.42 |
| Covariance contribution | -1.33 |
| Combinations/Pair | 2.00 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.29 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.76 |
| SVM RNA-class probability | 0.965810 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 12697947 91 - 27905053 --UAAACAUUUACGGACAAAAGACAAA------CAUACAUAUU-UUCGUAUCCAU-CAGAUACGAGUAUCUGUGGGGCACACACCCAC--CCAUAUUCAUGUG--- --.........................------..(((((...-...(((((...-..)))))((((((..(((((.......)))))--..)))))))))))--- ( -20.60, z-score = -2.72, R) >droSim1.chr3R 18742331 99 + 27517382 --UAAACAUCUACGGACAAAAGACAUACUUGUUCAUACAUAUU-UUCGUAUCCAU-CAGAUACGAGUAUCUGUGGGGCACACACCCACCGCCACAUUCAUGUG--- --...........((((((.........)))))).(((((...-.(((((((...-..)))))))((..(.(((((.......))))).)..))....)))))--- ( -23.10, z-score = -2.32, R) >droSec1.super_12 666756 97 + 2123299 --UAAACAUCUACGGACAAAAGACAUACUUGUUCAUACAUAUU-UUCGUAUCCAU-CAGAUACGAGUAUCUGUGGGGCACACACCCAC--CCACAUUCAUGUG--- --...........((((((.........)))))).(((((...-((((((((...-..))))))))....((((((...........)--)))))...)))))--- ( -23.30, z-score = -2.59, R) >droYak2.chr3R 17139642 97 + 28832112 --UAAACAUCUACGGACAAAAGACAUACUUGCACAUACAUAUU-UUCGUAUCCAU-CAGAUACGAGUAUCUGUGGGGCAAACACACGC--CCGCAUUCAUGUG--- --......((....))......((((...(((.((((.((((.-.(((((((...-..))))))))))).))))((((........))--)))))...)))).--- ( -27.00, z-score = -3.91, R) >droEre2.scaffold_4770 8810642 97 + 17746568 --UAAACAUCUACGGACAAAAGACAUACUUGCUCAUACAUAUU-UUCGUAUCCAU-CAGAUACGAGUAGCUGUGCGGCACACAAACAC--CCACAUUCAUGUG--- --......((....)).............((((..((((....-((((((((...-..))))))))....)))).)))).........--.((((....))))--- ( -17.00, z-score = -1.05, R) >dp4.chr2 17688494 99 - 30794189 UCCACAGCUCCGCUGGUGGAUAACUUAUGCAUACAUUCAUGCUAUAUCCACACA----GAUACAUGGGUAUCUGUGGCAAAUCUAAGCAUGUAUAAUCAUAUA--- ((((((((...))).)))))....((((((((..................((((----(((((....)))))))))((........)))))))))).......--- ( -28.40, z-score = -2.39, R) >droPer1.super_0 11543450 106 - 11822988 UCCACAGCUCCGCUGGUGGAUAACUUAUGCAUACAUUCAUGCUAUAUCCAUCCAUGUGGAUGGAUGUGUAUCUGUGGCAAAUCCAAGCAUGUAUAAUCAUAUAUCU ((((((((...))).)))))....((((((((.(((..((((.((((((((((....))))))))))))))..)))((........)))))))))).......... ( -35.90, z-score = -3.08, R) >droMoj3.scaffold_6540 1963352 92 - 34148556 --UAUAUAUACACCGACAAAC--UUUAAAUAUACAUAUAUGAGAACCUUGU-----CAGAUGAGAGUAUACUCCAGAGUAAAUUAUUC--ACGAUUUCAAGUU--- --............(((((..--(((..((((....))))..)))..))))-----)......(((((((((....))))...)))))--.............--- ( -10.80, z-score = -0.18, R) >consensus __UAAACAUCUACGGACAAAAGACAUACUUGUACAUACAUAUU_UUCGUAUCCAU_CAGAUACGAGUAUCUGUGGGGCAAACACACAC__CCACAUUCAUGUG___ ............................................((((((((......)))))))).........................((((....))))... ( -6.75 = -5.42 + -1.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:21:01 2011