| Sequence ID | dm3.chr3R |
|---|---|
| Location | 12,681,987 – 12,682,089 |
| Length | 102 |
| Max. P | 0.999470 |

| Location | 12,681,987 – 12,682,089 |
|---|---|
| Length | 102 |
| Sequences | 15 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 85.08 |
| Shannon entropy | 0.32720 |
| G+C content | 0.45499 |
| Mean single sequence MFE | -36.22 |
| Consensus MFE | -28.89 |
| Energy contribution | -28.61 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.45 |
| Structure conservation index | 0.80 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.48 |
| SVM RNA-class probability | 0.998753 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
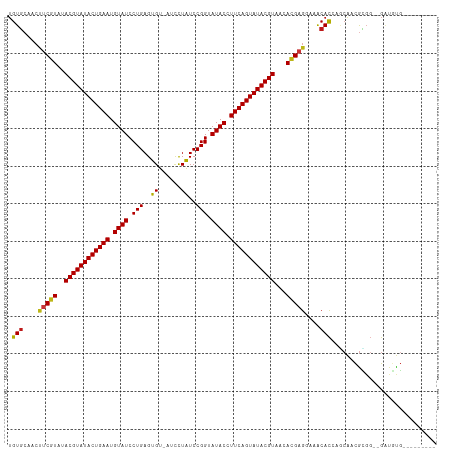
>dm3.chr3R 12681987 102 + 27905053 UGUGCGACCUCGUAAACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGAGAGCACUAACAACGUGG--GAUGUG--------- .((((...(((((..((((((((((((.((((.(((..((.-....))..))).)))).))))))))))))...)))))...))))...........--......--------- ( -35.80, z-score = -3.63, R) >droPer1.super_0 11526563 106 + 11822988 UGUGCGACUUCGUAUACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGGAAACACCAGCAACGCGG--GAUAUGUCGA----- ....((((.((((.(((((((((((((.((((.(((..((.-....))..))).)))).)))))))))))))..))))(....).((.((...)).)--)....)))).----- ( -35.90, z-score = -2.77, R) >dp4.chr2 17671757 106 + 30794189 UGUGCGACUUCGUAUACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGGAAACACCAGCAACGCGG--GAUACGUCGA----- ....((((.((((.(((((((((((((.((((.(((..((.-....))..))).)))).)))))))))))))..))))(....).((.((...)).)--)....)))).----- ( -36.20, z-score = -2.76, R) >droAna3.scaffold_13340 16482481 102 - 23697760 UGUGCAACUUCGUAUACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGGUAACACCUUCAACGCGG--GACGUG--------- .(((..(((((((.(((((((((((((.((((.(((..((.-....))..))).)))).)))))))))))))..)))))))..)))......((((.--..))))--------- ( -36.20, z-score = -3.59, R) >droEre2.scaffold_4770 8794075 102 - 17746568 UGUGCGACCUCGUAAACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGAAAGCACGAGCAACGUGG--GAUGUG--------- .((((...(((((..((((((((((((.((((.(((..((.-....))..))).)))).))))))))))))...)))))...))))...........--......--------- ( -36.30, z-score = -3.77, R) >droYak2.chr3R 17123068 102 - 28832112 UGUGCGACCUCGUAAACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGAAAGCACGAGCAACGUGG--GAUGUG--------- .((((...(((((..((((((((((((.((((.(((..((.-....))..))).)))).))))))))))))...)))))...))))...........--......--------- ( -36.30, z-score = -3.77, R) >droSec1.super_12 650978 102 - 2123299 UGUGCGACCUCGUAAACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGAUAGCACGAGCAACGUGG--GAUGUG--------- .((((...(((((..((((((((((((.((((.(((..((.-....))..))).)))).))))))))))))...)))))...))))...........--......--------- ( -36.30, z-score = -3.44, R) >droSim1.chr3R 18726089 102 - 27517382 UGUGCGACCUCGUAAACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGAUAGCACGAGCAACGUGG--GAUGUG--------- .((((...(((((..((((((((((((.((((.(((..((.-....))..))).)))).))))))))))))...)))))...))))...........--......--------- ( -36.30, z-score = -3.44, R) >anoGam1.chr2R 62093365 109 + 62725911 AGUGCCGCUUCAUGAACGUAUACUGAAUGUAUCCUGAGUGC-UACUUAUCCGGUAUACCUUCAGUAUACGUAACAAGAGGCGACACAAUCAUCGUUGAUGUCGUGAUAUG---- .(((.((((((.((.((((((((((((.((((((((((...-..))))...)).)))).))))))))))))..)).)))))).))).((((.((.......))))))...---- ( -39.60, z-score = -4.47, R) >droGri2.scaffold_14906 12815109 102 + 14172833 UGUGCAACUUCGUAUACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGGAAACACCAGCAACGCGG--GAUGUG--------- .......((((((.(((((((((((((.((((.(((..((.-....))..))).)))).)))))))))))))..))))))..(((((.((...)).)--).))).--------- ( -35.10, z-score = -3.29, R) >droMoj3.scaffold_6540 1943734 102 + 34148556 UGUGCAACUUCGUAUACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGGAAACACCAGCAACGCGG--GAUGUG--------- .......((((((.(((((((((((((.((((.(((..((.-....))..))).)))).)))))))))))))..))))))..(((((.((...)).)--).))).--------- ( -35.10, z-score = -3.29, R) >droVir3.scaffold_12855 9746428 102 - 10161210 UGUGCAACUUCGUAUACGUAUACUGAAUGUAUCCUGAGUGU-AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGGAAACACCAGCGACGCGG--GAUGUG--------- .......((((((.(((((((((((((.((((.(((..((.-....))..))).)))).)))))))))))))..))))))..(((((.((...)).)--).))).--------- ( -35.10, z-score = -3.00, R) >droWil1.scaffold_181130 11754524 102 - 16660200 UGUGCAACUUCGUAUACGUAUACUGAAUGUAUCCUGAGUGU-AUGCUAUCCGGUAUACCUUCAGUAUACGUAACACGAUGAAACACCAACAACGCGG--GAUGUG--------- .(((.....((((.(((((((((((((.((((((..(((..-..)))....)).)))).)))))))))))))..)))).....)))...........--......--------- ( -29.90, z-score = -1.99, R) >apiMel3.Group16 3880564 106 + 5631066 UGUGAAACCCCCUGUACGUAUACUGAAUGUAUCCUGAGUGUAUUUCUGUCCGGUAUACCUUCAGUAUACGUAACAGGAGGCUACACC-GAACUACGC-UGUCAUCAUG------ .((((..((.(((((((((((((((((.((((((.((..(.....)..)).)).)))).)))))))))))).))))).))..(((.(-(.....)).-)))..)))).------ ( -36.30, z-score = -4.16, R) >triCas2.ChLG2 10434522 111 - 12900155 UGUUAAACCCCCUGUACGUAUACUGAAUGUAUCCUGAGUGU-UUUCUGUCCGGUAUACCUUCAGUAUACGUAACAGGAGGCCACAUU-UCAACGGUC-CGGCCUCAGGGACUCC .......(((.((((((((((((((((.((((((.((..(.-...)..)).)).)))).)))))))))))).))))((((((.....-.........-.)))))).)))..... ( -42.86, z-score = -4.45, R) >consensus UGUGCAACUUCGUAUACGUAUACUGAAUGUAUCCUGAGUGU_AUCCUAUCCGGUAUACCUUCAGUAUACGUAACACGAGGAAACACCAGCAACGCGG__GAUGUG_________ .(((....(((((..((((((((((((.((((.(((..((......))..))).)))).))))))))))))...)))))....)))............................ (-28.89 = -28.61 + -0.27)


| Location | 12,681,987 – 12,682,089 |
|---|---|
| Length | 102 |
| Sequences | 15 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 85.08 |
| Shannon entropy | 0.32720 |
| G+C content | 0.45499 |
| Mean single sequence MFE | -38.97 |
| Consensus MFE | -31.08 |
| Energy contribution | -30.59 |
| Covariance contribution | -0.49 |
| Combinations/Pair | 1.16 |
| Mean z-score | -4.20 |
| Structure conservation index | 0.80 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.92 |
| SVM RNA-class probability | 0.999470 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
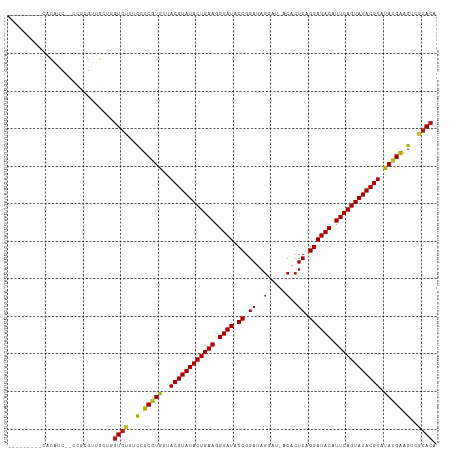
>dm3.chr3R 12681987 102 - 27905053 ---------CACAUC--CCACGUUGUUAGUGCUCUCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUUUACGAGGUCGCACA ---------......--...........((((..(((((((..((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))).)))))))..)))). ( -38.80, z-score = -4.60, R) >droPer1.super_0 11526563 106 - 11822988 -----UCGACAUAUC--CCGCGUUGCUGGUGUUUCCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUAUACGAAGUCGCACA -----.((((.....--....))))...((((..(.((((((.((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))))))))).)..)))). ( -35.80, z-score = -2.65, R) >dp4.chr2 17671757 106 - 30794189 -----UCGACGUAUC--CCGCGUUGCUGGUGUUUCCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUAUACGAAGUCGCACA -----.((((((...--..))))))...((((..(.((((((.((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))))))))).)..)))). ( -40.30, z-score = -3.62, R) >droAna3.scaffold_13340 16482481 102 + 23697760 ---------CACGUC--CCGCGUUGAAGGUGUUACCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUAUACGAAGUUGCACA ---------.(((..--...))).....((((.((.((((((.((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))))))))).)).)))). ( -38.00, z-score = -3.78, R) >droEre2.scaffold_4770 8794075 102 + 17746568 ---------CACAUC--CCACGUUGCUCGUGCUUUCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUUUACGAGGUCGCACA ---------......--...........((((..(((((((..((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))).)))))))..)))). ( -39.70, z-score = -5.13, R) >droYak2.chr3R 17123068 102 + 28832112 ---------CACAUC--CCACGUUGCUCGUGCUUUCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUUUACGAGGUCGCACA ---------......--...........((((..(((((((..((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))).)))))))..)))). ( -39.70, z-score = -5.13, R) >droSec1.super_12 650978 102 + 2123299 ---------CACAUC--CCACGUUGCUCGUGCUAUCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUUUACGAGGUCGCACA ---------......--...........((((.((((((((..((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))).)))))))).)))). ( -41.00, z-score = -5.30, R) >droSim1.chr3R 18726089 102 + 27517382 ---------CACAUC--CCACGUUGCUCGUGCUAUCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUUUACGAGGUCGCACA ---------......--...........((((.((((((((..((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))).)))))))).)))). ( -41.00, z-score = -5.30, R) >anoGam1.chr2R 62093365 109 - 62725911 ----CAUAUCACGACAUCAACGAUGAUUGUGUCGCCUCUUGUUACGUAUACUGAAGGUAUACCGGAUAAGUA-GCACUCAGGAUACAUUCAGUAUACGUUCAUGAAGCGGCACU ----..........((((...))))...(((((((.((.((..((((((((((((.((((.((.((......-....)).)))))).)))))))))))).)).)).))))))). ( -38.50, z-score = -4.40, R) >droGri2.scaffold_14906 12815109 102 - 14172833 ---------CACAUC--CCGCGUUGCUGGUGUUUCCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUAUACGAAGUUGCACA ---------......--...........((((..(.((((((.((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))))))))).)..)))). ( -36.10, z-score = -3.32, R) >droMoj3.scaffold_6540 1943734 102 - 34148556 ---------CACAUC--CCGCGUUGCUGGUGUUUCCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUAUACGAAGUUGCACA ---------......--...........((((..(.((((((.((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))))))))).)..)))). ( -36.10, z-score = -3.32, R) >droVir3.scaffold_12855 9746428 102 + 10161210 ---------CACAUC--CCGCGUCGCUGGUGUUUCCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU-ACACUCAGGAUACAUUCAGUAUACGUAUACGAAGUUGCACA ---------......--...........((((..(.((((((.((((((((((((.((((.((.((..(...-.)..)).)))))).)))))))))))))))))).)..)))). ( -36.10, z-score = -3.30, R) >droWil1.scaffold_181130 11754524 102 + 16660200 ---------CACAUC--CCGCGUUGUUGGUGUUUCAUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGCAU-ACACUCAGGAUACAUUCAGUAUACGUAUACGAAGUUGCACA ---------......--...........((((..(.((((((.((((((((((((.((((.((.((......-....)).)))))).)))))))))))))))))).)..)))). ( -35.40, z-score = -3.62, R) >apiMel3.Group16 3880564 106 - 5631066 ------CAUGAUGACA-GCGUAGUUC-GGUGUAGCCUCCUGUUACGUAUACUGAAGGUAUACCGGACAGAAAUACACUCAGGAUACAUUCAGUAUACGUACAGGGGGUUUCACA ------..........-.........-.(((.((((((((((.((((((((((((.((((.((.((...........)).)))))).)))))))))))))))))))))).))). ( -43.30, z-score = -5.45, R) >triCas2.ChLG2 10434522 111 + 12900155 GGAGUCCCUGAGGCCG-GACCGUUGA-AAUGUGGCCUCCUGUUACGUAUACUGAAGGUAUACCGGACAGAAA-ACACUCAGGAUACAUUCAGUAUACGUACAGGGGGUUUAACA .((..(((.(((((((-.(.......-..).)))))))((((.((((((((((((.((((.((.((......-....)).)))))).)))))))))))))))))))..)).... ( -44.80, z-score = -4.03, R) >consensus _________CACAUC__CCGCGUUGCUGGUGUUUCCUCGUGUUACGUAUACUGAAGGUAUACCGGAUAGGAU_ACACUCAGGAUACAUUCAGUAUACGUAUACGAAGUCGCACA ............................((((..(.(((((..((((((((((((.((((.((.((..(.....)..)).)))))).)))))))))))).))))).)..)))). (-31.08 = -30.59 + -0.49)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:20:58 2011