
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 7,655,117 – 7,655,234 |
| Length | 117 |
| Max. P | 0.521545 |

| Location | 7,655,117 – 7,655,234 |
|---|---|
| Length | 117 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.14 |
| Shannon entropy | 0.12318 |
| G+C content | 0.52011 |
| Mean single sequence MFE | -40.79 |
| Consensus MFE | -34.89 |
| Energy contribution | -34.62 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.86 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.521545 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 7655117 117 - 23011544 CUUCUAUUUGCAGCAAUCGGUCGGCGAGCUCGGAGCAAGAUAAC---UCAGAUCUAUCCGAGCACUCGGAGAAAUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU .......((((.((((((((.((((..(((((((...((((...---....)))).))))))).....((....)))))))))))).))))))((((((.....)))))).......... ( -42.70, z-score = -1.75, R) >droSim1.chr2L 7452472 117 - 22036055 CUUCUAUUUGCAGCAAUCGGUCGGCGAGCUCGGAGCAAGAUAAC---UCAGAUCUAUCCGAGCACUCGGAGAAAUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU .......((((.((((((((.((((..(((((((...((((...---....)))).))))))).....((....)))))))))))).))))))((((((.....)))))).......... ( -42.70, z-score = -1.75, R) >droSec1.super_3 3169133 117 - 7220098 CUUCUAUUUGCAGCAAUCGGUCGGCGAGCUCGGAGCAAGAUAAC---UCAGAUCUAUCCGAGCACUCGGAGAAAUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU .......((((.((((((((.((((..(((((((...((((...---....)))).))))))).....((....)))))))))))).))))))((((((.....)))))).......... ( -42.70, z-score = -1.75, R) >droYak2.chr2L 17086191 117 + 22324452 CUUCUAUUUGCAGCAAUCGGUCGGCGAGCUCGGAGCAAGAUAAC---UCAGAUCUAUCCGAGCACUCGGAGAAAUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU .......((((.((((((((.((((..(((((((...((((...---....)))).))))))).....((....)))))))))))).))))))((((((.....)))))).......... ( -42.70, z-score = -1.75, R) >droEre2.scaffold_4929 16575531 117 - 26641161 CUUCUAUUUGCAGCAAUCGGUCGGCGAGCUCGGAGCAAGAUAAC---UCAGAUCUGUCCGAGCACUCGGAGAAAUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU .......((((.((((((((.((((..(((((((...((((...---....)))).))))))).....((....)))))))))))).))))))((((((.....)))))).......... ( -42.70, z-score = -1.45, R) >droAna3.scaffold_12916 15599506 117 - 16180835 CUUCUAUUUGCAGCAAUCGGUCGGCGAGCUCGGAGCAAGAUAAC---UCAGAUCAGUCCGAGCAUUCGGAGAAAUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU .......((((.((((((((.(((((((((((((....(((...---....)))..))))))).(((...))).)))))))))))).))))))((((((.....)))))).......... ( -39.90, z-score = -0.99, R) >dp4.chr4_group3 1550304 120 - 11692001 CUUCUAUUUGCAGCAAUCGGUCGGCCAGCUCGGAGCAAGAUAACAUGUCCGACAUGUCCGAGCACUCGGAGAAGUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU ...........(((..(((((..((((((..(((.((........)))))(((.(.(((((....))))).).)))...))))))..)).)))((((((.....))))))..)))..... ( -41.20, z-score = -0.91, R) >droPer1.super_1 3018307 120 - 10282868 CUUCUAUUUGCAGCAAUCGGUCGGCCAGCUCGGAGCAAGAUAACAUGUCCGACAUGUCCGAGCACUCGGAGAAGUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU ...........(((..(((((..((((((..(((.((........)))))(((.(.(((((....))))).).)))...))))))..)).)))((((((.....))))))..)))..... ( -41.20, z-score = -0.91, R) >droWil1.scaffold_180703 661106 116 + 3946847 -UUCUAUUUACAGCAAUCGCUCGGCCAGUUCGGAGCAAGAUAAC---UCAGAUCUAUCCGAGCACUCGGAGAAAUCGCCGCUGGUUAGUGCGAGGUUAGACAAUCUUGCUAGGCUCUUUU -.((((....((((....((((((..((.((.(((........)---)).)).))..))))))....((.(....).))))))......((((((((....))))))))))))....... ( -34.10, z-score = -0.76, R) >droVir3.scaffold_13246 2169602 117 + 2672878 CUUCUAUUUGCAGCAACCGGUCGGCCAGCUCGGAGCAAGAUAAC---UCAGAUUUGUCCGAGCACUCGGAGAAAUCGCCACUGGUUAGCGCGAGGUUAGACAAUCUUGCUAGGCUCUUUU .........((.(((((((((.(((.......(((........)---)).(((((.(((((....))))).))))))))))))))).))((((((((....))))))))...))...... ( -42.50, z-score = -2.49, R) >droGri2.scaffold_15252 16748441 117 - 17193109 AUUCUAUUUGCAGCAACCGGUCGGCCAGCUCGGAGCAAGAUAAC---UCAGAUCAGUCCGAACACUCGGAGAAAUCGCCACUGGUUAGCGCGAGGUUAGACAAUCUGGCUAGGCUCUUUU .........((.(((((((((.(((.......(((........)---)).(((...(((((....)))))...))))))))))))).))))(((.((((.(.....).)))).))).... ( -34.90, z-score = -0.66, R) >droMoj3.scaffold_6500 31887736 117 + 32352404 CUUCUAUUUGCAGCAACCGGUCGGCCAGCUCGGAGCAAGAUAAC---UCAGAUCAAUCCGAGCACUCGGAGAAAUCGCCACUGGUUAGCGCGAGGUUAGACAAUCUUGCUAGGCUCUUUU .........((.(((((((((.(((..(((((((....(((...---....)))..))))))).....((....)))))))))))).))((((((((....))))))))...))...... ( -42.20, z-score = -2.77, R) >consensus CUUCUAUUUGCAGCAAUCGGUCGGCCAGCUCGGAGCAAGAUAAC___UCAGAUCUAUCCGAGCACUCGGAGAAAUCGCCGCUGGUUAGCGCGAGGUUAGACAAUCUAGCUAGGCUCUUCU .........((.(((((((((.(((..(((((((......................))))))).....((....)))))))))))).))))(((.((((.(......))))).))).... (-34.89 = -34.62 + -0.27)

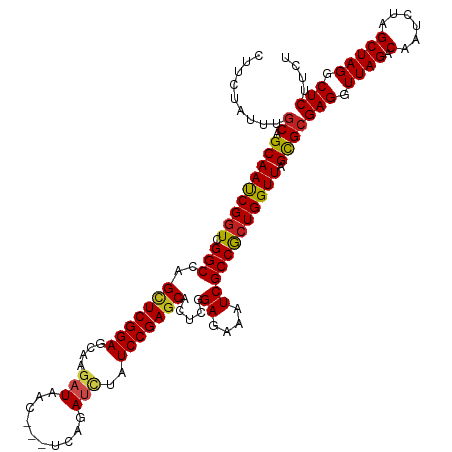
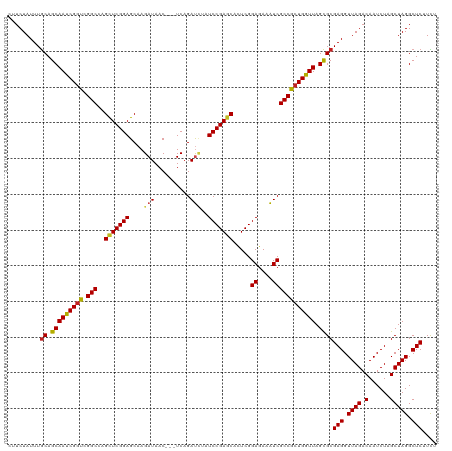
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:24:08 2011