
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 11,241,052 – 11,241,157 |
| Length | 105 |
| Max. P | 0.630940 |

| Location | 11,241,052 – 11,241,157 |
|---|---|
| Length | 105 |
| Sequences | 13 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 75.61 |
| Shannon entropy | 0.53254 |
| G+C content | 0.67907 |
| Mean single sequence MFE | -49.10 |
| Consensus MFE | -21.89 |
| Energy contribution | -21.49 |
| Covariance contribution | -0.39 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.45 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.630940 |
| Prediction | RNA |
| WARNING | Out of training range. z-scores are NOT reliable. |
Download alignment: ClustalW | MAF
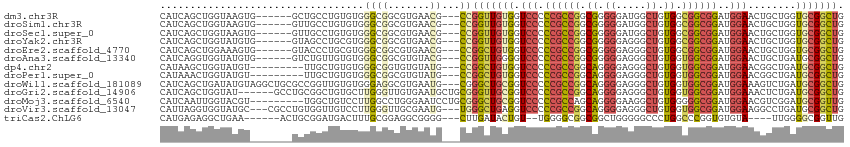
>dm3.chr3R 11241052 105 - 27905053 CAUCAGCUGGUAAGUG------GCUGCCUGUGUGGGCGGCGUGAACG---CCGGUUGUGGUCCCCCGCCGGCGGGGGAUGGCUGUGGCGGCGGAUGGAACUGCUGGUGCGGCUG ((.((((((((.....------(((((((....)))))))(....))---)))))))))((((((((....))))))))(((((((.((((((......)))))).))))))). ( -60.00, z-score = -3.90, R) >droSim1.chr3R 17277304 105 + 27517382 CAUCAGCUGGUAAGUG------GUUGCCUGUGUGGGCGGCGUGAACG---CCGGUUGUGGUCCCCCGCCGGCGGGGGAUGGCUGUGGCGGCGGAUGGAACUGCUGGUGCGGCUG ((.((((((((.....------(((((((....)))))))(....))---)))))))))((((((((....))))))))(((((((.((((((......)))))).))))))). ( -57.50, z-score = -3.34, R) >droSec1.super_0 10413291 105 + 21120651 CAUCAGCUGGUAAGUG------GUUGCCUGUGUGGGCGGCGUGAACG---CCGGUUGUGGUCCCCCGCCGGCGGGGGAUGGCUGUGGCGGCGGAUGGAACUGCUGGUGCGGCUG ((.((((((((.....------(((((((....)))))))(....))---)))))))))((((((((....))))))))(((((((.((((((......)))))).))))))). ( -57.50, z-score = -3.34, R) >droYak2.chr3R 13641261 105 + 28832112 CAUCAGCUGGUAUGUG------GUAGCCUGCGUGGGCGGCGUGAACG---CCGGUUGUGGUCCCCCGCCGGCGGGGGAGGGCUGUGGCGGCGGAUGGAACUGCUGGUGCGGCUG ...((((((.(((((.------.((((((.((..(.(((((....))---))).)..)).(((((((....)))))))))))))..))(((((......))))).))))))))) ( -55.80, z-score = -2.27, R) >droEre2.scaffold_4770 10767164 105 - 17746568 CAUCAGCUGGAAAGUG------GUACCCUGCGUGGGCGGCGUGAACG---CCGGCUGUGGUCCCCCGCCGGCGGGGGAGGGCUGUGGCGGCGGAUGGAACUGCUGGUGCGGCUG ((.(((((((...((.------.(((.((((....)))).))).)).---))))))))).(((((((....))))))).(((((((.((((((......)))))).))))))). ( -55.80, z-score = -2.00, R) >droAna3.scaffold_13340 12825350 105 - 23697760 CAUCAGGUGGUAUGUG------GUCUGUUGUGUGGGCGGCGUGUACG---CCGGUUGGGGUCCCCCGCCGGCGGGGGAGGGCUGUGGUGGCGGAUGGAACUGCUGAUGCGGCUG (((((.(..((...(.------((((((..(....(((((.(...((---(((((.(((....)))))))))).....).))))).)..)))))).).))..))))))...... ( -45.40, z-score = -0.62, R) >dp4.chr2 20967016 102 + 30794189 CAUAAGCUGGUAUGU---------UUGCUGUGUGGGCGGUGUGUAUG---CCGGCUGUGGUCCCCCGCCGGCAGGGGAGGGCUGUGGUGGCGGAUGGAACGGCUGAUGCGGCUG ((((.(((((((((.---------.((((((....))))))..))))---))))))))).(((.((((((.((.((.....)).)).))))))..))).((((((...)))))) ( -46.10, z-score = -1.69, R) >droPer1.super_0 10657912 102 - 11822988 CAUAAACUGGUAUGU---------UUGCUGUGUGGGCGGCGUGUAUG---CCGGCUGUGGUCCCCCGCCGGCAGGGGAGGGCUGUGGUGGCGGAUGGAACGGCUGAUGCGGCUG ((((..((((((((.---------.((((((....))))))..))))---)))).)))).(((.((((((.((.((.....)).)).))))))..))).((((((...)))))) ( -43.90, z-score = -0.86, R) >droWil1.scaffold_181089 2140852 111 - 12369635 CAUCAGCUGAUAUGUAGGCUGCGCCGGUUGUGUGGGAGGCGUGAAUG---CGGGCUGCGGUCCCCCGCCGGCAGGGGAGGGCUGUGGUGGCGGAUGGAAAGUCUGAUGCGGCUG ...((((((.(((.(((((((((((............)))))...((---(.(.(..((((((.((.((....)))).))))))..)).))).......))))))))))))))) ( -44.80, z-score = 0.23, R) >droGri2.scaffold_14906 4355375 108 - 14172833 CAUCAGCUGGUAU------GCCUGCGGCUGUGCUUGGGUUGUGAAUGCUGCGGGUUGCGGUCCCCCGCCGGCAGGGGAGGGCUGUGGUGGCGGAUGGAAACUCUGAUGCGGCUG ...(((((((((.------(((((((((..(((.......)))...))))))))))))..(((.((((((.((.((.....)).)).))))))..)))..........)))))) ( -46.00, z-score = -0.33, R) >droMoj3.scaffold_6540 23577964 105 + 34148556 CAUCAAUUGGUACGU---------UGGCUGUCCUUGGCCUGGGAAUCCUGCGGGCUGCGGUCCCCCGCCAGCAGGGGAAGGCUGUGGGGGCGGAUGGAACGUCGGAUGCGGUUG .(((.(((.(.((((---------(..(((((((((((((.....((((((.(((.(.(....)).))).))))))..)))))..))))))))....)))))).)))..))).. ( -41.20, z-score = 0.78, R) >droVir3.scaffold_13047 5885481 108 + 19223366 CAUUAGGUGGUAUGC---CGCCUGUGGUUGUCCUUGGGUUGCGAAUG---UGGGCUGAGGUCCCCCGCCGGCAGGGGAGGGCUGUGGUGGCGGAUGGAAGGCCUGAUGCGGCUG (((.((((((....)---))))))))..........(((((((....---((((((....(((.((((((.((.((.....)).)).))))))..))).)))))).))))))). ( -46.30, z-score = -0.74, R) >triCas2.ChLG6 1266497 99 + 13544221 CAUGAGAGGCUGAA------ACUGCGGAUGACUUUGCGGAGGCGGGG---CUUGAUACUGU--UGGGGCGGCGGCUGGGGGCCCUGGCCCGGUGUGUA----UUGGGGCGGUUG .............(------(((((......((((((....))))))---((..(((((..--((((.(((.(((.....)))))).))))..).)))----)..)))))))). ( -38.00, z-score = -1.02, R) >consensus CAUCAGCUGGUAUGU_______GUUGGCUGUGUGGGCGGCGUGAACG___CCGGCUGUGGUCCCCCGCCGGCGGGGGAGGGCUGUGGCGGCGGAUGGAACUGCUGAUGCGGCUG ....................................................(((((((.(((.((((((.((.((.....)).)).))))))..)))........))))))). (-21.89 = -21.49 + -0.39)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:17:17 2011