
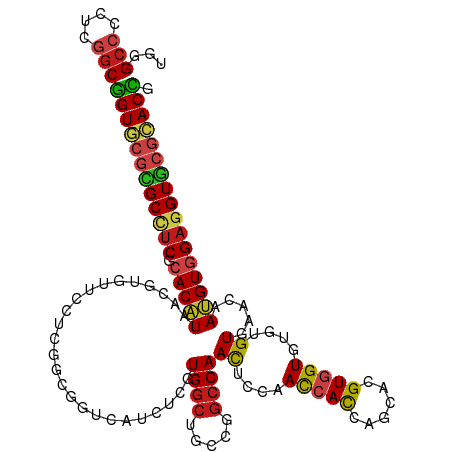
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 11,213,733 – 11,213,853 |
| Length | 120 |
| Max. P | 0.976545 |

| Location | 11,213,733 – 11,213,853 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.40 |
| Shannon entropy | 0.32007 |
| G+C content | 0.61923 |
| Mean single sequence MFE | -53.50 |
| Consensus MFE | -39.54 |
| Energy contribution | -39.72 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.74 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.95 |
| SVM RNA-class probability | 0.976545 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 11213733 120 + 27905053 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACUUGUUCCUCGGCGGUCAUCUCGUGGCUGCCGGCCAACUCGAACCACCAGCACGUGGUGUGUGUAAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((.....(((..(((((((((((...)))))))))))..)))....(((((((.....))))).)).......))))))))))))))))). ( -60.80, z-score = -3.67, R) >droWil1.scaffold_181089 2106955 120 + 12369635 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACGUGUUCCUCAGCGGUCAUCUCGUGGCUGCCGGCCAAUUCCAACCACCAGCACGUGGUGUGUGUGAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((....((((((.((((((((((...))))))))............(((((......))))).)).).)))))))))))))))))))))). ( -57.00, z-score = -2.39, R) >droMoj3.scaffold_6540 23547425 120 - 34148556 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACGUGUUCCUCGGCGGUUAUCUCAUGGCUGCCAGCCAACUCCAACCACCAGCACGUGGUGUGUGUGAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((....(((((...(((((((((...))))))))).....((....(((((......)))))....)))))))))))))))))))))))). ( -58.30, z-score = -3.03, R) >droVir3.scaffold_13047 5857523 120 - 19223366 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACGUGUUCCUCGGCGGUUAUCUCGUGGCUGCCGGCCAACUCCAACCACCAGCACGUGGUGUGUGUGAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((....(((((.(((((((((((...)))))))))))...((....(((((......)))))....)))))))))))))))))))))))). ( -60.60, z-score = -3.00, R) >anoGam1.chr2R 44660503 120 + 62725911 UAGGACCAUCGGCUGUAAGGGCUACACACAUUACCUGCUCCAGUGCCGUCAUCUCCUGGCUACCGGCCAACUCCAACCAUCAGCACGUCGUCUGCGUGAACAACGUGGAGGUCAGAACGG ...((((...(((((...((((..............)).)).((((((........)))).)))))))...((((........(((((.....))))).......))))))))....... ( -32.80, z-score = 0.86, R) >droPer1.super_0 10628458 120 + 11822988 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACGUGUUCCUCGGCGGUCAUCUCGUGGCAGCCGGCCAAUUCCAACCACCAACACGUGGUGUGUGUGAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((....(((((.(((((.(((((...))))).))))).......((..(((((.....)))))..)).)))))))))))))))))))))). ( -58.70, z-score = -2.78, R) >dp4.chr2 20937724 120 - 30794189 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACGUGUUCCUCGGCGGUCAUCUCGUGGCAGCCGGCCAAUUCCAACCACCAGCACGUGGUGUGUGUGAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((....(((((.(((((.(((((...))))).))))).......((..(((((.....)))))..)).)))))))))))))))))))))). ( -58.60, z-score = -2.47, R) >droGri2.scaffold_14906 4324261 120 + 14172833 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACGUGUUCCUCGGCGGUUAUCUCGUGGCUGCCGGCCAAUUCCAACCACCAGCAUGUGGUGUGUGUGAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((....(((((.(((((((((((...))))))))))).......((..(((((.....)))))..)).)))))))))))))))))))))). ( -60.40, z-score = -3.27, R) >droEre2.scaffold_4770 10739701 120 + 17746568 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACUUGUUCCUCGGCGGUCAUCUCGUGGCUGCCGGCCAACUCGAACCACCAGCACGUGGUGUGUGUAAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((.....(((..(((((((((((...)))))))))))..)))....(((((((.....))))).)).......))))))))))))))))). ( -60.80, z-score = -3.67, R) >droYak2.chr3R 13613887 120 - 28832112 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACUUGUUCCUCGGCGGUCAUCUCGUGGCUGCCGGCCAACUCGAACCACCAGCACGUGGUGUGUGUAAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((.....(((..(((((((((((...)))))))))))..)))....(((((((.....))))).)).......))))))))))))))))). ( -60.80, z-score = -3.67, R) >droAna3.scaffold_13340 12798186 120 + 23697760 UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACGUGUUCCUCGGCGGUCAUCUCGUGGCUGCCGGCCAACUCCAACCACCAGCAUGUGGUGUGUGUGAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((....(((((.(((((((((((...)))))))))))...((....(((((......)))))....)))))))))))))))))))))))). ( -62.60, z-score = -3.52, R) >apiMel3.GroupUn 354805676 120 - 399230636 UGGGCCCAACCGCUGUCAAGGCAUCGAACGUAACCGCCACUAGUGCAGUCAUAUCUUGGAUGCCGAGCAAUAGCAAUCAUCAACACGUGGUUUGUGUGAAUAACGUGGAGGUUAGGACGG ....((.(((((((((...((((((.((.(((((.(((....).)).))..))).)).)))))).....))))).........(((((.((((....)))).)))))..)))).)).... ( -33.90, z-score = 0.34, R) >triCas2.ChLG6 1306938 120 - 13544221 UGGGCCCCAGCGCAGUCCGAGCCUCUAACAUAACCUCAACAUCGGCUGUCAUCAGUUGGCUACCAGCCAACUCGAACCACCAACACGUCGUAUGUGUCAACAAUGUGGAAGUACGAACUG ...((....)).((((.((.((.((((.(((.......(((((((((((..(((((((((.....))))))).)).......))).)))).)))).......))))))).)).)).)))) ( -30.14, z-score = -0.26, R) >consensus UGGGCCCCUCGGCGGUGCGCGCCUCGCACAUAACGUGUUCCUCGGCGGUCAUCUCGUGGCUGCCGGCCAACUCCAACCACCAGCACGUGGUGUGUGUGAACAAUGUGGAGGUGCGCACCG ...(((....)))((((((((((((.(((((.........................((((.....))))((....(((((......)))))....)).....))))))))))))))))). (-39.54 = -39.72 + 0.17)


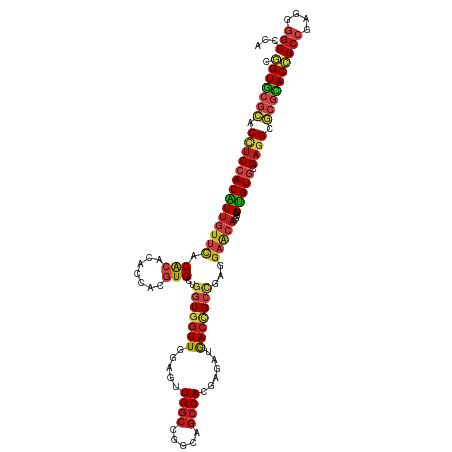
| Location | 11,213,733 – 11,213,853 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.40 |
| Shannon entropy | 0.32007 |
| G+C content | 0.61923 |
| Mean single sequence MFE | -50.93 |
| Consensus MFE | -40.45 |
| Energy contribution | -41.76 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.79 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.13 |
| SVM RNA-class probability | 0.897247 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 11213733 120 - 27905053 CGGUGCGCACCUCCACAUUGUUUACACACACCACGUGCUGGUGGUUCGAGUUGGCCGGCAGCCACGAGAUGACCGCCGAGGAACAAGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((...(((.....)))((((((((((..((.(((.....)))))..))..)))))).)))))))....))))).)))).)))))))(((....)))... ( -53.30, z-score = -1.93, R) >droWil1.scaffold_181089 2106955 120 - 12369635 CGGUGCGCACCUCCACAUUGUUCACACACACCACGUGCUGGUGGUUGGAAUUGGCCGGCAGCCACGAGAUGACCGCUGAGGAACACGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((...(((.....)))((((((((((....((((.....)))).....)))))))).)))))))....))))).)))).)))))))(((....)))... ( -53.30, z-score = -1.64, R) >droMoj3.scaffold_6540 23547425 120 + 34148556 CGGUGCGCACCUCCACAUUGUUCACACACACCACGUGCUGGUGGUUGGAGUUGGCUGGCAGCCAUGAGAUAACCGCCGAGGAACACGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((...(((.....)))((((((((((....((((.....)))).....)))))))).)))))))....))))).)))).)))))))(((....)))... ( -57.90, z-score = -2.88, R) >droVir3.scaffold_13047 5857523 120 + 19223366 CGGUGCGCACCUCCACAUUGUUCACACACACCACGUGCUGGUGGUUGGAGUUGGCCGGCAGCCACGAGAUAACCGCCGAGGAACACGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((...(((.....)))((((((((((..((.(((.....)))))....)))))))).)))))))....))))).)))).)))))))(((....)))... ( -56.60, z-score = -2.40, R) >anoGam1.chr2R 44660503 120 - 62725911 CCGUUCUGACCUCCACGUUGUUCACGCAGACGACGUGCUGAUGGUUGGAGUUGGCCGGUAGCCAGGAGAUGACGGCACUGGAGCAGGUAAUGUGUGUAGCCCUUACAGCCGAUGGUCCUA ((.(((..((((((((((((((......)))))))))..)).)))..))).((((.....)))))).(((.(((((....(((..(((.((....)).))))))...)))).).)))... ( -38.10, z-score = 0.87, R) >droPer1.super_0 10628458 120 - 11822988 CGGUGCGCACCUCCACAUUGUUCACACACACCACGUGUUGGUGGUUGGAAUUGGCCGGCUGCCACGAGAUGACCGCCGAGGAACACGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((...(((.....)))((((((((((....((((.....)))).....)))))))))).)))))....))))).)))).)))))))(((....)))... ( -56.90, z-score = -2.50, R) >dp4.chr2 20937724 120 + 30794189 CGGUGCGCACCUCCACAUUGUUCACACACACCACGUGCUGGUGGUUGGAAUUGGCCGGCUGCCACGAGAUGACCGCCGAGGAACACGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((...(((.....)))((((((((((....((((.....)))).....)))))))).)))))))....))))).)))).)))))))(((....)))... ( -55.40, z-score = -2.00, R) >droGri2.scaffold_14906 4324261 120 - 14172833 CGGUGCGCACCUCCACAUUGUUCACACACACCACAUGCUGGUGGUUGGAAUUGGCCGGCAGCCACGAGAUAACCGCCGAGGAACACGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((.....((.....))((((((((((....((((.....)))).....)))))))).)))))))....))))).)))).)))))))(((....)))... ( -52.90, z-score = -2.20, R) >droEre2.scaffold_4770 10739701 120 - 17746568 CGGUGCGCACCUCCACAUUGUUUACACACACCACGUGCUGGUGGUUCGAGUUGGCCGGCAGCCACGAGAUGACCGCCGAGGAACAAGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((...(((.....)))((((((((((..((.(((.....)))))..))..)))))).)))))))....))))).)))).)))))))(((....)))... ( -53.30, z-score = -1.93, R) >droYak2.chr3R 13613887 120 + 28832112 CGGUGCGCACCUCCACAUUGUUUACACACACCACGUGCUGGUGGUUCGAGUUGGCCGGCAGCCACGAGAUGACCGCCGAGGAACAAGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((...(((.....)))((((((((((..((.(((.....)))))..))..)))))).)))))))....))))).)))).)))))))(((....)))... ( -53.30, z-score = -1.93, R) >droAna3.scaffold_13340 12798186 120 - 23697760 CGGUGCGCACCUCCACAUUGUUCACACACACCACAUGCUGGUGGUUGGAGUUGGCCGGCAGCCACGAGAUGACCGCCGAGGAACACGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((.((..(((((.....)))))..))...(((((.((((.........)).))))))).)))))....))))).)))).)))))))(((....)))... ( -53.50, z-score = -1.65, R) >apiMel3.GroupUn 354805676 120 + 399230636 CCGUCCUAACCUCCACGUUAUUCACACAAACCACGUGUUGAUGAUUGCUAUUGCUCGGCAUCCAAGAUAUGACUGCACUAGUGGCGGUUACGUUCGAUGCCUUGACAGCGGUUGGGCCCA ..(.(((((((.....((((((.((((.......)))).)))))).(((....(..((((((...((..(((((((.(....))))))))..)).))))))..)..)))))))))))... ( -38.30, z-score = -1.73, R) >triCas2.ChLG6 1306938 120 + 13544221 CAGUUCGUACUUCCACAUUGUUGACACAUACGACGUGUUGGUGGUUCGAGUUGGCUGGUAGCCAACUGAUGACAGCCGAUGUUGAGGUUAUGUUAGAGGCUCGGACUGCGCUGGGGCCCA (((((((..((((...((..(..((((.......))))..)..))((.(((((((.....)))))))))((((((((........)))..)))))))))..))))))).((....))... ( -39.30, z-score = -0.08, R) >consensus CGGUGCGCACCUCCACAUUGUUCACACACACCACGUGCUGGUGGUUGGAGUUGGCCGGCAGCCACGAGAUGACCGCCGAGGAACACGUUAUGUGCGAGGCGCGCACCGCCGAGGGGCCCA .(((((((.((((((((((((((.(((.......)))..(((((((.....((((.....))))......)))))))...)))))....))))).)))).)))))))(((....)))... (-40.45 = -41.76 + 1.31)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:17:11 2011