
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 11,206,873 – 11,206,970 |
| Length | 97 |
| Max. P | 0.951034 |

| Location | 11,206,873 – 11,206,970 |
|---|---|
| Length | 97 |
| Sequences | 12 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 84.68 |
| Shannon entropy | 0.31810 |
| G+C content | 0.65413 |
| Mean single sequence MFE | -42.83 |
| Consensus MFE | -35.63 |
| Energy contribution | -35.52 |
| Covariance contribution | -0.10 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.83 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.57 |
| SVM RNA-class probability | 0.951034 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 11206873 97 + 27905053 ---------UUCCAUUGCAGUCGUCCG------AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCAUCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCACCC ---------......((((.(((.(((------.(.((((((....)).)))).).)).).))))))).........((((((((((((....))))))))))))....... ( -43.20, z-score = -2.13, R) >droVir3.scaffold_13047 5851117 99 - 19223366 -------UAUUUAUUCGCAGUCGUCGC------AAGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCAUCAUCAGUCGGCGGCCGCUGCAGUGGCCGCCGGCGUAGCGC -------........(((..(((((..------..)((((((....)).))))..))))...((((......)))).((((((((((((....))))))))))))...))). ( -44.60, z-score = -2.73, R) >droGri2.scaffold_14906 4317180 103 + 14172833 ---UGUCGUUGCAGUCGUCAGUGCAGU------CGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCAUCAUCAGUCGGCGGCCGCUGCCGUGGCCGCCGGCGUAGCGC ---...((((((........(((((.(------((.((((((....)).))))...))).....)))))........((((((((((((....)))))))))))))))))). ( -49.60, z-score = -1.92, R) >droMoj3.scaffold_6540 23540912 99 - 34148556 -------UAUCCAUUCGCAGACGUCAG------AAGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCAUCAUCAGUCGGCGGCCGCUGCCGUGGCCGCCGGCGUAGCGC -------........(((.(((.((.(------.(.((((((....)).)))).).))))))((((......)))).((((((((((((....))))))))))))...))). ( -44.60, z-score = -2.52, R) >droWil1.scaffold_181089 2101089 111 + 12369635 -UUUGUUUGAUUACUUGCAGUCGUUAGUUAGUGAGAGGCAGCCACAGCAUGCCGUACGAGUCGAUGCAUCAUCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGCCAGUGCCC -.........(((((.((........)).)))))..((((((....)).))))((((.....((((......)))).(.((((((((((....)))))))))).).)))).. ( -39.00, z-score = -0.81, R) >droPer1.super_0 10617918 97 + 11822988 ---------UUGCAUUUCAGUCGUCAU------AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCACCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCGCUC ---------.((((((..(.((((...------.(.((((((....)).)))).))))).).)))))).........((((((((((((....))))))))))))....... ( -41.40, z-score = -1.64, R) >dp4.chr2 20927389 97 - 30794189 ---------UUGCAUUUCAGUCGUCAU------AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCACCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCGCUC ---------.((((((..(.((((...------.(.((((((....)).)))).))))).).)))))).........((((((((((((....))))))))))))....... ( -41.40, z-score = -1.64, R) >droAna3.scaffold_13340 12791066 97 + 23697760 ---------UCUUUUUCCAGUCGUCAU------AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCAUCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCGCCC ---------...........((((...------.(.((((((....)).)))).)))))...((((......)))).((((((((((((....))))))))))))....... ( -37.40, z-score = -1.06, R) >droEre2.scaffold_4770 10732813 97 + 17746568 ---------UUCCAUUGCAGUCGUCCG------AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCAUCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCACCC ---------......((((.(((.(((------.(.((((((....)).)))).).)).).))))))).........((((((((((((....))))))))))))....... ( -43.20, z-score = -2.13, R) >droYak2.chr3R 13606851 106 - 28832112 UUCUAUUGCCUCCAUUGCAGUCGUCCG------AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCAUCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCACCC ...............((((.(((.(((------.(.((((((....)).)))).).)).).))))))).........((((((((((((....))))))))))))....... ( -43.20, z-score = -1.43, R) >droSec1.super_0 10379616 97 - 21120651 ---------UUCCAUUGCAGUCGUCCG------AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCACCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCACCC ---------......((((.(((.(((------.(.((((((....)).)))).).)).).))))))).........((((((((((((....))))))))))))....... ( -43.20, z-score = -1.99, R) >droSim1.chr3R 17251857 97 - 27517382 ---------UUCCAUUGCAGUCGUCCG------AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCACCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCACCC ---------......((((.(((.(((------.(.((((((....)).)))).).)).).))))))).........((((((((((((....))))))))))))....... ( -43.20, z-score = -1.99, R) >consensus _________UUCCAUUGCAGUCGUCAG______AGGGGCAGCCACAGCAUGCCGUACGAGUCGAUGCACCAUCAUCAGUCGGCGGCCGCUGCCGUGGCCGCUGGCACCGCCC ....................................((((((....)).)))).........((((......)))).((((((((((((....))))))))))))....... (-35.63 = -35.52 + -0.10)

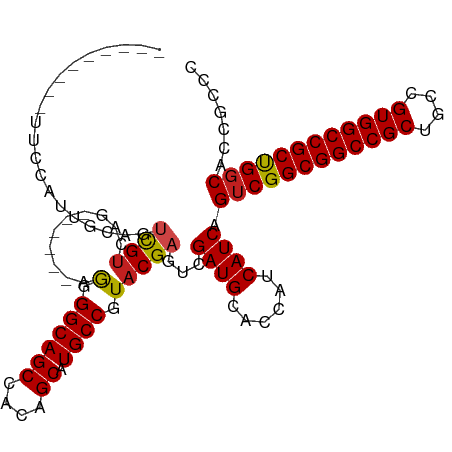
| Location | 11,206,873 – 11,206,970 |
|---|---|
| Length | 97 |
| Sequences | 12 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 84.68 |
| Shannon entropy | 0.31810 |
| G+C content | 0.65413 |
| Mean single sequence MFE | -42.31 |
| Consensus MFE | -36.91 |
| Energy contribution | -36.99 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.80 |
| Structure conservation index | 0.87 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.517719 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3R 11206873 97 - 27905053 GGGUGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGAUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU------CGGACGACUGCAAUGGAA--------- .(((.((.((.((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))....------.)))).))).........--------- ( -42.30, z-score = -0.37, R) >droVir3.scaffold_13047 5851117 99 + 19223366 GCGCUACGCCGGCGGCCACUGCAGCGGCCGCCGACUGAUGAUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCUU------GCGACGACUGCGAAUAAAUA------- .(((..(((.(((((((((.(((...((((.(((.(((((......)))))..)))..)))).))).)))))))))...------)))......)))........------- ( -42.30, z-score = -1.89, R) >droGri2.scaffold_14906 4317180 103 - 14172833 GCGCUACGCCGGCGGCCACGGCAGCGGCCGCCGACUGAUGAUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCG------ACUGCACUGACGACUGCAACGACA--- (((...))).(((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))).((------..((((........)))).))...--- ( -44.60, z-score = -1.48, R) >droMoj3.scaffold_6540 23540912 99 + 34148556 GCGCUACGCCGGCGGCCACGGCAGCGGCCGCCGACUGAUGAUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCUU------CUGACGUCUGCGAAUGGAUA------- .(((.((((.(((((((((((((...((((.(((.(((((......)))))..)))..)))).)))))))))))))...------..).)))..)))........------- ( -44.80, z-score = -1.53, R) >droWil1.scaffold_181089 2101089 111 - 12369635 GGGCACUGGCAGCGGCCACGGCAGCGGCCGCCGACUGAUGAUGAUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCUCUCACUAACUAACGACUGCAAGUAAUCAAACAAA- ((..(((.(((((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))..........(....)..))).)))..)).......- ( -42.90, z-score = -2.55, R) >droPer1.super_0 10617918 97 - 11822988 GAGCGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGGUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU------AUGACGACUGAAAUGCAA--------- ..(((....).((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))....------..............))..--------- ( -39.10, z-score = -0.12, R) >dp4.chr2 20927389 97 + 30794189 GAGCGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGGUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU------AUGACGACUGAAAUGCAA--------- ..(((....).((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))....------..............))..--------- ( -39.10, z-score = -0.12, R) >droAna3.scaffold_13340 12791066 97 - 23697760 GGGCGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGAUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU------AUGACGACUGGAAAAAGA--------- (((.(....).((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))))).------..................--------- ( -40.50, z-score = -0.91, R) >droEre2.scaffold_4770 10732813 97 - 17746568 GGGUGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGAUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU------CGGACGACUGCAAUGGAA--------- .(((.((.((.((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))....------.)))).))).........--------- ( -42.30, z-score = -0.37, R) >droYak2.chr3R 13606851 106 + 28832112 GGGUGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGAUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU------CGGACGACUGCAAUGGAGGCAAUAGAA ......((((.((((((((((((...((((.(((.(((((......)))))..)))..)))).)))))))))))).((.------(((....)))....)).))))...... ( -45.00, z-score = -0.43, R) >droSec1.super_0 10379616 97 + 21120651 GGGUGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGGUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU------CGGACGACUGCAAUGGAA--------- .(((.((.((.((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))....------.)))).))).........--------- ( -42.40, z-score = 0.10, R) >droSim1.chr3R 17251857 97 + 27517382 GGGUGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGGUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU------CGGACGACUGCAAUGGAA--------- .(((.((.((.((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))....------.)))).))).........--------- ( -42.40, z-score = 0.10, R) >consensus GGGCGGUGCCAGCGGCCACGGCAGCGGCCGCCGACUGAUGAUGGUGCAUCGACUCGUACGGCAUGCUGUGGCUGCCCCU______CGGACGACUGCAAUGGAA_________ ...........((((((((((((...((((.(((.(((((......)))))..)))..)))).))))))))))))..................................... (-36.91 = -36.99 + 0.08)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:17:08 2011