
| Sequence ID | dm3.chr3R |
|---|---|
| Location | 11,025,033 – 11,025,192 |
| Length | 159 |
| Max. P | 0.990331 |

| Location | 11,025,033 – 11,025,192 |
|---|---|
| Length | 159 |
| Sequences | 15 |
| Columns | 176 |
| Reading direction | forward |
| Mean pairwise identity | 80.81 |
| Shannon entropy | 0.42184 |
| G+C content | 0.47471 |
| Mean single sequence MFE | -54.23 |
| Consensus MFE | -21.19 |
| Energy contribution | -21.24 |
| Covariance contribution | 0.05 |
| Combinations/Pair | 1.56 |
| Mean z-score | -3.47 |
| Structure conservation index | 0.39 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.41 |
| SVM RNA-class probability | 0.990331 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
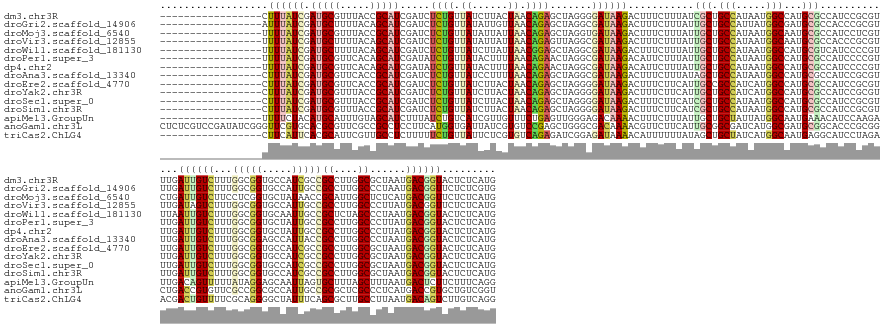
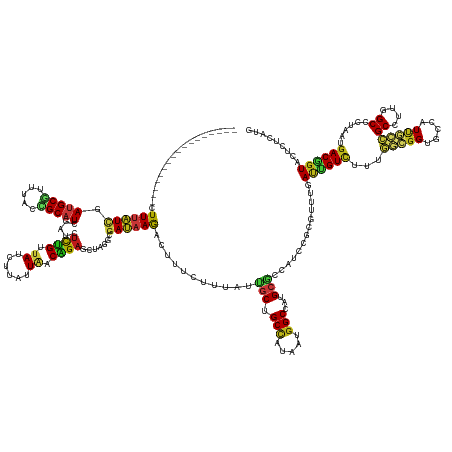
>dm3.chr3R 11025033 159 + 27905053 -----------------CUUUAUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUCUUACUAACAGAGCUAGGGGAUAAGACUUUCUUUAUCGCUGCCAUAAUGGCCAUGCGCCAUCCGCGUUUGAUUGUCUUUGGCGGUGCCAUCGCCGCCUUGGCGCUAAUGACGGUACUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))..((((((((((......))))))((((.(((..((((((.((((.....)))).))...(((...(((((((....)))))))..)))))))))).)))).)))).... ( -56.70, z-score = -3.73, R) >droGri2.scaffold_14906 2829748 159 + 14172833 -----------------AUUUAUCGAUGCUUUUACAGCAUCGAUCUCUGUUAUAUUGUUAACAGAGCUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUUAUGGCGAUGCGCCACCCGCGUUUGAUUGUCUUUGGCGGUGCCAUUGCCGCCUUGGCCCUAAUGACGGUUCUCUCGUG -----------------....(((((((((.....)))))))))((((((((......))))))))...(((((((((......)))))))))((((...((((((((((((.((.(((......)))....)).)))).))))))))...)))).......((((.....)))). ( -56.70, z-score = -4.33, R) >droMoj3.scaffold_6540 7031115 159 + 34148556 -----------------UUUUAUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUAUUAUUAACAGAGCUAGGUGAUAAGACUUUCUUUAUUGCUGCCAUAAUGGCAAUGCGCCAUCCUCGUCUGAUUGUCUUCCUCGGUGCUAUAACCGCAUUGGCUCUCAUGACGGUUCUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))...((..(((((......)))))..))(((...(((((.....)))))....((((((..(((.....((((......))))....)))..))).))))))......... ( -50.20, z-score = -4.47, R) >droVir3.scaffold_12855 6655652 159 + 10161210 -----------------UUUUAUCGAUGCUUUUACAGCAUCGAUCUCUGUUAUAUUAUUAACAGAGUUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAUGGCAAUGCGCCACCCGCGUUUGAUAGUCUUUGGCGGUGCCAUUGCCGCCUUGGCCCUUAUGACGGUUCUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))...(((((((((......)))))))))((((...((((((((((((.((.((........))....)).)))).))))))))...))))...(((((........))))) ( -56.20, z-score = -4.91, R) >droWil1.scaffold_181130 9422077 159 - 16660200 -----------------UUUUAUCGAUGCUUUUACAGCAUCGAUCUCUGUUAUCUUAUUAACGGAGCUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAUGGCCAUGCGUCAUCCCCGUUUAAUUGUCUUUGGCGGUGCAAUUGCCGCUCUAGCCCUAAUGACGGUACUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))...(((((((((......)))))))))(((.....)))((((.(......(((((.......((..((((((......))))))..)).......)))))....).)))) ( -49.94, z-score = -3.91, R) >droPer1.super_3 3761453 159 + 7375914 -----------------UUUUAUCGAUGCGUUCACAGCAUCGAUAUCUGUUAUACUUUUAACAGAACUAGGCGAUAAGACAUUCUUUAUUGCUGCCAUAAUGGCCAUGCGCCAUCCCCGUUUGAUUGUCUUUGGCGGUGCUAUUGCCGCCUUGGCCCUUAUGACGGUACUCUCAUG -----------------...(((((((((.......)))))))))(((((((......)))))))(((.(((((((((......)))))))))..(((((.(((((.((((((....((......))....))))(((......)))))..))))).)))))..)))......... ( -53.70, z-score = -4.56, R) >dp4.chr2 21010277 159 + 30794189 -----------------UUUUAUCGAUGCGUUCACAGCAUCGAUAUCUGUUAUACUUUUAACAGAACUAGGCGAUAAGACAUUCUUUAUUGCUGCCAUAAUGGCCAUGCGCCAUCCCCGUUUGAUUGUCUUUGGCGGUGCUAUUGCCGCCUUGGCCCUUAUGACGGUACUCUCAUG -----------------...(((((((((.......)))))))))(((((((......)))))))(((.(((((((((......)))))))))..(((((.(((((.((((((....((......))....))))(((......)))))..))))).)))))..)))......... ( -53.70, z-score = -4.56, R) >droAna3.scaffold_13340 6114753 159 + 23697760 -----------------CUUUAUCGAUGCGUUCACCGCAUCGAUCUCUGUUAUCCUUUUAACAGAGCUAGGCGAUAAGACUUUCUUUAUAGCUGCCAUAAUGGCCAUGCGCCAUCCGCGUUUGAUUGUCUUUGGCGGAGCCAUUACCGCCUUGGCCCUAAUGACGGUACUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))...(((.(((((......))))).((.(((.....)))...)))))..(((((((.....(((...(((((........)))))..)))...)))).))).......... ( -51.00, z-score = -3.23, R) >droEre2.scaffold_4770 10546600 159 + 17746568 -----------------CUUUAUCGAUGCGUUCACCGCAUCGAUCUCUGUUAUCUUACUAACAGAGCUAGGGGAUAAGACUUUCUUCAUUGCCGCCAUCAUGGCCAUGCGCCAUCCGCGUUUGAUUGUCUUUGGCGGUGCCAUCGCCGCCUUGGCGCUAAUGACGGUACUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))...((.((((((.....))).))).))(((.(((((((((.((((.....)))).))...(((...(((((((....)))))))..)))))).)))).)))......... ( -58.00, z-score = -3.77, R) >droYak2.chr3R 13422649 159 - 28832112 -----------------CUUUAUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUCUUACUAACAGAGCUAGGGGAUAAGACUUUCUUCAUUGCUGCCAUCAUGGCCAUGCGCCAUCCGCGUUUGAUUGUCUUUGGCGGUGCCAUCGCCGCCUUGGCGCUAAUGACGGUACUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))...(((((........))))).....((((.(((((((((.((((.....)))).))...(((...(((((((....)))))))..)))))).)))).))))........ ( -56.90, z-score = -3.69, R) >droSec1.super_0 10193910 159 - 21120651 -----------------CUUUAUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUCUUACUAACAGAGCUAGGGGAUAAGACUUUCUUCAUCGCUGCCAUAAUGGCCAUGCGCCAUCCGCGUUUGAUUGUCUUUGGCGGUGCCAUCGCCGCCUUGGCGCUAAUGACGGUACUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))..((((((((((.....))).)))((((.(((..((((((.((((.....)))).))...(((...(((((((....)))))))..)))))))))).)))).)))).... ( -55.80, z-score = -3.39, R) >droSim1.chr3R 17076404 159 - 27517382 -----------------CUUUAUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUCUUACUAACAGAGCUAGGGGAUAAGACUUUCUUCAUCGCUGCCAUAAUGGCCAUGCGCCAUCCGCGUUUGAUUGUCUUUGGCGGUGCCAUCGCCGCCUUGGCGCUAAUGACGGUACUCUCAUG -----------------....(((((((((.....)))))))))((((((((......))))))))..((((((((((.....))).)))((((.(((..((((((.((((.....)))).))...(((...(((((((....)))))))..)))))))))).)))).)))).... ( -55.80, z-score = -3.39, R) >apiMel3.GroupUn 169499101 159 - 399230636 -----------------UUUUCUACAUGCAUUUGUAGCAUCUUUAUCUGUCAUCGUUGUUUCUGAGUUGGGAGACAAAACUUUCUUUAUUGCUGCUAUUAUGGCAAUGAAACAUCCAAGAUUGACAGUUUUUAUAGGAGCAAUUAGUGCUUUAGCUUUAAUGACUCUUCUUUCAGG -----------------....(((((......))))).........((((((((.((((((((......)))))))).......(((((((((........)))))))))........)).)))))).......(((((..(((((.((....)).)))))..)))))........ ( -36.30, z-score = -0.86, R) >anoGam1.chr3L 34950232 176 + 41284009 CUCUCGUCCGAUAUCGGGUUCGUGCACGCGUUCGCCGCCUCCUUCAUGGUGAUUAUCGUGUCCGAGCUGGGCGACAAAACGUUCUUCAUUGCGGCGAUCAUGGCGAUGCGGCACCCGCGGCUGACCGUGUUCGCCGGCGCCAUUGCCGCGCUCGCCCUCAUGACCGUGCUGUCGGU .......((((((.((((((((..((((...((((((.........))))))....))))..))))).(((((((.....(.....)...(((((((...(((((...((((...(((((....)))))...)))).))))))))))))).))))))......)))...)))))). ( -75.30, z-score = -0.84, R) >triCas2.ChLG4 11348813 159 + 13894384 -----------------CUUCAUUCACGCAUUCGUUGCCUCUUUUUCUGUUAUUCUCGUGUCAGAGAUCGGAGAUAAAACAUUUUUUAUAGCUGCUAUCAUGGCAAUGAGGCAUCCUAGAACGACUGUUUUCGCAGGGGCUAUUUCAGCGCUUGCCUUAAUGACAGUCUUGUCAGG -----------------..........((.(((((((((....((((((.(((....))).)))))).(((..((((((....))))))..))).......)))))))))))..(((.((..(((((((...(((((.(((.....))).)))))......)))))))...))))) ( -47.20, z-score = -2.41, R) >consensus _________________CUUUAUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUCUUAUUAACAGAGCUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAUGGCCAUGCGCCAUCCGCGUUUGAUUGUCUUUGGCGGUGCCAUUGCCGCCUUGGCCCUAAUGACGGUACUCUCAUG ..................((((((.(((((.....))))).....((((.((......)).)))).......))))))...........(((.(((.....)))...))).............((((((...(((((.....)))))((....))......))))))......... (-21.19 = -21.24 + 0.05)
| Location | 11,025,033 – 11,025,192 |
|---|---|
| Length | 159 |
| Sequences | 15 |
| Columns | 176 |
| Reading direction | reverse |
| Mean pairwise identity | 80.81 |
| Shannon entropy | 0.42184 |
| G+C content | 0.47471 |
| Mean single sequence MFE | -52.41 |
| Consensus MFE | -17.59 |
| Energy contribution | -17.43 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.52 |
| Mean z-score | -3.04 |
| Structure conservation index | 0.34 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.22 |
| SVM RNA-class probability | 0.910997 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
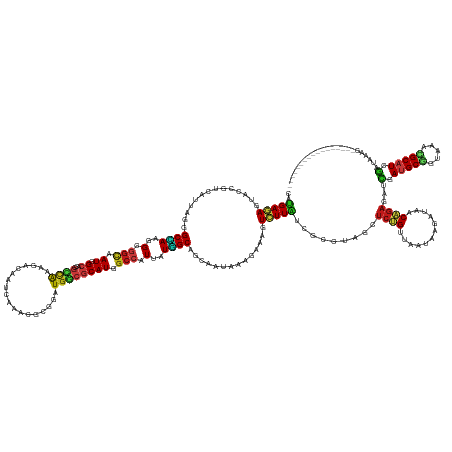
>dm3.chr3R 11025033 159 - 27905053 CAUGAGAGUACCGUCAUUAGCGCCAAGGCGGCGAUGGCACCGCCAAAGACAAUCAAACGCGGAUGGCGCAUGGCCAUUAUGGCAGCGAUAAAGAAAGUCUUAUCCCCUAGCUCUGUUAGUAAGAUAACAGAGAUCGAUGCGGUAAACGCAUCGAUAAAG----------------- .((((((.(.(((((....(((....(((((.(....).)))))...(.....)...))).)))))(((...(((.....))).)))........).)))))).......((((((((......))))))))(((((((((.....)))))))))....----------------- ( -56.50, z-score = -3.84, R) >droGri2.scaffold_14906 2829748 159 - 14172833 CACGAGAGAACCGUCAUUAGGGCCAAGGCGGCAAUGGCACCGCCAAAGACAAUCAAACGCGGGUGGCGCAUCGCCAUAAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAGCUCUGUUAACAAUAUAACAGAGAUCGAUGCUGUAAAAGCAUCGAUAAAU----------------- ..(....)...........((((...(((((........))))).(((((..((....(((.....)))...(((.....))).........))..)))))...))))..((((((((......))))))))(((((((((.....)))))))))....----------------- ( -52.80, z-score = -3.76, R) >droMoj3.scaffold_6540 7031115 159 - 34148556 CAUGAGAGAACCGUCAUGAGAGCCAAUGCGGUUAUAGCACCGAGGAAGACAAUCAGACGAGGAUGGCGCAUUGCCAUUAUGGCAGCAAUAAAGAAAGUCUUAUCACCUAGCUCUGUUAAUAAUAUAACAGAGAUCGAUGCGGUAAACGCAUCGAUAAAA----------------- ..((....(((((.(((........))))))))....))..((..(((((.(((.......)))...((..((((.....))))))..........))))).))......((((((((......))))))))(((((((((.....)))))))))....----------------- ( -47.00, z-score = -3.41, R) >droVir3.scaffold_12855 6655652 159 - 10161210 CAUGAGAGAACCGUCAUAAGGGCCAAGGCGGCAAUGGCACCGCCAAAGACUAUCAAACGCGGGUGGCGCAUUGCCAUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAACUCUGUUAAUAAUAUAACAGAGAUCGAUGCUGUAAAAGCAUCGAUAAAA----------------- .((((........))))..((((...(((((........))))).((((((.((....(((.....))).(((((.....))))).......)).))))))...))))..((((((((......))))))))(((((((((.....)))))))))....----------------- ( -57.00, z-score = -5.08, R) >droWil1.scaffold_181130 9422077 159 + 16660200 CAUGAGAGUACCGUCAUUAGGGCUAGAGCGGCAAUUGCACCGCCAAAGACAAUUAAACGGGGAUGACGCAUGGCCAUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAGCUCCGUUAAUAAGAUAACAGAGAUCGAUGCUGUAAAAGCAUCGAUAAAA----------------- .............((....(((((((.(((((.........))).(((((.........(......)((...(((.....))).))..........)))))...))))))).))((((......)))).)).(((((((((.....)))))))))....----------------- ( -43.40, z-score = -1.78, R) >droPer1.super_3 3761453 159 - 7375914 CAUGAGAGUACCGUCAUAAGGGCCAAGGCGGCAAUAGCACCGCCAAAGACAAUCAAACGGGGAUGGCGCAUGGCCAUUAUGGCAGCAAUAAAGAAUGUCUUAUCGCCUAGUUCUGUUAAAAGUAUAACAGAUAUCGAUGCUGUGAACGCAUCGAUAAAA----------------- .((((........))))..((((...(((((........))))).((((((.((.......((((((.....)))))).((....)).....)).))))))...))))...(((((((......)))))))(((((((((.(....))))))))))...----------------- ( -50.20, z-score = -2.89, R) >dp4.chr2 21010277 159 - 30794189 CAUGAGAGUACCGUCAUAAGGGCCAAGGCGGCAAUAGCACCGCCAAAGACAAUCAAACGGGGAUGGCGCAUGGCCAUUAUGGCAGCAAUAAAGAAUGUCUUAUCGCCUAGUUCUGUUAAAAGUAUAACAGAUAUCGAUGCUGUGAACGCAUCGAUAAAA----------------- .((((........))))..((((...(((((........))))).((((((.((.......((((((.....)))))).((....)).....)).))))))...))))...(((((((......)))))))(((((((((.(....))))))))))...----------------- ( -50.20, z-score = -2.89, R) >droAna3.scaffold_13340 6114753 159 - 23697760 CAUGAGAGUACCGUCAUUAGGGCCAAGGCGGUAAUGGCUCCGCCAAAGACAAUCAAACGCGGAUGGCGCAUGGCCAUUAUGGCAGCUAUAAAGAAAGUCUUAUCGCCUAGCUCUGUUAAAAGGAUAACAGAGAUCGAUGCGGUGAACGCAUCGAUAAAG----------------- .((((((.(...(((((...(((((.(((((........)))))..............(((.....))).)))))...)))))..((....))..).)))))).......((((((((......))))))))(((((((((.....)))))))))....----------------- ( -56.60, z-score = -3.66, R) >droEre2.scaffold_4770 10546600 159 - 17746568 CAUGAGAGUACCGUCAUUAGCGCCAAGGCGGCGAUGGCACCGCCAAAGACAAUCAAACGCGGAUGGCGCAUGGCCAUGAUGGCGGCAAUGAAGAAAGUCUUAUCCCCUAGCUCUGUUAGUAAGAUAACAGAGAUCGAUGCGGUGAACGCAUCGAUAAAG----------------- .((((((.(....(((((..(((((.(((((.(....).)))))..................(((((.....)))))..)))))..)))))....).)))))).......((((((((......))))))))(((((((((.....)))))))))....----------------- ( -59.20, z-score = -3.71, R) >droYak2.chr3R 13422649 159 + 28832112 CAUGAGAGUACCGUCAUUAGCGCCAAGGCGGCGAUGGCACCGCCAAAGACAAUCAAACGCGGAUGGCGCAUGGCCAUGAUGGCAGCAAUGAAGAAAGUCUUAUCCCCUAGCUCUGUUAGUAAGAUAACAGAGAUCGAUGCGGUAAACGCAUCGAUAAAG----------------- .((((((.(....(((((.((((((.(((((.(....).)))))..................(((((.....)))))..)))).)))))))....).)))))).......((((((((......))))))))(((((((((.....)))))))))....----------------- ( -58.90, z-score = -4.28, R) >droSec1.super_0 10193910 159 + 21120651 CAUGAGAGUACCGUCAUUAGCGCCAAGGCGGCGAUGGCACCGCCAAAGACAAUCAAACGCGGAUGGCGCAUGGCCAUUAUGGCAGCGAUGAAGAAAGUCUUAUCCCCUAGCUCUGUUAGUAAGAUAACAGAGAUCGAUGCGGUAAACGCAUCGAUAAAG----------------- .((((((.(....(((((.((((((.(((((.(....).))))).................((((((.....)))))).)))).)))))))....).)))))).......((((((((......))))))))(((((((((.....)))))))))....----------------- ( -59.90, z-score = -4.50, R) >droSim1.chr3R 17076404 159 + 27517382 CAUGAGAGUACCGUCAUUAGCGCCAAGGCGGCGAUGGCACCGCCAAAGACAAUCAAACGCGGAUGGCGCAUGGCCAUUAUGGCAGCGAUGAAGAAAGUCUUAUCCCCUAGCUCUGUUAGUAAGAUAACAGAGAUCGAUGCGGUAAACGCAUCGAUAAAG----------------- .((((((.(....(((((.((((((.(((((.(....).))))).................((((((.....)))))).)))).)))))))....).)))))).......((((((((......))))))))(((((((((.....)))))))))....----------------- ( -59.90, z-score = -4.50, R) >apiMel3.GroupUn 169499101 159 + 399230636 CCUGAAAGAAGAGUCAUUAAAGCUAAAGCACUAAUUGCUCCUAUAAAAACUGUCAAUCUUGGAUGUUUCAUUGCCAUAAUAGCAGCAAUAAAGAAAGUUUUGUCUCCCAACUCAGAAACAACGAUGACAGAUAAAGAUGCUACAAAUGCAUGUAGAAAA----------------- ..........((((.((((..((....))..)))).))))((((.....((((((...(((..(((((((((((..........))))).......(..(((.....)))..).)))))).)))))))))......((((.......))))))))....----------------- ( -29.50, z-score = -0.67, R) >anoGam1.chr3L 34950232 176 - 41284009 ACCGACAGCACGGUCAUGAGGGCGAGCGCGGCAAUGGCGCCGGCGAACACGGUCAGCCGCGGGUGCCGCAUCGCCAUGAUCGCCGCAAUGAAGAACGUUUUGUCGCCCAGCUCGGACACGAUAAUCACCAUGAAGGAGGCGGCGAACGCGUGCACGAACCCGAUAUCGGACGAGAG .((((..((((..(((((.((((((..(((((.((((((.(((((..(.(((....))).)..)))))...))))))....)))))((((.....))))...))))))...(((....))).......))))).....(((.....))))))).((....))...))))....... ( -70.60, z-score = -0.41, R) >triCas2.ChLG4 11348813 159 - 13894384 CCUGACAAGACUGUCAUUAAGGCAAGCGCUGAAAUAGCCCCUGCGAAAACAGUCGUUCUAGGAUGCCUCAUUGCCAUGAUAGCAGCUAUAAAAAAUGUUUUAUCUCCGAUCUCUGACACGAGAAUAACAGAAAAAGAGGCAACGAAUGCGUGAAUGAAG----------------- ((((....((((((..((...(((.(.((((...)))).).)))))..))))))....))))((((.((.(((((..(((((.(((..........)))))))).....((((......))))..............))))).))..))))........----------------- ( -34.40, z-score = -0.15, R) >consensus CAUGAGAGUACCGUCAUUAGGGCCAAGGCGGCAAUGGCACCGCCAAAGACAAUCAAACGCGGAUGGCGCAUGGCCAUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAGCUCUGUUAAUAAGAUAACAGAGAUCGAUGCGGUAAACGCAUCGAUAAAG_________________ ..(((((..............((((..(.(((.((.((...((((..................)))))))).))).)..))))..............))))).........((((............))))...(.(((((.....))))).)....................... (-17.59 = -17.43 + -0.16)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 00:16:49 2011