Overview on processed synteny regions for HOXD9_ENSG00000128709_5e5.protein.info
| Rank | AlnOverview | AlnScore | RatioSum | logRatioSum | focalRatioSum | TargetGenome | chr | startPos | endPos | hitsCount | queryCov | meanIdentity | ori | score | compara |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 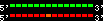 | 14000 | 865 | 56.7 | 272.45 | Homo_sapiens | 2 | 176695773 | 176697146 | 2 | 100% | 94% | 1 | 681 | within_species_paralog: HOXD9 |
| 2 | 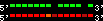 | 10726 | 182.96 | 25.99 | 53.31 | Mus_musculus | 2 | 74498895 | 74500257 | 2 | 100% | 75.6% | 1 | 556 | ortholog_one2one: Hoxd9 |
| 3 |  | 10283 | 264.58 | 26.1 | 48.75 | Macaca_mulatta | 12 | 39764093 | 39764701 | 1 | 58.5% | 76% | 1 | 303 | noComparaHits: |
| 4 |  | 9379 | 52.54 | 14.91 | 13.97 | Monodelphis_domestica | 4 | 187464529 | 187465949 | 2 | 95.9% | 61.6% | 1 | 389 | ortholog_one2one: HOXD9 |
| 5 |  | 8278 | 89.55 | 12.88 | 36.02 | Canis_familiaris | 36 | 22958266 | 22959115 | 2 | 48% | 73.9% | 1 | 268 | noComparaHits: ENSCAFG00000023404 |
| 6 | 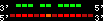 | 8093 | 155.18 | 20.14 | 58.36 | Bos_taurus | 2 | 16998544 | 16999927 | 2 | 100% | 82.1% | -1 | 577 | ortholog_one2one: HOXD9 |
| 7 |  | 6880 | 23.5 | 6.81 | 6.56 | Xenopus_tropicalis | scaffold_163 | 619634 | 620793 | 2 | 94.7% | 52.8% | -1 | 293 | ortholog_one2one: HOXD9 |
| 8 |  | 5683 | 19.22 | 3.28 | 4.76 | Gallus_gallus | 7 | 17409077 | 17409745 | 1 | 76.9% | 46.2% | -1 | 198 | noComparaHits: NP_997060.1 |
| 9 | 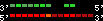 | 4111 | 11.75 | 1.89 | 3.79 | Danio_rerio | 9 | 1580195 | 1581290 | 2 | 99.4% | 47.9% | -1 | 264 | ortholog_one2many: hoxd9a |
| 10 | 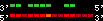 | 3930 | 10.4 | 1.93 | 3.46 | Takifugu_rubripes | scaffold_100 | 358386 | 359535 | 2 | 68.4% | 55.6% | -1 | 244.6 | ortholog_one2one: NEWSINFRUG00000124776 |
| 11 | 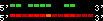 | 3922 | 10.57 | 1.99 | 3.42 | Gasterosteus_aculeatus | contig_5303 | 254123 | 255324 | 2 | 74.6% | 49.2% | 1 | 217.9 | noComparaHits: Q4VQD4_GASAC |
| 12 | 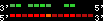 | 3922 | 10.57 | 1.99 | 3.42 | Gasterosteus_aculeatus | groupXVI | 9814156 | 9815357 | 2 | 74.6% | 49.2% | -1 | 217.9 | ortholog_one2one: Q4VQD4_GASAC |
| 13 | 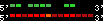 | 3778 | 10.12 | 1.68 | 3.32 | Oryzias_latipes | 21 | 24614922 | 24616093 | 2 | 74.6% | 50% | 1 | 243 | ortholog_one2one: HXD9_ORYLA |
| 14 | 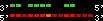 | 3743 | 9.8 | 1.51 | 3.22 | Tetraodon_nigroviridis | 2 | 11097829 | 11098947 | 1 | 98.5% | 39.1% | -1 | 207 | ortholog_one2one: GSTENG00031462001 |
| 15 | 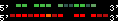 | 3343 | 8.63 | -0.47 | 2.98 | Xenopus_tropicalis | scaffold_56 | 1415310 | 1416623 | 2 | 68.4% | 55.2% | 1 | 249 | noComparaHits: ENSXETG00000000724 |
| 16 | 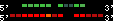 | 3255 | 7.79 | -0.22 | 3 | Xenopus_tropicalis | scaffold_226 | 388156 | 390489 | 2 | 76% | 53.2% | 1 | 252 | noComparaHits: hoxc9 |
| 17 | 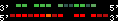 | 3177 | 8.37 | -0.72 | 2.89 | Homo_sapiens | 7 | 27169762 | 27171601 | 2 | 85.1% | 47.9% | -1 | 237.5 | within_species_paralog: HOXA9 |
| 18 | 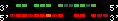 | 3171 | 8.39 | -0.71 | 2.91 | Mus_musculus | 6 | 52153842 | 52155687 | 2 | 91.8% | 46.3% | -1 | 239.3 | between_species_paralog: Hoxa9 |
| 19 | 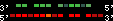 | 3152 | 8.31 | -0.79 | 2.85 | Gasterosteus_aculeatus | groupX | 9901362 | 9902621 | 2 | 54.4% | 58.5% | -1 | 214.1 | between_species_paralog: ENSGACG00000007123 |
| 20 | 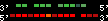 | 3134 | 8.31 | -0.8 | 2.85 | Oryzias_latipes | 11 | 10539576 | 10540702 | 2 | 80.7% | 43% | -1 | 213.2 | noComparaHits: Q9PTV6_ORYLA |
| 21 | 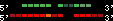 | 3126 | 8.41 | -0.68 | 3.06 | Danio_rerio | 23 | 35676857 | 35678677 | 2 | 79.2% | 52% | 1 | 264 | between_species_paralog: hoxc9a |
| 22 | 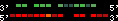 | 3024 | 8.15 | -0.98 | 2.85 | Canis_familiaris | 14 | 43293519 | 43295428 | 2 | 92.1% | 46.5% | -1 | 242.1 | between_species_paralog: 482372 |
| 23 | 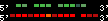 | 2884 | 7.97 | -1.17 | 2.79 | Tetraodon_nigroviridis | 21 | 3012226 | 3013378 | 2 | 80.4% | 46% | 1 | 219.3 | between_species_paralog: HOXA9
; GSTENG00017478001 |
| 24 | 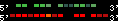 | 2813 | 7.94 | -1.25 | 2.62 | Monodelphis_domestica | 8 | 293276346 | 293276780 | 1 | 51.8% | 37.3% | 1 | 107 | between_species_paralog: HOXA9 |
| 25 | 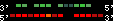 | 2812 | 7.95 | -1.26 | 2.77 | Gallus_gallus | 2 | 32579731 | 32581455 | 2 | 89.2% | 45.3% | -1 | 232.1 | between_species_paralog: HXA9_CHICK |
| 26 | 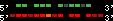 | 2787 | 7.89 | -1.32 | 2.74 | Macaca_mulatta | 3 | 99059984 | 99061835 | 2 | 85.1% | 48.5% | 1 | 240.6 | between_species_paralog: HOXA9 |
| 27 | 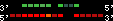 | 2767 | 6.72 | -0.3 | 2.71 | Takifugu_rubripes | scaffold_66 | 154997 | 156667 | 2 | 66.7% | 57.3% | -1 | 251 | between_species_paralog: HXC9_FUGRU |
| 28 | 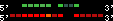 | 2766 | 6.7 | -0.31 | 2.72 | Tetraodon_nigroviridis | 9 | 4222544 | 4224247 | 2 | 66.7% | 57.6% | 1 | 252 | between_species_paralog: HOXC9
; GSTENG00028039001 |
| 29 | 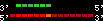 | 2764 | 7.83 | 1.12 | 3 | Danio_rerio | 2 | 12067419 | 12068514 | 2 | 99.4% | 47.9% | -1 | 264 | ortholog_one2many: hoxd9a |
| 30 | 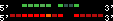 | 2764 | 6.71 | -0.31 | 2.72 | Oryzias_latipes | 7 | 12875582 | 12877170 | 2 | 66.7% | 58.7% | 1 | 255 | between_species_paralog: HXC9_ORYLA |
| 31 |  | 2699 | 6.63 | -0.39 | 2.77 | Macaca_mulatta | 11 | 51098616 | 51101080 | 2 | 67.8% | 59.2% | 1 | 266 | ortholog_one2many: HOXC9 |
| 32 | 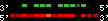 | 2669 | 7.12 | -1.01 | 2.63 | Takifugu_rubripes | scaffold_12 | 2360015 | 2361166 | 2 | 69% | 50% | -1 | 216.4 | between_species_paralog: HXA9_FUGRU |
| 33 | 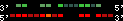 | 2635 | 7.39 | -0.7 | 2.44 | Danio_rerio | 3 | 23019703 | 23019927 | 1 | 21.9% | 86.7% | -1 | 144 | between_species_paralog: hoxb9a |
| 34 | 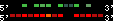 | 2603 | 6.68 | -0.34 | 2.63 | Gasterosteus_aculeatus | groupXII | 11616265 | 11618032 | 2 | 66.7% | 58.1% | 1 | 254 | between_species_paralog: ENSGACG00000009396 |
| 35 | 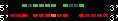 | 2597 | 6.53 | -0.53 | 2.74 | Homo_sapiens | 12 | 52680240 | 52682713 | 2 | 67.8% | 59.2% | 1 | 266 | within_species_paralog: HOXC9 |
| 36 | 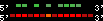 | 2517 | 7 | -1.15 | 2.19 | Danio_rerio | 19 | 13917746 | 13917979 | 1 | 22.8% | 82.1% | 1 | 139 | between_species_paralog: hoxa9a |
| 37 | 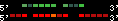 | 2474 | 6.33 | -0.76 | 2.68 | Mus_musculus | 15 | 102809687 | 102812161 | 2 | 67.8% | 59.2% | 1 | 266 | between_species_paralog: Hoxc9 |
| 38 | 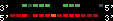 | 2109 | 5.86 | -1.34 | 1.79 | Canis_familiaris | 27 | ||||||||
| 39 |  | 2105 | 5.42 | -0.65 | 2.2 | Bos_taurus | 5 | 16811875 | 16814511 | 2 | 67.8% | 59.2% | -1 | 266 | between_species_paralog: HOXC9 |
| 40 |  | 2082 | 5.63 | -0.4 | 1.89 | Mus_musculus | 11 | 96090716 | 96090937 | 1 | 21.6% | 89.2% | 1 | 145 | between_species_paralog: Hoxb9 |
| 41 |  | 2075 | 5.62 | -0.4 | 1.88 | Homo_sapiens | 17 | 44055273 | 44055494 | 1 | 21.6% | 89.2% | -1 | 145 | within_species_paralog: HOXB9 |
| 42 |  | 2044 | 6.07 | -1.06 | 1.9 | Takifugu_rubripes | scaffold_41 | 540964 | 541188 | 1 | 21.9% | 86.7% | 1 | 142 | noComparaHits: NEWSINFRUG00000160069 |
| 43 | 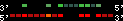 | 1994 | 5.46 | -0.58 | 1.85 | Canis_familiaris | 9 | 28212650 | 28212871 | 1 | 21.6% | 89.2% | -1 | 145 | ortholog_one2many: 608958 |
| 44 | 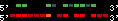 | 1985 | 6.01 | -1.14 | 1.89 | Gasterosteus_aculeatus | groupXI | 1626200 | 1626433 | 1 | 22.8% | 85.9% | 1 | 146 | noComparaHits: |
| 45 | 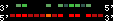 | 1976 | 5.98 | -1.18 | 1.88 | Tetraodon_nigroviridis | Un_random | 38140064 | 38140297 | 1 | 22.8% | 84.6% | -1 | 143 | noComparaHits: GSTENG00020456001 |
| 46 |  | 1924 | 5.41 | -0.68 | 1.83 | Gallus_gallus | 27 | 3567989 | 3568210 | 1 | 21.6% | 89.2% | 1 | 144 | between_species_paralog: HOXB9 |
| 47 | 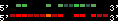 | 1899 | 5.91 | -1.26 | 1.86 | Oryzias_latipes | 8 | 24318538 | 24318762 | 1 | 21.9% | 86.7% | 1 | 142 | between_species_paralog: Q3V615_ORYLA |
| 48 | 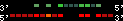 | 1870 | 5.25 | -0.87 | 1.89 | Xenopus_tropicalis | scaffold_334 | 579382 | 579603 | 1 | 21.6% | 86.5% | -1 | 143 | between_species_paralog: HOXB9 |
| 49 | 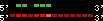 | 1848 | 4.73 | -0.28 | 2.05 | Bos_taurus | 4 | 40177480 | 40179364 | 2 | 88.3% | 47.5% | 1 | 244.5 | between_species_paralog: HOXA9 |
| 50 | 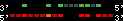 | 1826 | 5.27 | -0.81 | 1.8 | Macaca_mulatta | 16 | 32852664 | 32852885 | 1 | 21.6% | 89.2% | -1 | 145 | between_species_paralog: HOXB9 |
| 51 | 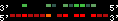 | 1823 | 5.28 | -0.84 | 1.77 | Monodelphis_domestica | 2 | 201204831 | 201205052 | 1 | 21.6% | 89.2% | -1 | 145 | between_species_paralog: HOXB9 |
| 52 | 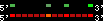 | 1758 | 6.6 | 1.21 | 1.61 | Gasterosteus_aculeatus | groupVI | 16162358 | 16162570 | 1 | 20.8% | 87.3% | 1 | 134 | noComparaHits: ENSGACG00000011902 |
| 53 | 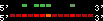 | 1631 | 4.44 | -0.65 | 2 | Danio_rerio | 16 | 21188212 | 21189729 | 2 | 56.1% | 57% | 1 | 203.6 | between_species_paralog: hoxa9b |
| 54 | 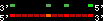 | 1586 | 4.74 | 0.45 | 1.38 | Oryzias_latipes | 15 | 4374398 | 4374607 | 1 | 20.5% | 87.1% | -1 | 133 | noComparaHits: Q9PVR1_ORYLA |
| 55 | 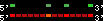 | 1570 | 4.76 | 0.42 | 1.38 | Takifugu_rubripes | scaffold_39 | 584702 | 584911 | 1 | 20.5% | 85.7% | 1 | 132 | noComparaHits: Q4VN43_FUGRU |
| 56 | 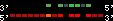 | 1459 | 4.27 | -0.85 | 1.57 | Gasterosteus_aculeatus | contig_2344 | 65914 | 66156 | 1 | 23.7% | 81.5% | -1 | 144 | noComparaHits: |
| 57 | 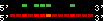 | 1430 | 4.18 | -0.95 | 1.83 | Oryzias_latipes | 16 | 13131568 | 13131795 | 1 | 22.2% | 78.9% | 1 | 134 | between_species_paralog: Q3V626_ORYLA |
| 58 | 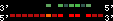 | 1342 | 4.11 | -1.02 | 1.53 | Bos_taurus | 19 | 31239444 | 31239665 | 1 | 21.6% | 89.2% | -1 | 145 | between_species_paralog: HOXB9 |
| 59 |  | 1333 | 4.07 | -1.08 | 1.77 | Gasterosteus_aculeatus | contig_1095 | 43335 | 43559 | 1 | 21.9% | 77.3% | 1 | 130 | noComparaHits: ENSGACG00000008310 |
| 60 |  | 1333 | 4.07 | -1.08 | 1.77 | Gasterosteus_aculeatus | groupXX | 9727575 | 9727799 | 1 | 21.9% | 77.3% | 1 | 130 | between_species_paralog: ENSGACG00000008310 |
| 61 | 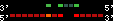 | 1318 | 2.93 | -0.06 | 1.54 | Gasterosteus_aculeatus | contig_2643 | 45188 | 46955 | 2 | 66.7% | 58.1% | -1 | 254 | noComparaHits: ENSGACG00000009396 |
| 62 |  | 1305 | 4.04 | -1.11 | 1.77 | Takifugu_rubripes | scaffold_48 | 1063108 | 1063341 | 1 | 22.8% | 79.5% | -1 | 137 | between_species_paralog: NEWSINFRUG00000138062 |
| 63 |  | 1305 | 4.04 | -1.12 | 1.83 | Tetraodon_nigroviridis | 8 | 6620992 | 6621216 | 1 | 21.9% | 81.3% | 1 | 135 | between_species_paralog: HOXAb9 |
| 64 |  | 1224 | 2.95 | -0.04 | 1.01 | Gasterosteus_aculeatus | contig_2345 | ||||||||
| 65 |  | 1192 | 3.21 | 0 | 1.13 | Tetraodon_nigroviridis | 17 | 9576378 | 9576557 | 1 | 17.5% | 85% | 1 | 113 | noComparaHits: GSTENG00018723001
; HOXDb9 |
| 66 | 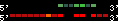 | 1158 | 3.42 | -0.71 | 0.77 | Bos_taurus | 4 | ||||||||
| 67 | 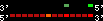 | 1095 | 4.14 | 0.88 | 0.61 | Gasterosteus_aculeatus | contig_4903 | ||||||||
| 68 | 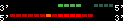 | 937 | 3.14 | -1.04 | 0.74 | Danio_rerio | 12 | ||||||||
| 69 | 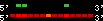 | 925 | 2.58 | -0.49 | 0.52 | Takifugu_rubripes | scaffold_346 | ||||||||
| 70 | 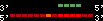 | 755 | 2.9 | -1.34 | 0.66 | Oryzias_latipes | 19 | ||||||||
| 71 | 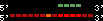 | 744 | 2.91 | -1.33 | 0.67 | Takifugu_rubripes | scaffold_130 | ||||||||
| 72 | 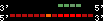 | 724 | 2.9 | -1.35 | 0.66 | Tetraodon_nigroviridis | 2 | ||||||||
| 73 | 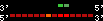 | 710 | 1.65 | -0.42 | 0.5 | Danio_rerio | 3 | ||||||||
| 74 | 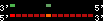 | 551 | 2.34 | 0.18 | 0.98 | Gasterosteus_aculeatus | contig_4905 | 15877 | 16089 | 1 | 20.8% | 87.3% | -1 | 134 | noComparaHits: ENSGACG00000011902 |
| 75 | 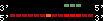 | 536 | 2.29 | -0.86 | 0.46 | Gasterosteus_aculeatus | contig_6417 | ||||||||
| 76 | 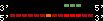 | 536 | 2.29 | -0.86 | 0.46 | Gasterosteus_aculeatus | groupV | ||||||||
| 77 | 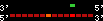 | 535 | 1.22 | 0.2 | 0.24 | Tetraodon_nigroviridis | Un_random | ||||||||
| 78 | 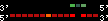 | 517 | 1.84 | -0.16 | 0.34 | Gasterosteus_aculeatus | contig_9872 | ||||||||
| 79 | 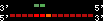 | 486 | 1.48 | -0.61 | 0.63 | Tetraodon_nigroviridis | 3 | ||||||||
| 80 | 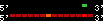 | 462 | 0.28 | -1.26 | 0.04 | Bos_taurus | 21 | ||||||||
| 81 | 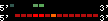 | 446 | 1.55 | -0.5 | 0.45 | Danio_rerio | 11 | ||||||||
| 82 |  | 430 | 1.28 | -0.88 | 0.37 | Gasterosteus_aculeatus | contig_9874 | ||||||||
| 83 |  | 408 | 1.03 | 0.03 | 0.21 | Takifugu_rubripes | scaffold_3110 | ||||||||
| 84 |  | 407 | 0.9 | -0.1 | 0.3 | Gasterosteus_aculeatus | contig_2645 | ||||||||
| 85 |  | 406 | 1.38 | -0.75 | 0.97 | Gasterosteus_aculeatus | contig_9877 | 17464 | 17697 | 1 | 22.8% | 85.9% | -1 | 146 | noComparaHits: |
| 86 |  | 402 | 1.39 | -0.73 | 0.59 | Gallus_gallus | Un_random | ||||||||
| 87 |  | 400 | 0.9 | -0.1 | 0.3 | Gallus_gallus | Un_random | ||||||||
| 88 |  | 385 | 1 | 0 | 0.2 | Gallus_gallus | Un_random | ||||||||
| 89 |  | 384 | 1 | 0 | 0.2 | Danio_rerio | 6 | ||||||||
| 90 |  | 309 | 0.74 | -0.29 | 0.19 | Gallus_gallus | Un_random | ||||||||
| 91 |  | 304 | 1.43 | -0.66 | 1.07 | Gallus_gallus | Un_random | 20047888 | 20048409 | 1 | 53.2% | 41.8% | -1 | 108 | ortholog_one2many: HXD9_CHICK |
| 92 |  | 248 | 0.8 | -0.21 | 0.8 | Macaca_mulatta | 1099214175475 | 321 | 578 | 1 | 25.1% | 94.2% | 1 | 169 | noComparaHits: |
| 93 |  | 221 | 0.66 | -0.41 | 0.17 | Takifugu_rubripes | scaffold_3809 | ||||||||
| 94 |  | 211 | 0.77 | -0.25 | 0.77 | Danio_rerio | 24 | 25218450 | 25218674 | 1 | 21.9% | 86.7% | 1 | 144 | between_species_paralog: hoxb9a |
| 95 |  | 208 | 0.76 | -0.26 | 0.76 | Takifugu_rubripes | scaffold_4108 | 5963 | 6187 | 1 | 21.9% | 86.7% | 1 | 142 | noComparaHits: NEWSINFRUG00000159744 |
| 96 |  | 204 | 0.73 | -0.3 | 0.37 | Gasterosteus_aculeatus | contig_2644 | ||||||||
| 97 |  | 201 | 0.62 | -0.47 | 0.21 | Tetraodon_nigroviridis | Un_random | ||||||||
| 98 |  | 200 | 0.64 | -0.44 | 0.16 | Monodelphis_domestica | Un | ||||||||
| 99 |  | 181 | 0.63 | -0.45 | 0.16 | Gallus_gallus | Un_random | ||||||||
| 100 |  | 181 | 0.63 | -0.45 | 0.16 | Takifugu_rubripes | scaffold_95 | ||||||||
| 101 |  | 117 | 0.64 | -0.44 | 0.13 | Bos_taurus | 12 | ||||||||
| 102 |  | 114 | 0.64 | -0.44 | 0.13 | Monodelphis_domestica | 4 | ||||||||
| 103 | 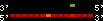 | 113 | 0.64 | -0.44 | 0.13 | Gallus_gallus | 1 | ||||||||
| 104 | 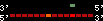 | 112 | 0.64 | -0.44 | 0.13 | Canis_familiaris | 25 | ||||||||
| 105 | 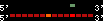 | 110 | 0.64 | -0.44 | 0.13 | Mus_musculus | 5 | ||||||||
| 106 | 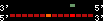 | 109 | 0.63 | -0.45 | 0.13 | Tetraodon_nigroviridis | 6 |
Data table for reference protein loci
QueryProtein(s)=ENSP00000249499 (contained in p7)QueryGenome=Homo_sapiens
| ref-loci | geneName | protID | transID | geneID | protName | chr | startPos | endPos | meanPos | ori | numExons | protLength | selfscore |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | KIAA1715 | ENSP00000272748 | ENST00000272748 | ENSG00000144320 | LNP_HUMAN | 2 | 176502941 | 176568558 | 176520512 | -1 | 12 | 428 | 789.2 |
| p2 | EVX2 | ENSP00000312385 | ENST00000308618 | ENSG00000174279 | EVX2_HUMAN | 2 | 176653081 | 176656750 | 176653792 | -1 | 3 | 476 | 815 |
| p3 | HOXD13 | ENSP00000249505 | ENST00000249505 | ENSG00000128714 | HXD13_HUMAN | 2 | 176665889 | 176667704 | 176666387 | 1 | 2 | 335 | 648 |
| p4 | HOXD12 | ENSP00000303578 | ENST00000306412 | ENSG00000170178 | HXD12_HUMAN | 2 | 176672776 | 176673734 | 176673190 | 1 | 3 | 278 | 558 |
| p5 | HOXD11 | ENSP00000249504 | ENST00000249504 | ENSG00000128713 | NP_067015.2 | 2 | 176680330 | 176682116 | 176680834 | 1 | 2 | 338 | 556 |
| p6 | HOXD10 | ENSP00000249501 | ENST00000249501 | ENSG00000128710 | HXD10_HUMAN | 2 | 176689808 | 176692205 | 176690315 | 1 | 2 | 340 | 693 |
| p7 | HOXD9 | ENSP00000249499 | ENST00000249499 | ENSG00000128709 | HXD9_HUMAN | 2 | 176695773 | 176697149 | 176696283 | 1 | 2 | 342 | 681 |
| p8 | Q8NAT4_HUMAN | ENSP00000316098 | ENST00000313266 | ENSG00000175892 | Q8NAT4_HUMAN | 2 | 176701687 | 176702151 | 176701915 | -1 | 1 | 154 | 303 |
| p9 | HOXD8 | ENSP00000315949 | ENST00000313173 | ENSG00000175879 | HXD8_HUMAN | 2 | 176703341 | 176704586 | 176703773 | 1 | 2 | 290 | 546 |
| p10 | HOXD4 | ENSP00000302548 | ENST00000306324 | ENSG00000170166 | HXD4_HUMAN | 2 | 176724608 | 176725916 | 176724986 | 1 | 2 | 255 | 550 |
| p11 | HOXD3 | ENSP00000249440 | ENST00000249440 | ENSG00000128652 | HXD3_HUMAN | 2 | 176742089 | 176745248 | 176744595 | 1 | 2 | 432 | 917 |
| p12 | HOXD1 | ENSP00000328598 | ENST00000331462 | ENSG00000128645 | HXD1_HUMAN | 2 | 176761776 | 176763116 | 176762265 | 1 | 2 | 328 | 631 |
| p13 | - | ENSP00000346209 | ENST00000331781 | ENSG00000182553 | - | 2 | 176773882 | 176774226 | 176774050 | -1 | 1 | 114 | 227 |
| p14 | MTX2 | ENSP00000249442 | ENST00000249442 | ENSG00000128654 | MTX2_HUMAN | 2 | 176842604 | 176910638 | 176901860 | 1 | 10 | 263 | 455 |
Parameter used for the loci-alignments
qLoci-Score_CutOff: 100 bitsqLoci-MaxProtOverlap: 40 aa
loci size factor: 2 (additional loci size constraint: 0 nt)
loci-grouping type: loci intervalls
max distance between non-highlighted (orange) target columns: 500000 nt
RelativeScore color index:
| <= 33 | <= 66 | <= 100 | <= 133 | <= 166 | <= 200 | <= 233 | <= 266 | <= 300 | <= 333 | <= 366 | <= 400 | <= 433 | <= 466 | <= 500 | <= 533 | <= 566 | <= 600 | <= 633 | <= 666 | <= 700 | <= 733 | <= 766 | <= 800 | <= 833 | <= 866 | <= 900 | <= 933 | <= 966 | <= 1000 | relSc>1000 |
| <= 33 | <= 66 | <= 100 | <= 133 | <= 166 | <= 200 | <= 233 | <= 266 | <= 300 | <= 333 | <= 366 | <= 400 | <= 433 | <= 466 | <= 500 | <= 533 | <= 566 | <= 600 | <= 633 | <= 666 | <= 700 | <= 733 | <= 766 | <= 800 | <= 833 | <= 866 | <= 900 | <= 933 | <= 966 | <= 1000 | relSc>1000 |
region 1 (region 1 for Hsap)
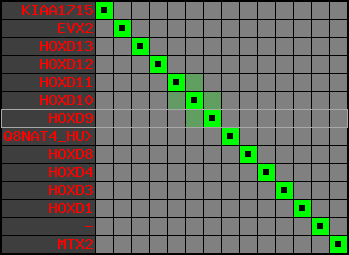 |
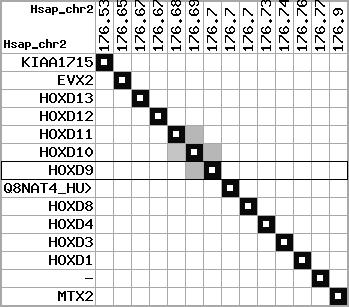 |
 |
retrieve target region: getEnsemblProteins.pl -h Homo_sapiens -l 2 -i 176502944:176910635 -f 31e4 some_geneID_to_be_selected
scoreRatio_sum=865 log_scoreRatio_sum=56.7 focal_scoreRatio_sum=272.45
AlnScore=14000 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Homo_sapiens
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 789.2 | 2 | 176502944 | 176565395 | -1 | 8 | 87.1% | 98.9% | 1131 | 1 / 0.01 = 100 | noComparaHits: KIAA1715 | 1 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 815 | 2 | 176653084 | 176656750 | -1 | 3 | 100% | 80.6% | 1578 | 0.62 / 0.01 = 62 | within_species_paralog: EVX2 | 1 |
| p3 | q3 | +/+ | ENSP00000249505 | HOXD13 | q3 | q3 | 648 | 2 | 176665889 | 176667701 | 1 | 2 | 100% | 94.9% | 1008 | 0.61 / 0.01 = 61 | within_species_paralog: HOXD13 | 1 |
| p4 | q4 | +/+ | ENSP00000303578 | HOXD12 | q4 | q4 | 558 | 2 | 176672776 | 176673731 | 1 | 2 | 100% | 93.7% | 897 | 0.66 / 0.01 = 66 | noComparaHits: HOXD12 | 1 |
| p5 | q5 | +/+ | ENSP00000249504 | HOXD11 | q5 | q5 | 556 | 2 | 176680330 | 176682113 | 1 | 2 | 100% | 75.6% | 1119 | 0.53 / 0.01 = 53 | within_species_paralog: HOXD11 | 1 |
| p6 | q6 | +/+ | ENSP00000249501 | HOXD10 | q6 | q6 | 693 | 2 | 176689808 | 176692202 | 1 | 2 | 100% | 100% | 1020 | 0.58 / 0.01 = 58 | noComparaHits: HOXD10 | 1 |
| p7 | q7 | +/+ | ENSP00000249499 | HOXD9 | q7 | q7 | 681 | 2 | 176695773 | 176697146 | 1 | 2 | 100% | 94% | 1044 | 0.6 / 0.01 = 60 | within_species_paralog: HOXD9 | 1 |
| p8 | q8 | +/+ | ENSP00000316098 | Q8NAT4_HUMAN | q8 | q8 | 303 | 2 | 176701690 | 176702151 | -1 | 1 | 100% | 100% | 462 | 1 / 0.01 = 100 | noComparaHits: Q8NAT4_HUMAN | 1 |
| p9 | q9 | +/+ | ENSP00000315949 | HOXD8 | q9 | q9 | 546 | 2 | 176703341 | 176704583 | 1 | 2 | 100% | 90% | 870 | 0.49 / 0.01 = 49 | within_species_paralog: HOXD8 | 1 |
| p10 | q10 | +/+ | ENSP00000302548 | HOXD4 | q10 | q10 | 550 | 2 | 176724608 | 176725913 | 1 | 2 | 100% | 99.6% | 768 | 0.51 / 0.01 = 51 | within_species_paralog: HOXD4 | 1 |
| p11 | q11 | +/+ | ENSP00000249440 | HOXD3 | q11 | q11 | 917 | 2 | 176742089 | 176745245 | 1 | 2 | 100% | 99.8% | 1305 | 0.56 / 0.01 = 56 | within_species_paralog: HOXD3 | 1 |
| p12 | q12 | +/+ | ENSP00000328598 | HOXD1 | q12 | q12 | 631 | 2 | 176761776 | 176763113 | 1 | 2 | 100% | 93.1% | 1005 | 0.79 / 0.01 = 79 | noComparaHits: HOXD1 | 1 |
| p13 | q13 | +/+ | ENSP00000346209 | - | q13 | q13 | 227 | 2 | 176773885 | 176774226 | -1 | 1 | 100% | 100% | 342 | 0.15 / 0.01 = 15 | within_species_paralog: ENSG00000182553 | 1 |
| p14 | q14 | +/+ | ENSP00000249442 | MTX2 | q14 | q14 | 455 | 2 | 176896354 | 176910635 | 1 | 6 | 79.8% | 97.7% | 645 | 0.55 / 0.01 = 55 | noComparaHits: MTX2 | 1 |
region 2 (region 1 for Mmus)
 |
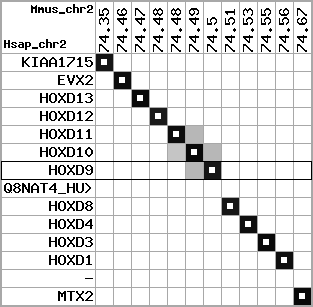 |
 |
retrieve target region: getEnsemblProteins.pl -h Mus_musculus -l 2 -i 74322901:74677310 -f 27e4 some_geneID_to_be_selected
scoreRatio_sum=182.96 log_scoreRatio_sum=25.99 focal_scoreRatio_sum=53.31
AlnScore=10726 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Mus_musculus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 649.1 | 2 | 74322901 | 74369970 | -1 | 8 | 86.2% | 81.8% | 1134 | 1 / 0.17 = 5.88 | ortholog_one2one: Lnp | 1 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 746 | 2 | 74456458 | 74460258 | -1 | 3 | 100% | 79.9% | 1434 | 0.62 / 0.08 = 7.75 | ortholog_one2one: Evx2 | 1 |
| p3 | q3 | +/+ | ENSP00000249505 | HOXD13 | q3 | q3 | 616 | 2 | 74469173 | 74470972 | 1 | 2 | 100% | 86.6% | 1041 | 0.61 / 0.04 = 15.25 | ortholog_one2one: Hoxd13 | 1 |
| p4 | q4 | +/+ | ENSP00000303578 | HOXD12 | q4 | q4 | 445 | 2 | 74475926 | 74476888 | 1 | 1 | 100% | 72.4% | 963 | 0.66 / 0.2 = 3.3 | ortholog_one2one: Hoxd12 | 1 |
| p5 | q5 | +/+ | ENSP00000249504 | HOXD11 | q5 | q5 | 503 | 2 | 74483232 | 74484974 | 1 | 2 | 97% | 75.9% | 990 | 0.53 / 0.09 = 5.89 | ortholog_one2one: Hoxd11 | 1 |
| p6 | q6 | +/+ | ENSP00000249501 | HOXD10 | q6 | q6 | 685 | 2 | 74492819 | 74495204 | 1 | 2 | 100% | 98.8% | 1020 | 0.58 / 0.01 = 58 | ortholog_one2one: Hoxd10 | 1 |
| p7 | q7 | +/+ | ENSP00000249499 | HOXD9 | q7 | q7 | 556 | 2 | 74498895 | 74500257 | 1 | 2 | 100% | 75.6% | 1119 | 0.6 / 0.18 = 3.33 | ortholog_one2one: Hoxd9 | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q8 | +/+ | ENSP00000315949 | HOXD8 | q8 | q8 | 450 | 2 | 74506387 | 74507636 | 1 | 2 | 99% | 77.1% | 855 | 0.49 / 0.17 = 2.88 | ortholog_one2one: Hoxd8 | 1 |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 494 | 2 | 74528118 | 74529423 | 1 | 2 | 100% | 89.1% | 777 | 0.51 / 0.1 = 5.1 | noComparaHits: Hoxd3 | 1 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 880 | 2 | 74544851 | 74547915 | 1 | 2 | 100% | 94.6% | 1323 | 0.56 / 0.04 = 14 | ortholog_one2one: Hoxd3 | 1 |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 554 | 2 | 74563941 | 74565225 | 1 | 2 | 100% | 81.4% | 1002 | 0.79 / 0.12 = 6.58 | ortholog_one2one: Hoxd1 | 1 |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q12 | +/+ | ENSP00000249442 | MTX2 | q12 | q12 | 446 | 2 | 74660229 | 74677310 | 1 | 6 | 80.2% | 87.8% | 702 | 0.55 / 0.01 = 55 | ortholog_one2one: Mtx2 | 1 |
region 3 (region 1 for Mmul)
 |
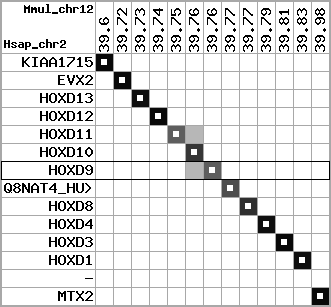 |
 |
retrieve target region: getEnsemblProteins.pl -h Macaca_mulatta -l 12 -i 39563563:39986152 -f 32e4 some_geneID_to_be_selected
scoreRatio_sum=264.58 log_scoreRatio_sum=26.1 focal_scoreRatio_sum=48.75
AlnScore=10283 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Macaca_mulatta
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 765.2 | 12 | 39563563 | 39631521 | -1 | 8 | 87.1% | 95.8% | 1131 | 1 / 0.03 = 33.33 | ortholog_one2one: KIAA1715 | 1 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 814 | 12 | 39720761 | 39724430 | -1 | 3 | 100% | 87.1% | 1434 | 0.62 / 0.01 = 62 | ortholog_one2one: EVX2 | 1 |
| p3 | q3 | +/+ | ENSP00000249505 | HOXD13 | q3 | q3 | 564 | 12 | 39733939 | 39735562 | 1 | 2 | 81.2% | 99.6% | 819 | 0.61 / 0.12 = 5.08 | ortholog_one2one: HOXD13 | 1 |
| p4 | q4 | +/+ | ENSP00000303578 | HOXD12 | q4 | q4 | 543 | 12 | 39740821 | 39741776 | 1 | 2 | 100% | 91.7% | 897 | 0.66 / 0.02 = 33 | ortholog_one2one: HOXD12 | 1 |
| p5 | q5 | +/+ | ENSP00000249504 | HOXD11 | q5 | q5 | 255 | 12 | 39748740 | 39750377 | 1 | 2 | 35.8% | 98.3% | 363 | 0.53 / 0.54 = 0.98 | noComparaHits: | 2 |
| p6 | q6 | +/+ | ENSP00000249501 | HOXD10 | q6 | q6 | 420 | 12 | 39758095 | 39760422 | 1 | 2 | 59.7% | 100% | 609 | 0.58 / 0.39 = 1.49 | ortholog_one2one: HOXD10 | 1 |
| p7 | q7 | +/+ | ENSP00000249499 | HOXD9 | q7 | q7 | 303 | 12 | 39764093 | 39764701 | 1 | 1 | 58.5% | 76% | 609 | 0.6 / 0.55 = 1.09 | noComparaHits: | 1 |
| p8 | q8 | +/+ | ENSP00000316098 | Q8NAT4_HUMAN | q8 | q8 | 159 | 12 | 39770022 | 39770417 | -1 | 1 | 81.8% | 65.7% | 396 | 1 / 0.47 = 2.13 | noComparaHits: ENSMMUG00000023505 | 1 |
| p9 | q9 | +/+ | ENSP00000315949 | HOXD8 | q9 | q9 | 361 | 12 | 39772034 | 39772943 | 1 | 2 | 59% | 99.4% | 513 | 0.49 / 0.33 = 1.48 | ortholog_one2one: HOXD8 | 1 |
| p10 | q10 | +/+ | ENSP00000302548 | HOXD4 | q10 | q10 | 494 | 12 | 39793366 | 39794671 | 1 | 2 | 100% | 90.2% | 768 | 0.51 / 0.1 = 5.1 | ortholog_one2one: HOXD4 | 1 |
| p11 | q11 | +/+ | ENSP00000249440 | HOXD3 | q11 | q11 | 912 | 12 | 39810758 | 39813914 | 1 | 2 | 100% | 97.3% | 1335 | 0.56 / 0.01 = 56 | ortholog_one2one: HOXD3 | 1 |
| p12 | q12 | +/+ | ENSP00000328598 | HOXD1 | q12 | q12 | 566 | 12 | 39830419 | 39831755 | 1 | 2 | 100% | 84.6% | 996 | 0.79 / 0.1 = 7.9 | ortholog_one2one: HOXD1 | 1 |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q13 | +/+ | ENSP00000249442 | MTX2 | q13 | q13 | 452.3 | 12 | 39968889 | 39986152 | 1 | 6 | 79.8% | 97.2% | 645 | 0.55 / 0.01 = 55 | ortholog_one2one: MTX2 | 1 |
region 4 (region 1 for Mdom)
 |
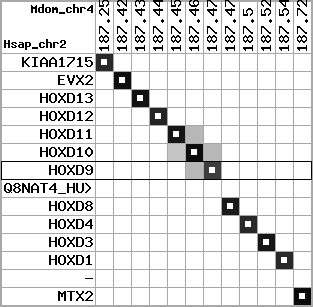 |
 |
retrieve target region: getEnsemblProteins.pl -h Monodelphis_domestica -l 4 -i 187208613:187739147 -f 40e4 some_geneID_to_be_selected
scoreRatio_sum=52.54 log_scoreRatio_sum=14.91 focal_scoreRatio_sum=13.97
AlnScore=9379 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Monodelphis_domestica
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 541.3 | 4 | 187208613 | 187285683 | -1 | 6 | 85.3% | 68.1% | 1182 | 1 / 0.31 = 3.23 | ortholog_one2many: ENSMODG00000009536 | 1 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 707.6 | 4 | 187417681 | 187421910 | -1 | 4 | 83.8% | 86.3% | 1218 | 0.62 / 0.13 = 4.77 | ortholog_one2one: EVX2 | 1 |
| p3 | q3 | +/+ | ENSP00000249505 | HOXD13 | q3 | q3 | 562 | 4 | 187431202 | 187433173 | 1 | 2 | 100% | 82.1% | 999 | 0.61 / 0.13 = 4.69 | ortholog_one2one: HOXD13 | 1 |
| p4 | q4 | +/+ | ENSP00000303578 | HOXD12 | q4 | q4 | 403 | 4 | 187439427 | 187440514 | 1 | 2 | 98.9% | 73.8% | 816 | 0.66 / 0.27 = 2.44 | ortholog_one2one: HOXD12 | 1 |
| p5 | q5 | +/+ | ENSP00000249504 | HOXD11 | q5 | q5 | 447 | 4 | 187447938 | 187449659 | 1 | 2 | 100% | 67.1% | 915 | 0.53 / 0.19 = 2.79 | ortholog_one2one: HOXD11 | 1 |
| p6 | q6 | +/+ | ENSP00000249501 | HOXD10 | q6 | q6 | 645 | 4 | 187458195 | 187460683 | 1 | 2 | 100% | 92.6% | 1020 | 0.58 / 0.06 = 9.67 | ortholog_one2one: HOXD10 | 1 |
| p7 | q7 | +/+ | ENSP00000249499 | HOXD9 | q7 | q7 | 389 | 4 | 187464529 | 187465949 | 1 | 2 | 95.9% | 61.6% | 993 | 0.6 / 0.42 = 1.43 | ortholog_one2one: HOXD9 | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q8 | +/+ | ENSP00000315949 | HOXD8 | q8 | q8 | 422 | 4 | 187472546 | 187473913 | 1 | 2 | 100% | 71.3% | 921 | 0.49 / 0.22 = 2.23 | ortholog_one2one: HOXD8 | 1 |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 385 | 4 | 187497377 | 187498661 | 1 | 2 | 100% | 74.2% | 714 | 0.51 / 0.3 = 1.7 | ortholog_one2one: HOXD4 | 1 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 759 | 4 | 187515814 | 187518908 | 1 | 2 | 100% | 83.5% | 1299 | 0.56 / 0.17 = 3.29 | ortholog_one2one: HOXD3 | 1 |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 431 | 4 | 187537162 | 187538614 | 1 | 2 | 100% | 65.2% | 1014 | 0.79 / 0.31 = 2.55 | ortholog_one2one: HOXD1 | 1 |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q12 | +/+ | ENSP00000249442 | MTX2 | q12 | q12 | 434.6 | 4 | 187709797 | 187739147 | 1 | 6 | 78.7% | 89.7% | 672 | 0.55 / 0.04 = 13.75 | ortholog_one2one: MTX2 | 1 |
region 5 (region 1 for Cfam)
 |
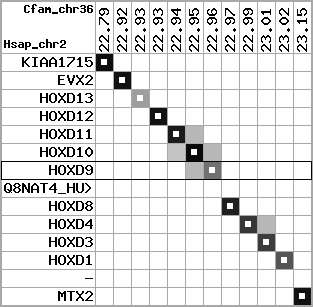 |
 |
retrieve target region: getEnsemblProteins.pl -h Canis_familiaris -l 36 -i 22759346:23161054 -f 30e4 some_geneID_to_be_selected
scoreRatio_sum=89.55 log_scoreRatio_sum=12.88 focal_scoreRatio_sum=36.02
AlnScore=8278 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Canis_familiaris
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 692.8 | 36 | 22759346 | 22826897 | -1 | 7 | 86.9% | 81% | 1245 | 1 / 0.12 = 8.33 | ortholog_one2one: 478809 | 1 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 681 | 36 | 22914522 | 22918242 | -1 | 3 | 100% | 69.1% | 1584 | 0.62 / 0.16 = 3.88 | ortholog_one2one: EVX2 | 1 |
| p3 | q3 | +/+ | ENSP00000249505 | HOXD13 | q3 | q3 | 172 | 36 | 22928994 | 22929335 | 1 | 1 | 34.3% | 77.4% | 342 | 0.61 / 0.73 = 0.84 | noComparaHits: | 3 |
| p4 | q4 | +/+ | ENSP00000303578 | HOXD12 | q4 | q4 | 478 | 36 | 22934436 | 22935399 | 1 | 2 | 100% | 84.5% | 849 | 0.66 / 0.14 = 4.71 | ortholog_one2one: HOXD12 | 1 |
| p5 | q5 | +/+ | ENSP00000249504 | HOXD11 | q5 | q5 | 439 | 36 | 22941982 | 22943757 | 1 | 2 | 100% | 64.1% | 1095 | 0.53 / 0.21 = 2.52 | ortholog_one2one: HOXD11 | 1 |
| p6 | q6 | +/+ | ENSP00000249501 | HOXD10 | q6 | q6 | 693 | 36 | 22951611 | 22954001 | 1 | 2 | 100% | 100% | 1020 | 0.58 / 0.01 = 58 | ortholog_one2one: 488415 | 1 |
| p7 | q7 | +/+ | ENSP00000249499 | HOXD9 | q7 | q7 | 268 | 36 | 22958266 | 22959115 | 1 | 2 | 48% | 73.9% | 540 | 0.6 / 0.6 = 1 | noComparaHits: ENSCAFG00000023404 | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q8 | +/+ | ENSP00000315949 | HOXD8 | q8 | q8 | 404 | 36 | 22965937 | 22966947 | 1 | 2 | 68.6% | 94.5% | 597 | 0.49 / 0.26 = 1.88 | ortholog_one2one: HOXD8 | 1 |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 333 | 36 | 22987182 | 22988389 | 1 | 2 | 82.4% | 76.4% | 615 | 0.51 / 0.39 = 1.31 | ortholog_one2one: HOXD4 | 1 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 531 | 36 | 23006364 | 23007248 | 1 | 1 | 71.5% | 85.2% | 885 | 0.56 / 0.42 = 1.33 | ortholog_one2one: HOXD3 | 1 |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 302 | 36 | 23022458 | 23023340 | 1 | 2 | 69.8% | 69.7% | 675 | 0.79 / 0.52 = 1.52 | noComparaHits: | 1 |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q12 | +/+ | ENSP00000249442 | MTX2 | q12 | q12 | 394.3 | 36 | 23143436 | 23161054 | 1 | 5 | 79.5% | 87.7% | 618 | 0.55 / 0.13 = 4.23 | ortholog_one2one: 478811 | 1 |
region 6 (region 1 for Btau)
 |
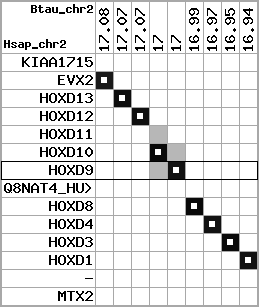 |
 |
retrieve target region: getEnsemblProteins.pl -h Bos_taurus -l 2 -i 16934939:17086630 -f 11e4 some_geneID_to_be_selected
scoreRatio_sum=155.18 log_scoreRatio_sum=20.14 focal_scoreRatio_sum=58.36
AlnScore=8093 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Bos_taurus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 630.7 | 2 | 17082934 | 17086630 | 1 | 4 | 77.3% | 85.5% | 1107 | 0.62 / 0.22 = 2.82 | ortholog_one2one: EVX2 | 1 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 596 | 2 | 17072582 | 17074328 | -1 | 2 | 96.1% | 90.7% | 966 | 0.61 / 0.08 = 7.63 | ortholog_one2one: HOXD13 | 1 |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 490 | 2 | 17066540 | 17067497 | -1 | 2 | 100% | 84.6% | 873 | 0.66 / 0.12 = 5.5 | ortholog_one2one: HOXD12 | 1 |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | q4 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 693 | 2 | 17003673 | 17006048 | -1 | 2 | 100% | 94% | 1092 | 0.58 / 0.01 = 58 | ortholog_one2one: HOXD10 | 1 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 577 | 2 | 16998544 | 16999927 | -1 | 2 | 100% | 82.1% | 1050 | 0.6 / 0.15 = 4 | ortholog_one2one: HOXD9 | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 546 | 2 | 16991108 | 16992329 | -1 | 2 | 100% | 84.3% | 930 | 0.49 / 0.01 = 49 | ortholog_one2one: HOXD8 | 1 |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 477 | 2 | 16969446 | 16970762 | -1 | 2 | 100% | 86.5% | 777 | 0.51 / 0.13 = 3.92 | ortholog_one2one: HOXD4 | 1 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 883 | 2 | 16951347 | 16954688 | -1 | 2 | 100% | 95% | 1314 | 0.56 / 0.03 = 18.67 | ortholog_one2one: HOXD3 | 1 |
| p12 | q9 | +/+ | ENSP00000328598 | HOXD1 | q9 | q9 | 537 | 2 | 16934939 | 16936242 | -1 | 2 | 100% | 80.9% | 987 | 0.79 / 0.14 = 5.64 | ortholog_one2one: HOXD1 | 1 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 7 (region 1 for Xtro)
 |
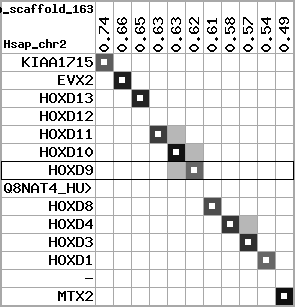 |
 |
retrieve target region: getEnsemblProteins.pl -h Xenopus_tropicalis -l scaffold_163 -i 476364:744115 -f 20e4 some_geneID_to_be_selected
scoreRatio_sum=23.5 log_scoreRatio_sum=6.81 focal_scoreRatio_sum=6.56
AlnScore=6880 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Xenopus_tropicalis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 360.5 | scaffold_163 | 728459 | 744115 | 1 | 5 | 54.7% | 67.5% | 726 | 1 / 0.54 = 1.85 | ortholog_one2one: TEgg059c18.1 | 1 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 621 | scaffold_163 | 659972 | 664035 | 1 | 3 | 98.9% | 69.6% | 1269 | 0.62 / 0.23 = 2.7 | ortholog_one2one: EVX2 | 1 |
| p3 | q3 | +/+ | ENSP00000249505 | HOXD13 | q3 | q3 | 457 | scaffold_163 | 649601 | 652611 | -1 | 2 | 100% | 69.1% | 894 | 0.61 / 0.29 = 2.1 | ortholog_one2one: HOXD13 | 1 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 327 | scaffold_163 | 633881 | 635453 | -1 | 2 | 95.3% | 56.3% | 825 | 0.53 / 0.41 = 1.29 | noComparaHits: | 1 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 592 | scaffold_163 | 624911 | 627904 | -1 | 2 | 100% | 85.6% | 1014 | 0.58 / 0.14 = 4.14 | ortholog_one2one: HOXD10 | 1 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 293 | scaffold_163 | 619634 | 620793 | -1 | 2 | 94.7% | 52.8% | 765 | 0.6 / 0.56 = 1.07 | ortholog_one2one: HOXD9 | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q7 | +/+ | ENSP00000315949 | HOXD8 | q7 | q7 | 282 | scaffold_163 | 608268 | 609787 | -1 | 2 | 95.5% | 54.9% | 657 | 0.49 / 0.48 = 1.02 | ortholog_one2one: HOXD8 | 1 |
| p10 | q8 | +/+ | ENSP00000302548 | HOXD4 | q8 | q8 | 337 | scaffold_163 | 582346 | 583439 | -1 | 2 | 100% | 63.7% | 777 | 0.51 / 0.38 = 1.34 | between_species_paralog: HOXD4 | 1 |
| p11 | q9 | +/+ | ENSP00000249440 | HOXD3 | q9 | q9 | 594 | scaffold_163 | 567719 | 569361 | -1 | 2 | 100% | 68.2% | 1248 | 0.56 / 0.35 = 1.6 | ortholog_one2one: HOXD3 | 1 |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 271 | scaffold_163 | 534709 | 536091 | -1 | 2 | 99.4% | 48.6% | 906 | 0.79 / 0.57 = 1.39 | ortholog_one2one: hoxd1 | 1 |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q11 | +/+ | ENSP00000249442 | MTX2 | q11 | q11 | 404.2 | scaffold_163 | 476364 | 496298 | -1 | 6 | 78.7% | 84.1% | 642 | 0.55 / 0.11 = 5 | ortholog_one2one: mtx2 | 1 |
region 8 (region 1 for Ggal)
 |
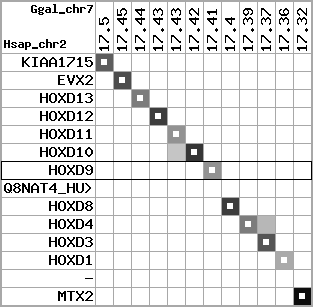 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l 7 -i 17315309:17512501 -f 15e4 some_geneID_to_be_selected
scoreRatio_sum=19.22 log_scoreRatio_sum=3.28 focal_scoreRatio_sum=4.76
AlnScore=5683 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 350 | 7 | 17486635 | 17512501 | 1 | 4 | 48.1% | 77.4% | 636 | 1 / 0.55 = 1.82 | ortholog_one2one: 424141 | 1 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 419.8 | 7 | 17444164 | 17447372 | 1 | 3 | 69.5% | 69.3% | 897 | 0.62 / 0.48 = 1.29 | ortholog_one2one: EVX2 | 1 |
| p3 | q3 | +/+ | ENSP00000249505 | HOXD13 | q3 | q3 | 221.8 | 7 | 17438755 | 17439670 | -1 | 2 | 32.8% | 93.7% | 333 | 0.61 / 0.65 = 0.94 | ortholog_one2one: HXD13_CHICK | 3 |
| p4 | q4 | +/+ | ENSP00000303578 | HOXD12 | q4 | q4 | 332 | 7 | 17431970 | 17433038 | -1 | 2 | 97.5% | 64.1% | 807 | 0.66 / 0.4 = 1.65 | ortholog_one2one: HXD12_CHICK | 1 |
| p5 | q5 | +/+ | ENSP00000249504 | HOXD11 | q5 | q5 | 160 | 7 | 17425088 | 17425324 | -1 | 1 | 23.4% | 96.2% | 237 | 0.53 / 0.71 = 0.75 | ortholog_one2one: NP_989951.1 | 2 |
| p6 | q6 | +/+ | ENSP00000249501 | HOXD10 | q6 | q6 | 439 | 7 | 17416029 | 17416769 | -1 | 1 | 72.9% | 88.4% | 741 | 0.58 / 0.36 = 1.61 | ortholog_one2one: NP_997060.1 | 1 |
| p7 | q7 | +/+ | ENSP00000249499 | HOXD9 | q7 | q7 | 198 | 7 | 17409077 | 17409745 | -1 | 1 | 76.9% | 46.2% | 669 | 0.6 / 0.7 = 0.86 | noComparaHits: NP_997060.1 | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q8 | +/+ | ENSP00000315949 | HOXD8 | q8 | q8 | 323 | 7 | 17402517 | 17404435 | -1 | 2 | 57.9% | 88.7% | 504 | 0.49 / 0.4 = 1.23 | noComparaHits: NP_997060.1 | 1 |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 196 | 7 | 17386750 | 17387178 | -1 | 1 | 61.2% | 67.5% | 429 | 0.51 / 0.64 = 0.8 | ortholog_one2one: HXD4_CHICK | 1 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 457 | 7 | 17369712 | 17370758 | -1 | 1 | 82.4% | 64.6% | 1047 | 0.56 / 0.5 = 1.12 | ortholog_one2one: HOXD3 | 1 |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 147.4 | 7 | 17361528 | 17362441 | -1 | 2 | 74.7% | 37% | 810 | 0.79 / 0.76 = 1.04 | noComparaHits: HM1_CHICK | 1 |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q12 | +/+ | ENSP00000249442 | MTX2 | q12 | q12 | 412 | 7 | 17315309 | 17322161 | -1 | 6 | 80.2% | 83.4% | 687 | 0.55 / 0.09 = 6.11 | ortholog_one2one: 424138 | 1 |
region 9 (region 1 for Drer)
 |
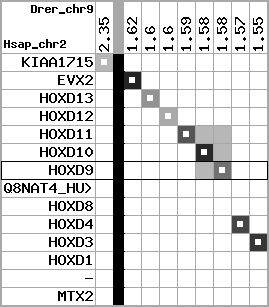 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 9 -i 1553343:2354366 -f 60e4 some_geneID_to_be_selected
scoreRatio_sum=11.75 log_scoreRatio_sum=1.89 focal_scoreRatio_sum=3.79
AlnScore=4111 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 144.4 | 9 | 2338539 | 2354366 | 1 | 2 | 23.1% | 60.6% | 297 | 1 / 0.81 = 1.23 | ortholog_one2many: ENSDARG00000063646 | 2 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 588 | 9 | 1618717 | 1621062 | 1 | 3 | 100% | 66.7% | 1260 | 0.62 / 0.27 = 2.3 | ortholog_one2many: evx2 | 1 |
| p3 | q3 | +/+ | ENSP00000249505 | HOXD13 | q3 | q3 | 180.3 | 9 | 1603330 | 1605831 | -1 | 2 | 65.1% | 41.9% | 657 | 0.61 / 0.72 = 0.85 | noComparaHits: hoxd13a | 6 |
| p4 | q4 | +/+ | ENSP00000303578 | HOXD12 | q4 | q4 | 119 | 9 | 1597990 | 1598232 | -1 | 1 | 29.1% | 70.4% | 243 | 0.66 / 0.78 = 0.85 | noComparaHits: hoxd12a | 2 |
| p5 | q5 | +/+ | ENSP00000249504 | HOXD11 | q5 | q5 | 271 | 9 | 1591957 | 1593647 | -1 | 2 | 100% | 46.5% | 846 | 0.53 / 0.51 = 1.04 | between_species_paralog: hoxd11a | 1 |
| p6 | q6 | +/+ | ENSP00000249501 | HOXD10 | q6 | q6 | 472 | 9 | 1583877 | 1585587 | -1 | 2 | 99.7% | 69.7% | 996 | 0.58 / 0.31 = 1.87 | ortholog_one2many: hoxd10a | 1 |
| p7 | q7 | +/+ | ENSP00000249499 | HOXD9 | q7 | q7 | 264 | 9 | 1580195 | 1581290 | -1 | 2 | 99.4% | 47.9% | 780 | 0.6 / 0.61 = 0.98 | ortholog_one2many: hoxd9a | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q8 | +/+ | ENSP00000302548 | HOXD4 | q8 | q8 | 305 | 9 | 1565271 | 1566370 | -1 | 2 | 100% | 60.9% | 711 | 0.51 / 0.44 = 1.16 | ortholog_one2one: hoxd4a | 1 |
| p11 | q9 | +/+ | ENSP00000249440 | HOXD3 | q9 | q9 | 562 | 9 | 1553343 | 1554792 | -1 | 2 | 100% | 65.1% | 1242 | 0.56 / 0.38 = 1.47 | noComparaHits: ENSDARG00000059280
; hoxd3a |
1 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 10 (region 1 for Trub)
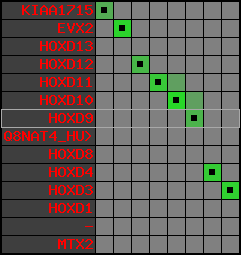 |
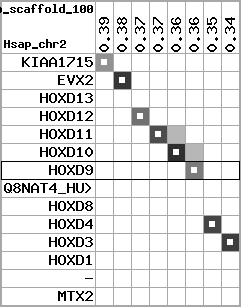 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_100 -i 339085:394942 -f 42e3 some_geneID_to_be_selected
scoreRatio_sum=10.4 log_scoreRatio_sum=1.93 focal_scoreRatio_sum=3.46
AlnScore=3930 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 223.8 | scaffold_100 | 390711 | 394942 | 1 | 3 | 40.7% | 49.1% | 666 | 1 / 0.71 = 1.41 | ortholog_one2one: NEWSINFRUG00000143408 | 2 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 513 | scaffold_100 | 378378 | 380283 | 1 | 3 | 97.9% | 60.8% | 1248 | 0.62 / 0.37 = 1.68 | ortholog_one2one: NEWSINFRUG00000158421 | 1 |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 205.5 | scaffold_100 | 371715 | 372790 | -1 | 2 | 73.7% | 54.2% | 630 | 0.66 / 0.63 = 1.05 | noComparaHits: NEWSINFRUG00000120943 | 1 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 280 | scaffold_100 | 366031 | 367381 | -1 | 2 | 100% | 47.7% | 858 | 0.53 / 0.49 = 1.08 | between_species_paralog: NEWSINFRUG00000162044 | 1 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 451 | scaffold_100 | 361352 | 362879 | -1 | 2 | 99.1% | 68.2% | 999 | 0.58 / 0.34 = 1.71 | ortholog_one2one: NEWSINFRUG00000124778 | 1 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 244.6 | scaffold_100 | 358386 | 359535 | -1 | 2 | 68.4% | 55.6% | 621 | 0.6 / 0.64 = 0.94 | ortholog_one2one: NEWSINFRUG00000124776 | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 308 | scaffold_100 | 348414 | 349464 | -1 | 2 | 100% | 62.2% | 699 | 0.51 / 0.44 = 1.16 | ortholog_one2one: NEWSINFRUG00000124775 | 1 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 536 | scaffold_100 | 339085 | 340364 | -1 | 2 | 96.3% | 63.8% | 1227 | 0.56 / 0.41 = 1.37 | ortholog_one2one: NEWSINFRUG00000124774 | 1 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 11 (region 1 for Gacu)
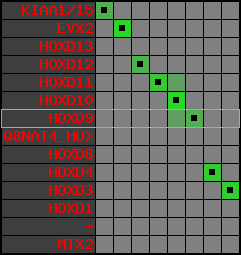 |
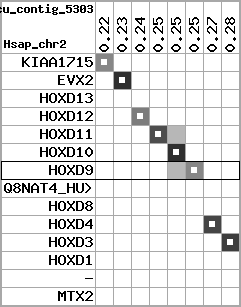 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_5303 -i 214375:277218 -f 47e3 some_geneID_to_be_selected
scoreRatio_sum=10.57 log_scoreRatio_sum=1.99 focal_scoreRatio_sum=3.42
AlnScore=3922 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 246.5 | contig_5303 | 214375 | 217990 | -1 | 3 | 39.7% | 50% | 711 | 1 / 0.68 = 1.47 | noComparaHits: ENSGACG00000004584 | 2 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 539 | contig_5303 | 229470 | 231529 | -1 | 3 | 99.6% | 61.7% | 1257 | 0.62 / 0.33 = 1.88 | noComparaHits: ENSGACG00000004579 | 1 |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 189.6 | contig_5303 | 239842 | 241010 | 1 | 2 | 59.7% | 56.5% | 510 | 0.66 / 0.66 = 1 | noComparaHits: ENSGACG00000004574 | 1 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 280 | contig_5303 | 245547 | 246912 | 1 | 2 | 89.6% | 51.3% | 783 | 0.53 / 0.49 = 1.08 | noComparaHits: ENSGACG00000004569 | 1 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 445 | contig_5303 | 250305 | 252028 | 1 | 2 | 98.5% | 67.8% | 1005 | 0.58 / 0.35 = 1.66 | noComparaHits: ENSGACG00000004564 | 1 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 217.9 | contig_5303 | 254123 | 255324 | 1 | 2 | 74.6% | 49.2% | 687 | 0.6 / 0.68 = 0.88 | noComparaHits: Q4VQD4_GASAC | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 304 | contig_5303 | 266163 | 267256 | 1 | 2 | 100% | 61.2% | 702 | 0.51 / 0.44 = 1.16 | noComparaHits: ENSGACG00000004551 | 1 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 551 | contig_5303 | 275876 | 277218 | 1 | 2 | 96.3% | 65.7% | 1218 | 0.56 / 0.39 = 1.44 | noComparaHits: ENSGACG00000004548 | 1 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 12 (region 2 for Gacu)
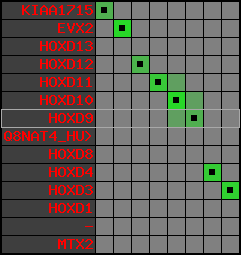 |
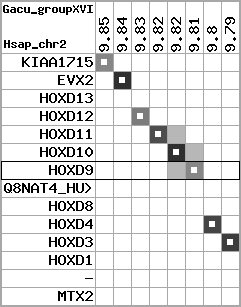 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l groupXVI -i 9792262:9855105 -f 47e3 some_geneID_to_be_selected
scoreRatio_sum=10.57 log_scoreRatio_sum=1.99 focal_scoreRatio_sum=3.42
AlnScore=3922 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 246.5 | groupXVI | 9851490 | 9855105 | 1 | 3 | 39.7% | 50% | 711 | 1 / 0.68 = 1.47 | ortholog_one2many: ENSGACG00000004584 | 2 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 539 | groupXVI | 9837951 | 9840010 | 1 | 3 | 99.6% | 61.7% | 1257 | 0.62 / 0.33 = 1.88 | ortholog_one2one: ENSGACG00000004579 | 1 |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 189.6 | groupXVI | 9828470 | 9829638 | -1 | 2 | 59.7% | 56.5% | 510 | 0.66 / 0.66 = 1 | noComparaHits: ENSGACG00000004574 | 1 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 280 | groupXVI | 9822568 | 9823933 | -1 | 2 | 89.6% | 51.3% | 783 | 0.53 / 0.49 = 1.08 | between_species_paralog: ENSGACG00000004569 | 1 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 445 | groupXVI | 9817452 | 9819175 | -1 | 2 | 98.5% | 67.8% | 1005 | 0.58 / 0.35 = 1.66 | ortholog_one2one: ENSGACG00000004564 | 1 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 217.9 | groupXVI | 9814156 | 9815357 | -1 | 2 | 74.6% | 49.2% | 687 | 0.6 / 0.68 = 0.88 | ortholog_one2one: Q4VQD4_GASAC | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 304 | groupXVI | 9802224 | 9803317 | -1 | 2 | 100% | 61.2% | 702 | 0.51 / 0.44 = 1.16 | ortholog_one2one: ENSGACG00000004551 | 1 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 551 | groupXVI | 9792262 | 9793604 | -1 | 2 | 96.3% | 65.7% | 1218 | 0.56 / 0.39 = 1.44 | ortholog_one2one: ENSGACG00000004548 | 1 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 13 (region 1 for Olat)
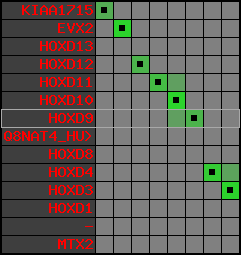 |
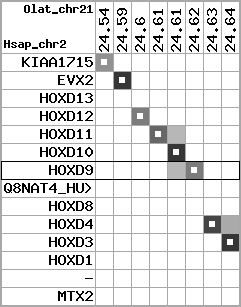 |
 |
retrieve target region: getEnsemblProteins.pl -h Oryzias_latipes -l 21 -i 24538670:24637174 -f 74e3 some_geneID_to_be_selected
scoreRatio_sum=10.12 log_scoreRatio_sum=1.68 focal_scoreRatio_sum=3.32
AlnScore=3778 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Oryzias_latipes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 217.6 | 21 | 24538670 | 24543652 | -1 | 3 | 38.3% | 58.5% | 474 | 1 / 0.72 = 1.39 | ortholog_one2many: ENSORLG00000017531 | 2 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 514 | 21 | 24590614 | 24592238 | -1 | 3 | 75.8% | 74% | 1014 | 0.62 / 0.36 = 1.72 | ortholog_one2one: Q2WFR6_ORYLA | 1 |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 191.1 | 21 | 24601819 | 24602905 | 1 | 2 | 57.6% | 57.9% | 492 | 0.66 / 0.65 = 1.02 | noComparaHits: Q3V5Z4_ORYLA | 1 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 225.2 | 21 | 24606914 | 24608290 | 1 | 2 | 68.3% | 52.4% | 564 | 0.53 / 0.59 = 0.9 | between_species_paralog: Q3V5Z5_ORYLA | 3 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 418 | 21 | 24611625 | 24613192 | 1 | 2 | 100% | 63.4% | 1044 | 0.58 / 0.39 = 1.49 | ortholog_one2one: Q9PVQ4_ORYLA | 1 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 243 | 21 | 24614922 | 24616093 | 1 | 2 | 74.6% | 50% | 717 | 0.6 / 0.64 = 0.94 | ortholog_one2one: HXD9_ORYLA | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 310 | 21 | 24626722 | 24627802 | 1 | 2 | 100% | 61.6% | 708 | 0.51 / 0.43 = 1.19 | ortholog_one2one: Q3V5Z8_ORYLA | 1 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 560 | 21 | 24635889 | 24637174 | 1 | 2 | 96.3% | 66.7% | 1203 | 0.56 / 0.38 = 1.47 | ortholog_one2one: Q3V5Z9_ORYLA | 1 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 14 (region 1 for Tnig)
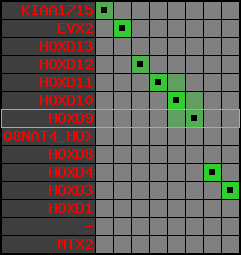 |
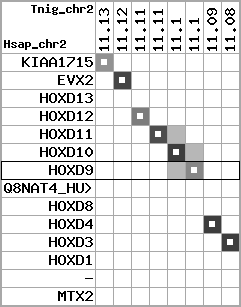 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l 2 -i 11075386:11132794 -f 43e3 some_geneID_to_be_selected
scoreRatio_sum=9.8 log_scoreRatio_sum=1.51 focal_scoreRatio_sum=3.22
AlnScore=3743 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 217.2 | 2 | 11128959 | 11132794 | 1 | 3 | 49.5% | 43% | 771 | 1 / 0.72 = 1.39 | ortholog_one2one: GSTENG00031457001 | 1 |
| p2 | q2 | +/+ | ENSP00000312385 | EVX2 | q2 | q2 | 447 | 2 | 11117044 | 11118889 | 1 | 3 | 92.4% | 55.9% | 1194 | 0.62 / 0.45 = 1.38 | ortholog_one2many: HOXD-EVX
; GSTENG00031458001 |
1 |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 197.1 | 2 | 11110368 | 11111376 | -1 | 2 | 68.7% | 53.2% | 609 | 0.66 / 0.64 = 1.03 | noComparaHits: GSTENG00031459001
; HOXD12 ; GSTENG00031460001 |
1 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 285 | 2 | 11104973 | 11106367 | -1 | 2 | 93.8% | 51.4% | 765 | 0.53 / 0.48 = 1.1 | between_species_paralog: HOXD11 | 1 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 414 | 2 | 11100577 | 11102062 | -1 | 2 | 99.1% | 64.4% | 993 | 0.58 / 0.4 = 1.45 | ortholog_one2many: GSTENG00031461001
; HOXD10 |
1 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 207 | 2 | 11097829 | 11098947 | -1 | 1 | 98.5% | 39.1% | 1119 | 0.6 / 0.69 = 0.87 | ortholog_one2one: GSTENG00031462001 | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 316.1 | 2 | 11087917 | 11088977 | -1 | 3 | 100% | 58.1% | 774 | 0.51 / 0.42 = 1.21 | between_species_paralog: GSTENG00031463001
; HOXD4 |
1 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 539 | 2 | 11075386 | 11076667 | -1 | 2 | 96.3% | 66% | 1185 | 0.56 / 0.41 = 1.37 | ortholog_one2many: GSTENG00031464001
; HOXD3 |
1 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 15 (region 2 for Xtro)
 |
 |
 |
retrieve target region: getEnsemblProteins.pl -h Xenopus_tropicalis -l scaffold_56 -i 1323527:1481190 -f 12e4 some_geneID_to_be_selected
scoreRatio_sum=8.63 log_scoreRatio_sum=-0.47 focal_scoreRatio_sum=2.98
AlnScore=3343 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Xenopus_tropicalis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 312 | scaffold_56 | 1323527 | 1325018 | -1 | 2 | 66.8% | 52.4% | 747 | 0.62 / 0.61 = 1.02 | between_species_paralog: EVX1 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 278 | scaffold_56 | 1384955 | 1386296 | 1 | 2 | 76.1% | 57% | 735 | 0.61 / 0.57 = 1.07 | between_species_paralog: HOXA13 | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 258 | scaffold_56 | 1397328 | 1399142 | 1 | 2 | 79% | 53.9% | 684 | 0.53 / 0.53 = 1 | between_species_paralog: HOXA11 | 2 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 282 | scaffold_56 | 1407366 | 1409254 | 1 | 2 | 98.8% | 48.7% | 1032 | 0.58 / 0.59 = 0.98 | noComparaHits: HOXA10 | 3 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 249 | scaffold_56 | 1415310 | 1416623 | 1 | 2 | 68.4% | 55.2% | 636 | 0.6 / 0.63 = 0.95 | noComparaHits: ENSXETG00000000724 | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 114 | scaffold_56 | 1423126 | 1423383 | 1 | 1 | 31% | 62.2% | 258 | 0.49 / 0.79 = 0.62 | noComparaHits: HOXA7 | 5 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 252.9 | scaffold_56 | 1443839 | 1444940 | 1 | 2 | 94.1% | 52.4% | 729 | 0.51 / 0.54 = 0.94 | ortholog_one2many: HOXA4 | 3 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 428 | scaffold_56 | 1459721 | 1465438 | 1 | 2 | 96.3% | 54.2% | 1239 | 0.56 / 0.53 = 1.06 | between_species_paralog: HOXA3 | 2 |
| --- | q11 | gapp | - | - | - | q11 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q12 | +/+ | ENSP00000328598 | HOXD1 | q12 | q12 | 125 | scaffold_56 | 1480876 | 1481115 | 1 | 1 | 25.6% | 73.8% | 240 | 0.79 / 0.8 = 0.99 | noComparaHits: hoxa1 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
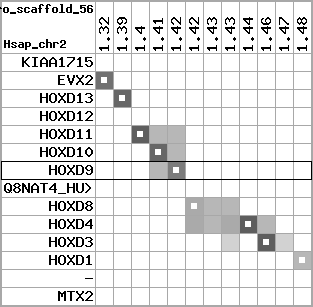
region 16 (region 3 for Xtro)
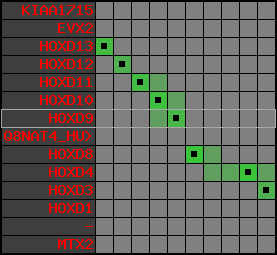 |
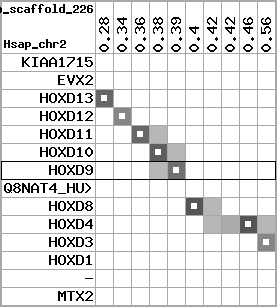 |
 |
retrieve target region: getEnsemblProteins.pl -h Xenopus_tropicalis -l scaffold_226 -i 281096:558409 -f 21e4 some_geneID_to_be_selected
scoreRatio_sum=7.79 log_scoreRatio_sum=-0.22 focal_scoreRatio_sum=3
AlnScore=3255 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Xenopus_tropicalis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 264 | scaffold_226 | 281096 | 284136 | 1 | 2 | 71.6% | 51.3% | 777 | 0.61 / 0.59 = 1.03 | between_species_paralog: ENSXETG00000025184 | 3 |
| p4 | q2 | +/+ | ENSP00000303578 | HOXD12 | q2 | q2 | 186.3 | scaffold_226 | 343006 | 344320 | 1 | 2 | 98.2% | 43.1% | 774 | 0.66 / 0.66 = 1 | noComparaHits: ENSXETG00000023470 | 1 |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 224.7 | scaffold_226 | 362428 | 364625 | 1 | 2 | 97.9% | 41.4% | 858 | 0.53 / 0.59 = 0.9 | ortholog_one2many: HOXC11 | 3 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 302 | scaffold_226 | 374651 | 378201 | 1 | 2 | 78.5% | 56.9% | 795 | 0.58 / 0.56 = 1.04 | noComparaHits: HOXC10 | 2 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 252 | scaffold_226 | 388156 | 390489 | 1 | 2 | 76% | 53.2% | 765 | 0.6 / 0.62 = 0.97 | noComparaHits: hoxc9 | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 273 | scaffold_226 | 396316 | 398530 | 1 | 2 | 90.7% | 56.6% | 648 | 0.49 / 0.5 = 0.98 | between_species_paralog: hoxc8 | 3 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 273.4 | scaffold_226 | 463073 | 464312 | 1 | 2 | 100% | 55.6% | 795 | 0.51 / 0.5 = 1.02 | between_species_paralog: HOXC4 | 2 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 303.6 | scaffold_226 | 555793 | 558409 | 1 | 2 | 96.3% | 44% | 1194 | 0.56 / 0.66 = 0.85 | noComparaHits: ENSXETG00000025181 | 4 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 17 (region 2 for Hsap)
 |
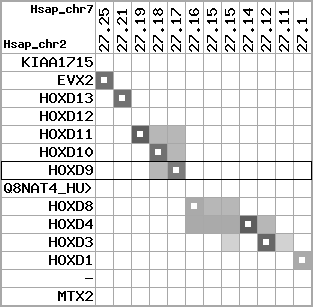 |
 |
retrieve target region: getEnsemblProteins.pl -h Homo_sapiens -l 7 -i 27100620:27252566 -f 11e4 some_geneID_to_be_selected
scoreRatio_sum=8.37 log_scoreRatio_sum=-0.72 focal_scoreRatio_sum=2.89
AlnScore=3177 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Homo_sapiens
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 305 | 7 | 27251188 | 27252566 | 1 | 2 | 69.7% | 52.3% | 828 | 0.62 / 0.62 = 1 | within_species_paralog: EVX1 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 252 | 7 | 27204348 | 27205966 | -1 | 2 | 90.4% | 49.2% | 909 | 0.61 / 0.61 = 1 | within_species_paralog: HOXA13 | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 260 | 7 | 27188946 | 27191285 | -1 | 2 | 79% | 55.4% | 690 | 0.53 / 0.53 = 1 | within_species_paralog: HOXA11 | 2 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 257.4 | 7 | 27178046 | 27180390 | -1 | 2 | 70.9% | 52.2% | 861 | 0.58 / 0.62 = 0.94 | noComparaHits: HOXA10 | 3 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 237.5 | 7 | 27169762 | 27171601 | -1 | 2 | 85.1% | 47.9% | 792 | 0.6 / 0.65 = 0.92 | within_species_paralog: HOXA9 | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 115 | 7 | 27161095 | 27161370 | -1 | 1 | 32.4% | 60.6% | 276 | 0.49 / 0.78 = 0.63 | noComparaHits: HOXA7 | 4 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 241.5 | 7 | 27135414 | 27136877 | -1 | 2 | 95.3% | 47.1% | 945 | 0.51 / 0.56 = 0.91 | within_species_paralog: HOXA4 | 4 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 376.8 | 7 | 27114062 | 27116784 | -1 | 2 | 96.3% | 48.5% | 1338 | 0.56 / 0.58 = 0.97 | within_species_paralog: HOXA3 | 3 |
| --- | q11 | gapp | - | - | - | q11 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q12 | +/+ | ENSP00000328598 | HOXD1 | q12 | q12 | 129 | 7 | 27100659 | 27100940 | -1 | 1 | 33.8% | 61.3% | 282 | 0.79 / 0.79 = 1 | noComparaHits: HOXA1 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 18 (region 2 for Mmus)
 |
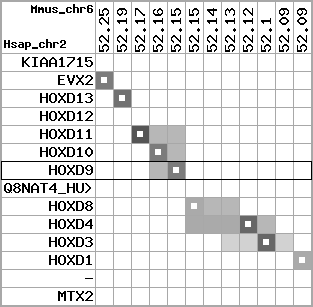 |
 |
retrieve target region: getEnsemblProteins.pl -h Mus_musculus -l 6 -i 52086299:52246434 -f 12e4 some_geneID_to_be_selected
scoreRatio_sum=8.39 log_scoreRatio_sum=-0.71 focal_scoreRatio_sum=2.91
AlnScore=3171 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Mus_musculus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 293 | 6 | 52245312 | 52246434 | 1 | 2 | 42.9% | 71.4% | 591 | 0.62 / 0.64 = 0.97 | between_species_paralog: Evx1 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 251 | 6 | 52188493 | 52189924 | -1 | 2 | 71.9% | 55% | 771 | 0.61 / 0.61 = 1 | between_species_paralog: Hoxa13 | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 273 | 6 | 52172949 | 52175302 | -1 | 2 | 87.3% | 54% | 777 | 0.53 / 0.5 = 1.06 | between_species_paralog: Hoxa11 | 2 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 250.9 | 6 | 52162092 | 52164457 | -1 | 2 | 70.9% | 50.3% | 882 | 0.58 / 0.63 = 0.92 | noComparaHits: Hoxa10 | 3 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 239.3 | 6 | 52153842 | 52155687 | -1 | 2 | 91.8% | 46.3% | 834 | 0.6 / 0.64 = 0.94 | between_species_paralog: Hoxa9 | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 112 | 6 | 52145417 | 52145611 | -1 | 1 | 22.4% | 76.9% | 195 | 0.49 / 0.79 = 0.62 | noComparaHits: Hoxa7 | 4 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 227.1 | 6 | 52119961 | 52121271 | -1 | 2 | 95.7% | 47.6% | 825 | 0.51 / 0.58 = 0.88 | ortholog_one2many: Hoxa4 | 4 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 395.9 | 6 | 52099526 | 52102232 | -1 | 2 | 96.3% | 49.7% | 1347 | 0.56 / 0.56 = 1 | between_species_paralog: Hoxa3 | 3 |
| --- | q11 | gapp | - | - | - | q11 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q12 | +/+ | ENSP00000328598 | HOXD1 | q12 | q12 | 131 | 6 | 52086392 | 52086673 | -1 | 1 | 33.8% | 62.2% | 282 | 0.79 / 0.79 = 1 | noComparaHits: Hoxa1 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 19 (region 3 for Gacu)
 |
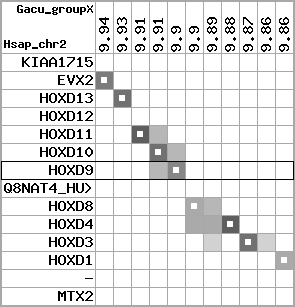 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l groupX -i 9855319:9936730 -f 61e3 some_geneID_to_be_selected
scoreRatio_sum=8.31 log_scoreRatio_sum=-0.79 focal_scoreRatio_sum=2.85
AlnScore=3152 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 299 | groupX | 9935674 | 9936730 | 1 | 2 | 67.6% | 49.7% | 798 | 0.62 / 0.63 = 0.98 | between_species_paralog: ENSGACG00000007148 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 249 | groupX | 9924933 | 9926178 | -1 | 2 | 72.2% | 56.9% | 687 | 0.61 / 0.61 = 1 | between_species_paralog: Q9PWC4_GASAC | 4 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 251 | groupX | 9914075 | 9915690 | -1 | 2 | 84.6% | 50.2% | 807 | 0.53 / 0.54 = 0.98 | between_species_paralog: ENSGACG00000007132 | 2 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 271 | groupX | 9906816 | 9908590 | -1 | 2 | 97.9% | 49.4% | 1038 | 0.58 / 0.6 = 0.97 | noComparaHits: ENSGACG00000007128 | 2 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 214.1 | groupX | 9901362 | 9902621 | -1 | 2 | 54.4% | 58.5% | 537 | 0.6 / 0.68 = 0.88 | between_species_paralog: ENSGACG00000007123 | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 118 | groupX | 9896091 | 9896378 | -1 | 1 | 32.4% | 61.5% | 288 | 0.49 / 0.78 = 0.63 | noComparaHits: ENSGACG00000007112 | 2 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q8 | +/+ | ENSP00000302548 | HOXD4 | q8 | q8 | 250.6 | groupX | 9880458 | 9881722 | -1 | 2 | 95.3% | 53.8% | 732 | 0.51 / 0.54 = 0.94 | between_species_paralog: ENSGACG00000007100 | 4 |
| p11 | q9 | +/+ | ENSP00000249440 | HOXD3 | q9 | q9 | 365.3 | groupX | 9866274 | 9869714 | -1 | 2 | 96.1% | 49.2% | 1245 | 0.56 / 0.6 = 0.93 | between_species_paralog: ENSGACG00000007094 | 2 |
| --- | q10 | gapp | - | - | - | q10 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 127 | groupX | 9855319 | 9855645 | -1 | 1 | 32% | 60.6% | 327 | 0.79 / 0.79 = 1 | noComparaHits: ENSGACG00000007085 | 1 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 20 (region 2 for Olat)
 |
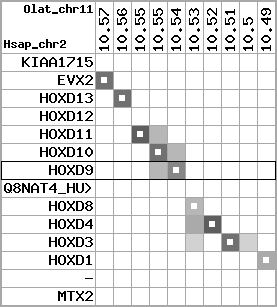 |
 |
retrieve target region: getEnsemblProteins.pl -h Oryzias_latipes -l 11 -i 10492554:10572561 -f 60e3 some_geneID_to_be_selected
scoreRatio_sum=8.31 log_scoreRatio_sum=-0.8 focal_scoreRatio_sum=2.85
AlnScore=3134 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Oryzias_latipes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 302 | 11 | 10571579 | 10572561 | 1 | 2 | 64.5% | 52% | 753 | 0.62 / 0.62 = 1 | between_species_paralog: Q2WFV6_ORYLA | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 249 | 11 | 10561902 | 10563169 | -1 | 2 | 93.7% | 48.3% | 795 | 0.61 / 0.61 = 1 | between_species_paralog: Q3V628_ORYLA | 3 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 256 | 11 | 10551758 | 10553425 | -1 | 2 | 77.2% | 54.7% | 711 | 0.53 / 0.53 = 1 | between_species_paralog: Q3V629_ORYLA | 1 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 273 | 11 | 10545005 | 10546843 | -1 | 2 | 96.5% | 49% | 1032 | 0.58 / 0.6 = 0.97 | noComparaHits: Q3V630_ORYLA | 2 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 213.2 | 11 | 10539576 | 10540702 | -1 | 2 | 80.7% | 43% | 768 | 0.6 / 0.68 = 0.88 | noComparaHits: Q9PTV6_ORYLA | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 102 | 11 | 10525404 | 10525595 | -1 | 1 | 22.1% | 70.3% | 192 | 0.49 / 0.81 = 0.6 | noComparaHits: Q9PTV6_ORYLA | 7 |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 242.6 | 11 | 10516220 | 10517399 | -1 | 2 | 90.2% | 49.5% | 777 | 0.51 / 0.55 = 0.93 | noComparaHits: Q9PTV6_ORYLA | 4 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 362.9 | 11 | 10503391 | 10506703 | -1 | 2 | 96.1% | 48.4% | 1242 | 0.56 / 0.6 = 0.93 | between_species_paralog: Q3V635_ORYLA | 2 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 130 | 11 | 10492596 | 10492934 | -1 | 1 | 34.5% | 56.6% | 339 | 0.79 / 0.79 = 1 | noComparaHits: Q3V637_ORYLA | 1 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 21 (region 2 for Drer)
 |
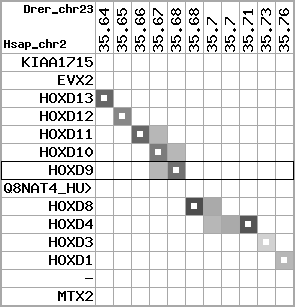 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 23 -i 35634616:35764976 -f 98e3 some_geneID_to_be_selected
scoreRatio_sum=8.41 log_scoreRatio_sum=-0.68 focal_scoreRatio_sum=3.06
AlnScore=3126 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 274 | 23 | 35634616 | 35637143 | 1 | 2 | 79.1% | 52.8% | 750 | 0.61 / 0.57 = 1.07 | between_species_paralog: hoxc13a | 1 |
| p4 | q2 | +/+ | ENSP00000303578 | HOXD12 | q2 | q2 | 176.7 | 23 | 35645087 | 35646667 | 1 | 2 | 60.4% | 54.1% | 474 | 0.66 / 0.68 = 0.97 | noComparaHits: Q4PR84_BRARE | 1 |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 223.2 | 23 | 35656454 | 35658679 | 1 | 2 | 76% | 49.8% | 645 | 0.53 / 0.59 = 0.9 | between_species_paralog: hoxc11a | 4 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 239.5 | 23 | 35665105 | 35667836 | 1 | 2 | 98.2% | 44.8% | 1005 | 0.58 / 0.65 = 0.89 | noComparaHits: Q4PR86_BRARE | 4 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 264 | 23 | 35676857 | 35678677 | 1 | 2 | 79.2% | 52% | 708 | 0.6 / 0.61 = 0.98 | between_species_paralog: hoxc9a | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 284 | 23 | 35682935 | 35684928 | 1 | 2 | 97.9% | 54.6% | 726 | 0.49 / 0.47 = 1.04 | ortholog_one2many: hoxc8a | 1 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 258 | 23 | 35712551 | 35713772 | 1 | 2 | 91.8% | 56.8% | 702 | 0.51 / 0.53 = 0.96 | between_species_paralog: hoxc4a | 3 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 107 | 23 | 35729107 | 35729475 | 1 | 1 | 28.5% | 48.1% | 369 | 0.56 / 0.88 = 0.64 | noComparaHits: hoxc3a | 4 |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 109 | 23 | 35764719 | 35764910 | 1 | 1 | 19.5% | 84.4% | 192 | 0.79 / 0.82 = 0.96 | noComparaHits: hoxc1a | 5 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 22 (region 2 for Cfam)
 |
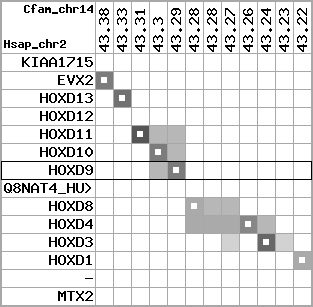 |
 |
retrieve target region: getEnsemblProteins.pl -h Canis_familiaris -l 14 -i 43223946:43377670 -f 12e4 some_geneID_to_be_selected
scoreRatio_sum=8.15 log_scoreRatio_sum=-0.98 focal_scoreRatio_sum=2.85
AlnScore=3024 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Canis_familiaris
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 282 | 14 | 43376231 | 43377670 | 1 | 2 | 68.9% | 49.4% | 792 | 0.62 / 0.65 = 0.95 | between_species_paralog: 482376 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 244 | 14 | 43328789 | 43330229 | -1 | 2 | 70.7% | 55.8% | 717 | 0.61 / 0.62 = 0.98 | ortholog_one2many: 482375 | 1 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 270 | 14 | 43312987 | 43315443 | -1 | 2 | 99.4% | 49.4% | 879 | 0.53 / 0.51 = 1.04 | between_species_paralog: HOXA11 | 2 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 252.8 | 14 | 43301964 | 43304339 | -1 | 2 | 70.9% | 51% | 864 | 0.58 / 0.63 = 0.92 | noComparaHits: 482373 | 3 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 242.1 | 14 | 43293519 | 43295428 | -1 | 2 | 92.1% | 46.5% | 837 | 0.6 / 0.64 = 0.94 | between_species_paralog: 482372 | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 113 | 14 | 43284774 | 43284974 | -1 | 1 | 23.4% | 77.9% | 201 | 0.49 / 0.79 = 0.62 | noComparaHits: 482371 | 4 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 171 | 14 | 43258571 | 43258876 | -1 | 1 | 39.2% | 82.4% | 306 | 0.51 / 0.68 = 0.75 | between_species_paralog: Q9XT68_CANFA | 3 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 375.1 | 14 | 43237520 | 43240239 | -1 | 2 | 96.3% | 47.5% | 1335 | 0.56 / 0.59 = 0.95 | between_species_paralog: HOXA3 | 2 |
| --- | q11 | gapp | - | - | - | q11 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q12 | +/+ | ENSP00000328598 | HOXD1 | q12 | q12 | 131 | 14 | 43223985 | 43224266 | -1 | 1 | 33.8% | 62.2% | 282 | 0.79 / 0.79 = 1 | noComparaHits: 482367 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 23 (region 2 for Tnig)
 |
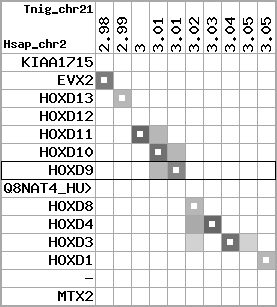 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l 21 -i 2648861:3053346 -f 30e4 some_geneID_to_be_selected
scoreRatio_sum=7.97 log_scoreRatio_sum=-1.17 focal_scoreRatio_sum=2.79
AlnScore=2884 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 289 | 21 | 2978001 | 2979139 | -1 | 2 | 68.1% | 48% | 840 | 0.62 / 0.64 = 0.97 | between_species_paralog: EVX-HOXA
; GSTENG00017475001 |
2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 130 | 21 | 2987051 | 2987620 | 1 | 1 | 58.2% | 42% | 570 | 0.61 / 0.79 = 0.77 | between_species_paralog: HOXA13
; GSTENG00017476001 |
3 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 237.7 | 21 | 2999952 | 3001417 | 1 | 2 | 83.1% | 49% | 768 | 0.53 / 0.57 = 0.93 | between_species_paralog: HOXA11 | 2 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 276 | 21 | 3006865 | 3008450 | 1 | 2 | 98.8% | 47.5% | 1053 | 0.58 / 0.6 = 0.97 | noComparaHits: GSTENG00017477001
; HOXA10 |
3 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 219.3 | 21 | 3012226 | 3013378 | 1 | 2 | 80.4% | 46% | 753 | 0.6 / 0.67 = 0.9 | between_species_paralog: HOXA9
; GSTENG00017478001 |
2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 105 | 21 | 3022350 | 3022565 | 1 | 1 | 24.8% | 63.9% | 216 | 0.49 / 0.8 = 0.61 | noComparaHits: GSTENG00017481001
; HOXA5 |
6 |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 226.9 | 21 | 3029847 | 3031009 | 1 | 2 | 87.1% | 52.2% | 651 | 0.51 / 0.58 = 0.88 | ortholog_one2many: HOXA4 | 4 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 367.3 | 21 | 3040373 | 3043259 | 1 | 2 | 96.3% | 49.4% | 1251 | 0.56 / 0.59 = 0.95 | between_species_paralog: HOXA3
; GSTENG00017482001 |
3 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 122 | 21 | 3053065 | 3053292 | 1 | 1 | 23.2% | 75% | 228 | 0.79 / 0.8 = 0.99 | noComparaHits: GSTENG00017483001
; HOXA1 |
1 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 24 (region 2 for Mdom)
 |
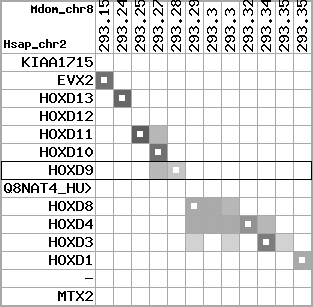 |
 |
retrieve target region: getEnsemblProteins.pl -h Monodelphis_domestica -l 8 -i 293153229:293354370 -f 15e4 some_geneID_to_be_selected
scoreRatio_sum=7.94 log_scoreRatio_sum=-1.25 focal_scoreRatio_sum=2.62
AlnScore=2813 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Monodelphis_domestica
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 318 | 8 | 293153229 | 293154729 | -1 | 2 | 61.8% | 57.8% | 690 | 0.62 / 0.6 = 1.03 | between_species_paralog: EVX1 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 264 | 8 | 293234916 | 293236747 | 1 | 2 | 90.4% | 50.1% | 942 | 0.61 / 0.59 = 1.03 | between_species_paralog: Q9TUC8_MONDO | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 258 | 8 | 293253079 | 293255747 | 1 | 2 | 79% | 54.2% | 690 | 0.53 / 0.53 = 1 | between_species_paralog: HOXA11 | 2 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 260 | 8 | 293266102 | 293268944 | 1 | 2 | 98.8% | 41.5% | 1212 | 0.58 / 0.62 = 0.94 | noComparaHits: HOXA10 | 2 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q4 | q5 | 107 | 8 | 293276346 | 293276780 | 1 | 1 | 51.8% | 37.3% | 435 | 0.6 / 0.84 = 0.71 | between_species_paralog: HOXA9 | 5 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 115 | 8 | 293289531 | 293289833 | 1 | 1 | 31.4% | 56.9% | 303 | 0.49 / 0.78 = 0.63 | noComparaHits: HOXA9 | 2 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 157.2 | 8 | 293316292 | 293317763 | 1 | 2 | 95.3% | 37.5% | 924 | 0.51 / 0.71 = 0.72 | between_species_paralog: HOXA4 | 3 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 329 | 8 | 293338320 | 293339276 | 1 | 1 | 79.4% | 52.7% | 957 | 0.56 / 0.64 = 0.88 | between_species_paralog: HOXA3 | 3 |
| --- | q11 | gapp | - | - | - | q11 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q12 | +/+ | ENSP00000328598 | HOXD1 | q12 | q12 | 127 | 8 | 293354020 | 293354274 | 1 | 1 | 29.3% | 66.7% | 255 | 0.79 / 0.79 = 1 | noComparaHits: HOXA1 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 25 (region 2 for Ggal)
 |
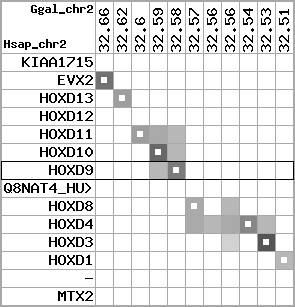 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l 2 -i 32513637:32659744 -f 11e4 some_geneID_to_be_selected
scoreRatio_sum=7.95 log_scoreRatio_sum=-1.26 focal_scoreRatio_sum=2.77
AlnScore=2812 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 319 | 2 | 32658369 | 32659744 | 1 | 2 | 62.2% | 56.4% | 708 | 0.62 / 0.6 = 1.03 | between_species_paralog: EVX1 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 154 | 2 | 32621241 | 32621825 | -1 | 1 | 63.3% | 44% | 585 | 0.61 / 0.76 = 0.8 | noComparaHits: HXA13_CHICK | 4 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 139 | 2 | 32600653 | 32601117 | -1 | 1 | 40.8% | 54.1% | 465 | 0.53 / 0.75 = 0.71 | between_species_paralog: HXA11_CHICK | 3 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 288 | 2 | 32588549 | 32590928 | -1 | 2 | 98.8% | 47.8% | 1068 | 0.58 / 0.58 = 1 | noComparaHits: ENSGALG00000011072
; HOXA10 ; ENSGALG00000021833 |
2 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 232.1 | 2 | 32579731 | 32581455 | -1 | 2 | 89.2% | 45.3% | 858 | 0.6 / 0.65 = 0.92 | between_species_paralog: HXA9_CHICK | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 115 | 2 | 32570317 | 32570610 | -1 | 1 | 33.1% | 56.1% | 294 | 0.49 / 0.78 = 0.63 | noComparaHits: HXA7_CHICK | 4 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 172 | 2 | 32544807 | 32545190 | -1 | 1 | 53.7% | 64.5% | 384 | 0.51 / 0.68 = 0.75 | between_species_paralog: HXA4_CHICK | 2 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 451 | 2 | 32524757 | 32527370 | -1 | 2 | 96.3% | 55.6% | 1242 | 0.56 / 0.5 = 1.12 | between_species_paralog: HXD3_CHICK | 2 |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 120 | 2 | 32513709 | 32513972 | -1 | 1 | 30.2% | 63.6% | 264 | 0.79 / 0.8 = 0.99 | noComparaHits: Q804C5_CHICK | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 26 (region 2 for Mmul)
 |
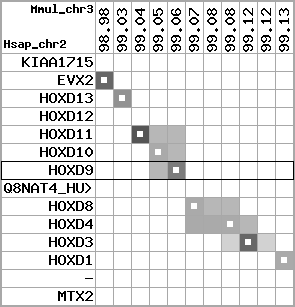 |
 |
retrieve target region: getEnsemblProteins.pl -h Macaca_mulatta -l 3 -i 98980072:99131547 -f 11e4 some_geneID_to_be_selected
scoreRatio_sum=7.89 log_scoreRatio_sum=-1.32 focal_scoreRatio_sum=2.74
AlnScore=2787 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Macaca_mulatta
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 327 | 3 | 98980072 | 98981451 | -1 | 2 | 69.7% | 54.8% | 828 | 0.62 / 0.59 = 1.05 | between_species_paralog: EVX1 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 193.2 | 3 | 99025864 | 99026971 | 1 | 2 | 38.2% | 69.8% | 387 | 0.61 / 0.7 = 0.87 | between_species_paralog: HOXA13 | 3 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 265 | 3 | 99039974 | 99042317 | 1 | 2 | 87% | 53.4% | 765 | 0.53 / 0.52 = 1.02 | between_species_paralog: HOXA11 | 1 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 160 | 3 | 99053148 | 99053396 | 1 | 1 | 24.4% | 92.8% | 249 | 0.58 / 0.76 = 0.76 | noComparaHits: HOXA10 | 3 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 240.6 | 3 | 99059984 | 99061835 | 1 | 2 | 85.1% | 48.5% | 792 | 0.6 / 0.64 = 0.94 | between_species_paralog: HOXA9 | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 114 | 3 | 99070144 | 99070356 | 1 | 1 | 24.8% | 73.6% | 213 | 0.49 / 0.79 = 0.62 | noComparaHits: HOXA7 | 4 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q8 | +/+ | ENSP00000302548 | HOXD4 | q8 | q8 | 125 | 3 | 99083848 | 99084063 | 1 | 1 | 28.2% | 83.3% | 216 | 0.51 / 0.77 = 0.66 | noComparaHits: HOXA5 | 5 |
| p11 | q9 | +/+ | ENSP00000249440 | HOXD3 | q9 | q9 | 380 | 3 | 99115329 | 99118052 | 1 | 2 | 96.3% | 48.1% | 1338 | 0.56 / 0.58 = 0.97 | between_species_paralog: HOXA3 | 2 |
| --- | q10 | gapp | - | - | - | q10 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 129 | 3 | 99131227 | 99131508 | 1 | 1 | 33.8% | 61.3% | 282 | 0.79 / 0.79 = 1 | noComparaHits: HOXA1 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 27 (region 2 for Trub)
 |
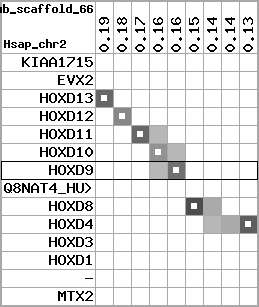 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_66 -i 126560:194452 -f 51e3 some_geneID_to_be_selected
scoreRatio_sum=6.72 log_scoreRatio_sum=-0.3 focal_scoreRatio_sum=2.71
AlnScore=2767 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 279 | scaffold_66 | 191852 | 194452 | -1 | 2 | 79.7% | 51.2% | 801 | 0.61 / 0.56 = 1.09 | between_species_paralog: NEWSINFRUG00000149366 | 1 |
| p4 | q2 | +/+ | ENSP00000303578 | HOXD12 | q2 | q2 | 181.8 | scaffold_66 | 183469 | 185171 | -1 | 2 | 85.6% | 42.4% | 705 | 0.66 / 0.67 = 0.99 | noComparaHits: NEWSINFRUG00000164408 | 2 |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 223.2 | scaffold_66 | 171700 | 173835 | -1 | 2 | 76% | 49% | 648 | 0.53 / 0.59 = 0.9 | between_species_paralog: NEWSINFRUG00000149367 | 3 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 206.6 | scaffold_66 | 162838 | 165131 | -1 | 2 | 69.7% | 48.8% | 717 | 0.58 / 0.7 = 0.83 | noComparaHits: NEWSINFRUG00000146333 | 3 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 251 | scaffold_66 | 154997 | 156667 | -1 | 2 | 66.7% | 57.3% | 591 | 0.6 / 0.63 = 0.95 | between_species_paralog: HXC9_FUGRU | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 279 | scaffold_66 | 149211 | 151058 | -1 | 2 | 92.8% | 55% | 693 | 0.49 / 0.48 = 1.02 | ortholog_one2many: O42503_FUGRU | 1 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 251.5 | scaffold_66 | 126560 | 127863 | -1 | 2 | 100% | 53.5% | 792 | 0.51 / 0.54 = 0.94 | between_species_paralog: NEWSINFRUG00000146324 | 3 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 28 (region 3 for Tnig)
 |
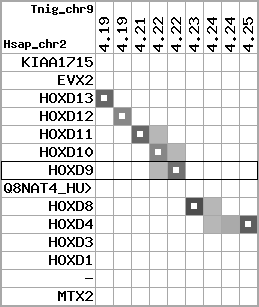 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l 9 -i 4184090:4253147 -f 52e3 some_geneID_to_be_selected
scoreRatio_sum=6.7 log_scoreRatio_sum=-0.31 focal_scoreRatio_sum=2.72
AlnScore=2766 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 271 | 9 | 4184090 | 4186692 | 1 | 2 | 79.7% | 51.5% | 756 | 0.61 / 0.58 = 1.05 | between_species_paralog: HOXC13
; GSTENG00028043001 |
1 |
| p4 | q2 | +/+ | ENSP00000303578 | HOXD12 | q2 | q2 | 185.2 | 9 | 4193048 | 4194863 | 1 | 2 | 98.9% | 41.2% | 801 | 0.66 / 0.66 = 1 | noComparaHits: HOXC12
; GSTENG00028042001 |
2 |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 224.3 | 9 | 4205466 | 4207565 | 1 | 2 | 76% | 49.4% | 648 | 0.53 / 0.59 = 0.9 | between_species_paralog: GSTENG00028041001
; HOXC11 |
4 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 209.8 | 9 | 4213950 | 4216213 | 1 | 2 | 84.4% | 43.6% | 870 | 0.58 / 0.69 = 0.84 | noComparaHits: HOXC10
; GSTENG00028041001 |
4 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 252 | 9 | 4222544 | 4224247 | 1 | 2 | 66.7% | 57.6% | 594 | 0.6 / 0.62 = 0.97 | between_species_paralog: HOXC9
; GSTENG00028039001 |
1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 276 | 9 | 4228016 | 4229940 | 1 | 2 | 89.7% | 57.3% | 639 | 0.49 / 0.49 = 1 | ortholog_one2many: HOXC8 | 1 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 252.5 | 9 | 4251930 | 4253138 | 1 | 2 | 91.8% | 57% | 705 | 0.51 / 0.54 = 0.94 | between_species_paralog: GSTENG00028038001
; HOXC4 |
2 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 29 (region 3 for Drer)
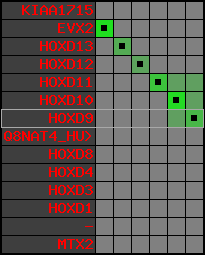 |
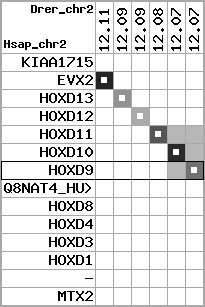 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 2 -i 12067413:12108344 -f 31e3 some_geneID_to_be_selected
scoreRatio_sum=7.83 log_scoreRatio_sum=1.12 focal_scoreRatio_sum=3
AlnScore=2764 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 588 | 2 | 12105996 | 12108344 | 1 | 3 | 100% | 66.7% | 1260 | 0.62 / 0.27 = 2.3 | ortholog_one2many: evx2 | 1 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 180.3 | 2 | 12090569 | 12093068 | -1 | 2 | 65.1% | 41.9% | 657 | 0.61 / 0.72 = 0.85 | noComparaHits: hoxd13a | 6 |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 119 | 2 | 12085228 | 12085470 | -1 | 1 | 29.1% | 70.4% | 243 | 0.66 / 0.78 = 0.85 | noComparaHits: hoxd12a | 2 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 271 | 2 | 12079179 | 12080879 | -1 | 2 | 100% | 46.5% | 846 | 0.53 / 0.51 = 1.04 | between_species_paralog: hoxd11a | 1 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 470 | 2 | 12071103 | 12072810 | -1 | 2 | 99.4% | 69.6% | 993 | 0.58 / 0.32 = 1.81 | ortholog_one2many: hoxd10a | 2 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 264 | 2 | 12067419 | 12068514 | -1 | 2 | 99.4% | 47.9% | 780 | 0.6 / 0.61 = 0.98 | ortholog_one2many: hoxd9a | 1 |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 30 (region 3 for Olat)
 |
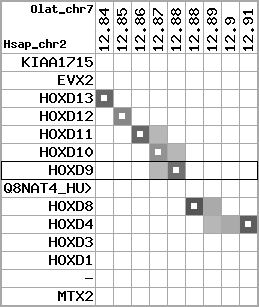 |
 |
retrieve target region: getEnsemblProteins.pl -h Oryzias_latipes -l 7 -i 12836772:12915743 -f 59e3 some_geneID_to_be_selected
scoreRatio_sum=6.71 log_scoreRatio_sum=-0.31 focal_scoreRatio_sum=2.72
AlnScore=2764 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Oryzias_latipes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 274 | 7 | 12836772 | 12839091 | 1 | 2 | 79.7% | 51.9% | 756 | 0.61 / 0.57 = 1.07 | between_species_paralog: Q3V600_ORYLA | 1 |
| p4 | q2 | +/+ | ENSP00000303578 | HOXD12 | q2 | q2 | 185.3 | 7 | 12845783 | 12847609 | 1 | 2 | 89.9% | 42.5% | 735 | 0.66 / 0.66 = 1 | noComparaHits: Q3V601_ORYLA | 2 |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 228.7 | 7 | 12857104 | 12859662 | 1 | 2 | 88.2% | 44.3% | 837 | 0.53 / 0.58 = 0.91 | between_species_paralog: Q90ZG3_ORYLA | 2 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 205.9 | 7 | 12866025 | 12868489 | 1 | 2 | 92.6% | 42.9% | 921 | 0.58 / 0.7 = 0.83 | noComparaHits: Q3V603_ORYLA | 4 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 255 | 7 | 12875582 | 12877170 | 1 | 2 | 66.7% | 58.7% | 591 | 0.6 / 0.62 = 0.97 | between_species_paralog: HXC9_ORYLA | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 274 | 7 | 12880841 | 12882567 | 1 | 2 | 90.7% | 56.6% | 648 | 0.49 / 0.49 = 1 | noComparaHits: HXC9_ORYLA
; Q3V609_ORYLA |
1 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 247.4 | 7 | 12904751 | 12905965 | 1 | 2 | 91.4% | 56.6% | 699 | 0.51 / 0.55 = 0.93 | between_species_paralog: HXC4_ORYLA | 3 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 31 (region 3 for Mmul)
 |
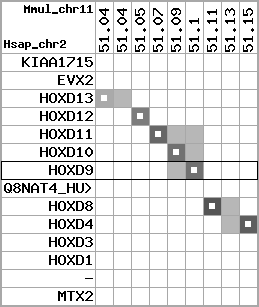 |
 |
retrieve target region: getEnsemblProteins.pl -h Macaca_mulatta -l 11 -i 51037199:51154580 -f 88e3 some_geneID_to_be_selected
scoreRatio_sum=6.63 log_scoreRatio_sum=-0.39 focal_scoreRatio_sum=2.77
AlnScore=2699 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Macaca_mulatta
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 142 | 11 | 51037199 | 51037708 | 1 | 1 | 49.9% | 41.8% | 510 | 0.61 / 0.78 = 0.78 | between_species_paralog: HOXC13 | 4 |
| --- | q2 | gapp | - | - | - | q2 | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 197.9 | 11 | 51052937 | 51054562 | 1 | 2 | 100% | 43.4% | 858 | 0.66 / 0.64 = 1.03 | noComparaHits: HOXC12 | 2 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 235.4 | 11 | 51071372 | 51073476 | 1 | 2 | 99.1% | 44.3% | 843 | 0.53 / 0.57 = 0.93 | ortholog_one2many: HOXC11 | 3 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 264.1 | 11 | 51083777 | 51087885 | 1 | 2 | 80% | 51.2% | 831 | 0.58 / 0.61 = 0.95 | noComparaHits: HOXC10 | 2 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 266 | 11 | 51098616 | 51101080 | 1 | 2 | 67.8% | 59.2% | 600 | 0.6 / 0.6 = 1 | ortholog_one2many: HOXC9 | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q7 | +/+ | ENSP00000315949 | HOXD8 | q7 | q7 | 270 | 11 | 51107689 | 51109683 | 1 | 2 | 89.7% | 52.4% | 666 | 0.49 / 0.5 = 0.98 | between_species_paralog: HOXC8 | 2 |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 253.3 | 11 | 51153294 | 51154580 | 1 | 2 | 100% | 52% | 792 | 0.51 / 0.53 = 0.96 | between_species_paralog: HOXC4 | 2 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 32 (region 3 for Trub)
 |
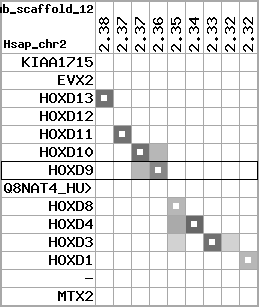 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_12 -i 2318883:2382177 -f 47e3 some_geneID_to_be_selected
scoreRatio_sum=7.12 log_scoreRatio_sum=-1.01 focal_scoreRatio_sum=2.63
AlnScore=2669 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 254 | scaffold_12 | 2381071 | 2382177 | -1 | 2 | 72.2% | 57.5% | 690 | 0.61 / 0.6 = 1.02 | between_species_paralog: NEWSINFRUG00000149643 | 4 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 216.2 | scaffold_12 | 2371832 | 2373459 | -1 | 2 | 44.7% | 71.4% | 447 | 0.53 / 0.61 = 0.87 | between_species_paralog: NEWSINFRUG00000157392 | 4 |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 276 | scaffold_12 | 2365048 | 2366742 | -1 | 2 | 95.9% | 49.3% | 1032 | 0.58 / 0.6 = 0.97 | noComparaHits: NEWSINFRUG00000149640 | 2 |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 216.4 | scaffold_12 | 2360015 | 2361166 | -1 | 2 | 69% | 50% | 633 | 0.6 / 0.68 = 0.88 | between_species_paralog: HXA9_FUGRU | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q5 | +/+ | ENSP00000315949 | HOXD8 | q5 | q5 | 103 | scaffold_12 | 2350075 | 2350314 | -1 | 1 | 27.2% | 60% | 240 | 0.49 / 0.81 = 0.6 | noComparaHits: | 7 |
| p10 | q6 | +/+ | ENSP00000302548 | HOXD4 | q6 | q6 | 235.3 | scaffold_12 | 2341356 | 2342542 | -1 | 2 | 90.2% | 53% | 675 | 0.51 / 0.57 = 0.89 | between_species_paralog: NEWSINFRUG00000149629 | 4 |
| p11 | q7 | +/+ | ENSP00000249440 | HOXD3 | q7 | q7 | 347.1 | scaffold_12 | 2329054 | 2331951 | -1 | 2 | 96.1% | 46.7% | 1245 | 0.56 / 0.62 = 0.9 | between_species_paralog: NEWSINFRUG00000149628 | 2 |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q9 | +/+ | ENSP00000328598 | HOXD1 | q9 | q9 | 124 | scaffold_12 | 2318949 | 2319182 | -1 | 1 | 23.8% | 75.6% | 234 | 0.79 / 0.8 = 0.99 | noComparaHits: NEWSINFRUG00000149627 | 1 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 33 (region 4 for Drer)
 |
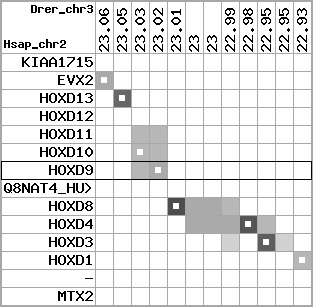 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 3 -i 22929834:23057956 -f 96e3 some_geneID_to_be_selected
scoreRatio_sum=7.39 log_scoreRatio_sum=-0.7 focal_scoreRatio_sum=2.44
AlnScore=2635 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 171.7 | 3 | 23057158 | 23057956 | 1 | 2 | 34% | 52.1% | 450 | 0.62 / 0.78 = 0.79 | noComparaHits: eve1 | 3 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 270 | 3 | 23044789 | 23046033 | -1 | 2 | 89.6% | 47.4% | 825 | 0.61 / 0.58 = 1.05 | between_species_paralog: ENSDARG00000056016
; hoxb13a |
2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | q4 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 139 | 3 | 23029315 | 23029581 | -1 | 1 | 26.2% | 71.9% | 267 | 0.58 / 0.79 = 0.73 | noComparaHits: hoxb10a | 5 |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 144 | 3 | 23019703 | 23019927 | -1 | 1 | 21.9% | 86.7% | 225 | 0.6 / 0.78 = 0.77 | between_species_paralog: hoxb9a | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q5 | +/+ | ENSP00000315949 | HOXD8 | q5 | q5 | 275 | 3 | 23010726 | 23012162 | -1 | 2 | 95.9% | 52.7% | 681 | 0.49 / 0.49 = 1 | between_species_paralog: hoxb8a | 2 |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 270 | 3 | 22976995 | 22978642 | -1 | 2 | 93.7% | 56.3% | 714 | 0.51 / 0.5 = 1.02 | between_species_paralog: hoxb4a | 2 |
| p11 | q10 | +/+ | ENSP00000249440 | HOXD3 | q10 | q10 | 420 | 3 | 22952393 | 22954315 | -1 | 2 | 96.3% | 53.1% | 1248 | 0.56 / 0.54 = 1.04 | between_species_paralog: hoxb3a | 2 |
| --- | q11 | gapp | - | - | - | q11 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q12 | +/+ | ENSP00000328598 | HOXD1 | q12 | q12 | 122 | 3 | 22929921 | 22930142 | -1 | 1 | 22.9% | 80% | 222 | 0.79 / 0.8 = 0.99 | noComparaHits: hoxb1a | 4 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 34 (region 4 for Gacu)
 |
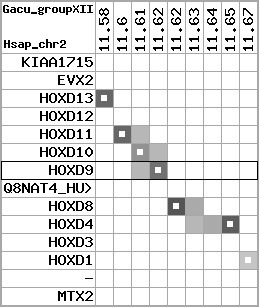 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l groupXII -i 11575579:11674038 -f 74e3 some_geneID_to_be_selected
scoreRatio_sum=6.68 log_scoreRatio_sum=-0.34 focal_scoreRatio_sum=2.63
AlnScore=2603 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 276 | groupXII | 11575579 | 11578147 | 1 | 2 | 79.1% | 52.2% | 756 | 0.61 / 0.57 = 1.07 | between_species_paralog: ENSGACG00000009389 | 1 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 226.5 | groupXII | 11597276 | 11599469 | 1 | 2 | 76.9% | 48.9% | 660 | 0.53 / 0.59 = 0.9 | between_species_paralog: ENSGACG00000009392 | 4 |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 201.3 | groupXII | 11606255 | 11609065 | 1 | 2 | 94.1% | 41.2% | 981 | 0.58 / 0.7 = 0.83 | noComparaHits: ENSGACG00000009394 | 3 |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 254 | groupXII | 11616265 | 11618032 | 1 | 2 | 66.7% | 58.1% | 591 | 0.6 / 0.62 = 0.97 | between_species_paralog: ENSGACG00000009396 | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q5 | +/+ | ENSP00000315949 | HOXD8 | q5 | q5 | 274 | groupXII | 11622080 | 11624013 | 1 | 2 | 90.3% | 54.9% | 672 | 0.49 / 0.49 = 1 | ortholog_one2many: ENSGACG00000009401 | 1 |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q8 | +/+ | ENSP00000302548 | HOXD4 | q8 | q8 | 257.1 | groupXII | 11647407 | 11648707 | 1 | 2 | 100% | 53.8% | 792 | 0.51 / 0.53 = 0.96 | between_species_paralog: ENSGACG00000009421 | 2 |
| p11 | --- | gapq | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q9 | +/+ | ENSP00000328598 | HOXD1 | q9 | q9 | 106 | groupXII | 11673811 | 11674017 | 1 | 1 | 21% | 72.5% | 207 | 0.79 / 0.83 = 0.95 | noComparaHits: ENSGACG00000009431 | 4 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 35 (region 3 for Hsap)
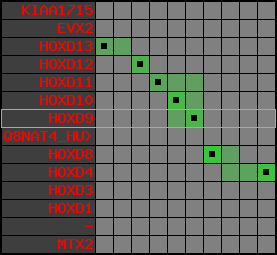 |
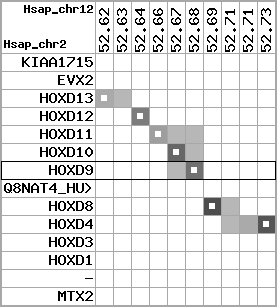 |
 |
retrieve target region: getEnsemblProteins.pl -h Homo_sapiens -l 12 -i 52619219:52735253 -f 87e3 some_geneID_to_be_selected
scoreRatio_sum=6.53 log_scoreRatio_sum=-0.53 focal_scoreRatio_sum=2.74
AlnScore=2597 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Homo_sapiens
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 151 | 12 | 52619219 | 52619716 | 1 | 1 | 50.7% | 43.3% | 498 | 0.61 / 0.76 = 0.8 | within_species_paralog: HOXC13 | 4 |
| --- | q2 | gapp | - | - | - | q2 | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 187.3 | 12 | 52634981 | 52636614 | 1 | 2 | 78.8% | 48.7% | 663 | 0.66 / 0.66 = 1 | noComparaHits: HOXC12 | 2 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 148 | 12 | 52654595 | 52655458 | 1 | 1 | 80.2% | 38.2% | 864 | 0.53 / 0.73 = 0.73 | within_species_paralog: HOXC11 | 3 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 288 | 12 | 52665383 | 52669494 | 1 | 2 | 92.1% | 50.5% | 954 | 0.58 / 0.58 = 1 | noComparaHits: HOXC10 | 2 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 266 | 12 | 52680240 | 52682713 | 1 | 2 | 67.8% | 59.2% | 600 | 0.6 / 0.6 = 1 | within_species_paralog: HOXC9 | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q7 | +/+ | ENSP00000315949 | HOXD8 | q7 | q7 | 278 | 12 | 52689336 | 52691360 | 1 | 2 | 92.1% | 52.5% | 687 | 0.49 / 0.49 = 1 | within_species_paralog: HOXC8 | 2 |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q10 | +/+ | ENSP00000302548 | HOXD4 | q10 | q10 | 265 | 12 | 52733974 | 52735253 | 1 | 2 | 100% | 54.4% | 792 | 0.51 / 0.51 = 1 | within_species_paralog: HOXC4 | 2 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 36 (region 5 for Drer)
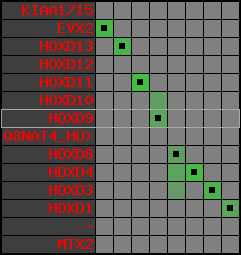 |
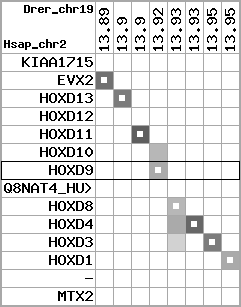 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 19 -i 13885840:13954198 -f 51e3 some_geneID_to_be_selected
scoreRatio_sum=7 log_scoreRatio_sum=-1.15 focal_scoreRatio_sum=2.19
AlnScore=2517 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 300 | 19 | 13885840 | 13886934 | -1 | 2 | 66.8% | 50.8% | 774 | 0.62 / 0.63 = 0.98 | between_species_paralog: evx1 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 224 | 19 | 13896550 | 13897422 | 1 | 1 | 76.1% | 45.3% | 873 | 0.61 / 0.65 = 0.94 | between_species_paralog: hoxa13a | 5 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 244 | 19 | 13904071 | 13905632 | 1 | 2 | 91.1% | 46.2% | 846 | 0.53 / 0.56 = 0.95 | between_species_paralog: hoxa11a | 3 |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q4 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 139 | 19 | 13917746 | 13917979 | 1 | 1 | 22.8% | 82.1% | 234 | 0.6 / 0.79 = 0.76 | between_species_paralog: hoxa9a | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q5 | +/+ | ENSP00000315949 | HOXD8 | q5 | q5 | 100 | 19 | 13927415 | 13927606 | 1 | 1 | 22.1% | 70.3% | 192 | 0.49 / 0.81 = 0.6 | noComparaHits: hoxa5a | noData |
| p10 | q6 | +/+ | ENSP00000302548 | HOXD4 | q6 | q6 | 228.9 | 19 | 13934404 | 13935531 | 1 | 2 | 89.8% | 51.5% | 681 | 0.51 / 0.58 = 0.88 | noComparaHits: ENSDARG00000057724
; hoxa3a |
4 |
| p11 | q7 | +/+ | ENSP00000249440 | HOXD3 | q7 | q7 | 337.4 | 19 | 13944458 | 13946294 | 1 | 2 | 96.3% | 45.4% | 1281 | 0.56 / 0.63 = 0.89 | between_species_paralog: hoxa3a | 3 |
| p12 | q8 | +/+ | ENSP00000328598 | HOXD1 | q8 | q8 | 128 | 19 | 13953818 | 13954138 | 1 | 1 | 32.6% | 61.7% | 321 | 0.79 / 0.79 = 1 | noComparaHits: hoxa1a | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 37 (region 3 for Mmus)
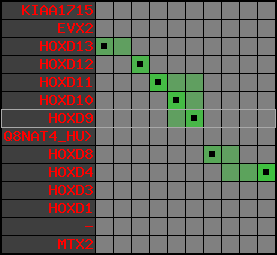 |
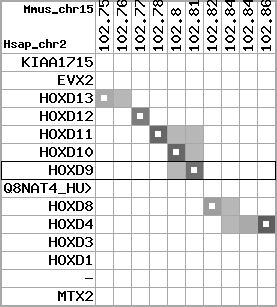 |
 |
retrieve target region: getEnsemblProteins.pl -h Mus_musculus -l 15 -i 102749444:102864023 -f 86e3 some_geneID_to_be_selected
scoreRatio_sum=6.33 log_scoreRatio_sum=-0.76 focal_scoreRatio_sum=2.68
AlnScore=2474 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Mus_musculus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 152 | 15 | 102749444 | 102749953 | 1 | 1 | 49.9% | 44.5% | 510 | 0.61 / 0.76 = 0.8 | between_species_paralog: Hoxc13 | 4 |
| --- | q2 | gapp | - | - | - | q2 | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 188.6 | 15 | 102764887 | 102766548 | 1 | 2 | 97.8% | 41.4% | 828 | 0.66 / 0.66 = 1 | noComparaHits: Hoxc12 | 2 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 233.9 | 15 | 102782620 | 102784729 | 1 | 2 | 99.1% | 44.2% | 846 | 0.53 / 0.57 = 0.93 | between_species_paralog: HXC11_MOUSE | 3 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 286 | 15 | 102794964 | 102799106 | 1 | 2 | 92.1% | 50.3% | 903 | 0.58 / 0.58 = 1 | noComparaHits: Hoxc10 | 2 |
| p7 | q6 | +/+ | ENSP00000249499 | HOXD9 | q6 | q6 | 266 | 15 | 102809687 | 102812161 | 1 | 2 | 67.8% | 59.2% | 600 | 0.6 / 0.6 = 1 | between_species_paralog: Hoxc9 | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q7 | +/+ | ENSP00000315949 | HOXD8 | q7 | q7 | 139 | 15 | 102820593 | 102820802 | 1 | 1 | 24.1% | 92.9% | 210 | 0.49 / 0.74 = 0.66 | between_species_paralog: Hoxc8 | 3 |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q10 | +/+ | ENSP00000302548 | HOXD4 | q10 | q10 | 252.9 | 15 | 102862757 | 102864023 | 1 | 2 | 100% | 51.5% | 792 | 0.51 / 0.54 = 0.94 | between_species_paralog: Hoxc4 | 2 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 38 (region 3 for Cfam)
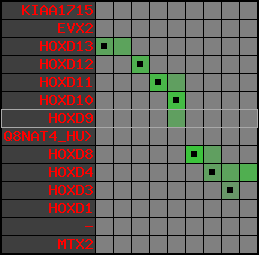 |
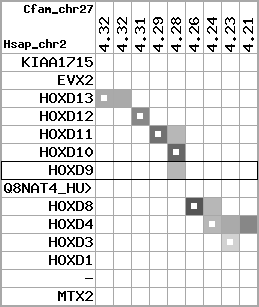 |
 |
retrieve target region: getEnsemblProteins.pl -h Canis_familiaris -l 27 -i 4211935:4324533 -f 84e3 some_geneID_to_be_selected
scoreRatio_sum=5.86 log_scoreRatio_sum=-1.34 focal_scoreRatio_sum=1.79
AlnScore=2109 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Canis_familiaris
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 142 | 27 | 4324138 | 4324533 | -1 | 1 | 42.1% | 47.9% | 396 | 0.61 / 0.78 = 0.78 | between_species_paralog: HOXC13 | 4 |
| --- | q2 | gapp | - | - | - | q2 | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 182.9 | 27 | 4307268 | 4308845 | -1 | 2 | 67.3% | 52.1% | 540 | 0.66 / 0.67 = 0.99 | noComparaHits: HOXC12 | 2 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 208.4 | 27 | 4288986 | 4290950 | -1 | 2 | 97.3% | 41.3% | 828 | 0.53 / 0.62 = 0.85 | between_species_paralog: HOXC11 | 3 |
| p6 | q5 | +/+ | ENSP00000249501 | HOXD10 | q5 | q5 | 281 | 27 | 4274733 | 4278842 | -1 | 2 | 92.1% | 48.9% | 903 | 0.58 / 0.59 = 0.98 | noComparaHits: HOXC10 | 2 |
| p7 | --- | gapq | ENSP00000249499 | HOXD9 | q5 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 269 | 27 | 4254363 | 4256431 | -1 | 2 | 90.7% | 55.5% | 648 | 0.49 / 0.5 = 0.98 | between_species_paralog: 607052 | 3 |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q9 | q7 | 110 | 27 | 4236052 | 4236291 | -1 | 1 | 31.4% | 63.8% | 240 | 0.51 / 0.8 = 0.64 | noComparaHits: 486499 | 10 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 103 | 27 | 4231110 | 4231502 | -1 | 1 | 25.9% | 49.6% | 393 | 0.56 / 0.88 = 0.64 | noComparaHits: 607625 | 5 |
| p12 | --- | gapq | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gapq | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q9 | gape | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
region 39 (region 2 for Btau)
 |
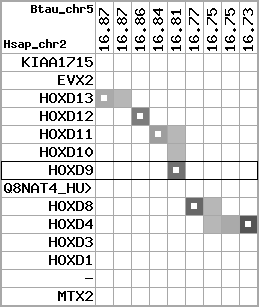 |
 |
retrieve target region: getEnsemblProteins.pl -h Bos_taurus -l 5 -i 16729786:16874708 -f 11e4 some_geneID_to_be_selected
scoreRatio_sum=5.42 log_scoreRatio_sum=-0.65 focal_scoreRatio_sum=2.2
AlnScore=2105 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Bos_taurus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 150 | 5 | 16874232 | 16874708 | -1 | 1 | 48.7% | 44.4% | 477 | 0.61 / 0.76 = 0.8 | between_species_paralog: HOXC13 | 4 |
| --- | q2 | gapp | - | - | - | q2 | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q3 | +/+ | ENSP00000303578 | HOXD12 | q3 | q3 | 190.2 | 5 | 16857536 | 16859124 | -1 | 2 | 100% | 41.8% | 849 | 0.66 / 0.65 = 1.02 | noComparaHits: HOXC12 | 2 |
| p5 | q4 | +/+ | ENSP00000249504 | HOXD11 | q4 | q4 | 144 | 5 | 16838896 | 16839789 | -1 | 1 | 76.6% | 38% | 894 | 0.53 / 0.74 = 0.72 | ortholog_one2many: HOXC11 | 3 |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q5 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 266 | 5 | 16811875 | 16814511 | -1 | 2 | 67.8% | 59.2% | 600 | 0.6 / 0.6 = 1 | between_species_paralog: HOXC9 | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q6 | +/+ | ENSP00000315949 | HOXD8 | q6 | q6 | 232.6 | 5 | 16772492 | 16774311 | -1 | 2 | 48.6% | 76.8% | 423 | 0.49 / 0.57 = 0.86 | between_species_paralog: HOXC8 | 2 |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q9 | +/+ | ENSP00000302548 | HOXD4 | q9 | q9 | 272 | 5 | 16729786 | 16731061 | -1 | 2 | 100% | 53.3% | 840 | 0.51 / 0.5 = 1.02 | between_species_paralog: HOXC4 | 2 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 40 (region 4 for Mmus)
 |
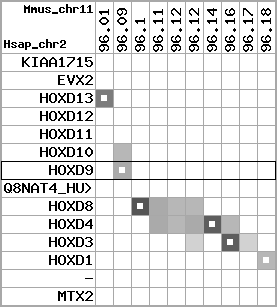 |
 |
retrieve target region: getEnsemblProteins.pl -h Mus_musculus -l 11 -i 96010704:96183205 -f 13e4 some_geneID_to_be_selected
scoreRatio_sum=5.63 log_scoreRatio_sum=-0.4 focal_scoreRatio_sum=1.89
AlnScore=2082 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Mus_musculus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 229 | 11 | 96010704 | 96012313 | 1 | 2 | 72.5% | 48% | 684 | 0.61 / 0.64 = 0.95 | between_species_paralog: Hoxb13 | 3 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 145 | 11 | 96090716 | 96090937 | 1 | 1 | 21.6% | 89.2% | 222 | 0.6 / 0.78 = 0.77 | between_species_paralog: Hoxb9 | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q3 | +/+ | ENSP00000315949 | HOXD8 | q3 | q3 | 270 | 11 | 96099053 | 96100527 | 1 | 2 | 98.6% | 52.6% | 699 | 0.49 / 0.5 = 0.98 | between_species_paralog: Hoxb8 | 2 |
| --- | q4 | gapp | - | - | - | q4 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q5 | gapp | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 240.3 | 11 | 96134860 | 96136373 | 1 | 2 | 89.8% | 54.7% | 708 | 0.51 / 0.56 = 0.91 | between_species_paralog: Hoxb4 | 3 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 418 | 11 | 96160327 | 96162486 | 1 | 2 | 96.8% | 54.8% | 1224 | 0.56 / 0.54 = 1.04 | between_species_paralog: Hoxb3 | 2 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 114 | 11 | 96182903 | 96183118 | 1 | 1 | 22.3% | 75.3% | 216 | 0.79 / 0.81 = 0.98 | noComparaHits: Hoxb1 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 41 (region 4 for Hsap)
 |
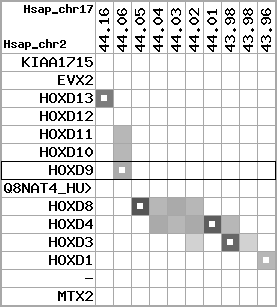 |
 |
retrieve target region: getEnsemblProteins.pl -h Homo_sapiens -l 17 -i 43961923:44160786 -f 15e4 some_geneID_to_be_selected
scoreRatio_sum=5.62 log_scoreRatio_sum=-0.4 focal_scoreRatio_sum=1.88
AlnScore=2075 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Homo_sapiens
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 227 | 17 | 44159157 | 44160786 | -1 | 2 | 70.7% | 48.1% | 663 | 0.61 / 0.64 = 0.95 | within_species_paralog: HOXB13 | 3 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 145 | 17 | 44055273 | 44055494 | -1 | 1 | 21.6% | 89.2% | 222 | 0.6 / 0.78 = 0.77 | within_species_paralog: HOXB9 | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q3 | +/+ | ENSP00000315949 | HOXD8 | q3 | q3 | 269 | 17 | 44045623 | 44047065 | -1 | 2 | 94.8% | 53.3% | 672 | 0.49 / 0.5 = 0.98 | within_species_paralog: HOXB8 | 3 |
| --- | q4 | gapp | - | - | - | q4 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q5 | gapp | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 249.4 | 17 | 44009128 | 44010680 | -1 | 2 | 89.8% | 54.1% | 726 | 0.51 / 0.54 = 0.94 | within_species_paralog: HOXB4 | 3 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 398 | 17 | 43982698 | 43984856 | -1 | 2 | 97.2% | 52.3% | 1233 | 0.56 / 0.56 = 1 | within_species_paralog: HOXB3 | 2 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 115 | 17 | 43961923 | 43962234 | -1 | 1 | 31.7% | 60% | 312 | 0.79 / 0.81 = 0.98 | noComparaHits: HOXB1 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 42 (region 4 for Trub)
 |
 |
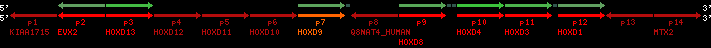 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_41 -i 485042:661622 -f 13e4 some_geneID_to_be_selected
scoreRatio_sum=6.07 log_scoreRatio_sum=-1.06 focal_scoreRatio_sum=1.9
AlnScore=2044 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 153.3 | scaffold_41 | 485042 | 487257 | -1 | 2 | 30.9% | 51% | 432 | 0.62 / 0.81 = 0.77 | noComparaHits: | 3 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 264 | scaffold_41 | 501905 | 503538 | 1 | 2 | 89% | 48% | 855 | 0.61 / 0.59 = 1.03 | between_species_paralog: NEWSINFRUG00000163355 | 3 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q3 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q3 | +/+ | ENSP00000249499 | HOXD9 | q3 | q3 | 142 | scaffold_41 | 540964 | 541188 | 1 | 1 | 21.9% | 86.7% | 225 | 0.6 / 0.79 = 0.76 | noComparaHits: NEWSINFRUG00000160069 | 4 |
| --- | q4 | gapp | - | - | - | q4 | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q5 | +/+ | ENSP00000315949 | HOXD8 | q5 | q5 | 146 | scaffold_41 | 555237 | 555512 | 1 | 1 | 30.3% | 79.3% | 276 | 0.49 / 0.73 = 0.67 | between_species_paralog: NEWSINFRUG00000136620 | 2 |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q7 | gapp | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q8 | +/+ | ENSP00000302548 | HOXD4 | q8 | q8 | 257.9 | scaffold_41 | 601559 | 603728 | 1 | 2 | 89.8% | 56.1% | 708 | 0.51 / 0.53 = 0.96 | between_species_paralog: HXB4_FUGRU | 2 |
| p11 | q9 | +/+ | ENSP00000249440 | HOXD3 | q9 | q9 | 334.7 | scaffold_41 | 633457 | 637089 | 1 | 2 | 99.3% | 45.6% | 1458 | 0.56 / 0.63 = 0.89 | between_species_paralog: Q6IEH9_FUGRU | 3 |
| --- | q10 | gapp | - | - | - | q10 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q11 | +/+ | ENSP00000328598 | HOXD1 | q11 | q11 | 120 | scaffold_41 | 661200 | 661538 | 1 | 1 | 33.5% | 59.1% | 339 | 0.79 / 0.8 = 0.99 | noComparaHits: Q6IEI1_FUGRU | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 43 (region 4 for Cfam)
 |
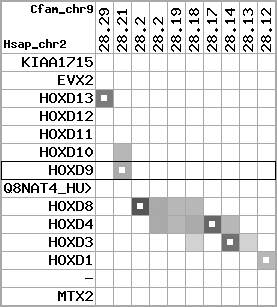 |
 |
retrieve target region: getEnsemblProteins.pl -h Canis_familiaris -l 9 -i 28119456:28287807 -f 13e4 some_geneID_to_be_selected
scoreRatio_sum=5.46 log_scoreRatio_sum=-0.58 focal_scoreRatio_sum=1.85
AlnScore=1994 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Canis_familiaris
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 226 | 9 | 28285962 | 28287807 | -1 | 2 | 71.3% | 48.2% | 669 | 0.61 / 0.65 = 0.94 | between_species_paralog: 491060 | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 145 | 9 | 28212650 | 28212871 | -1 | 1 | 21.6% | 89.2% | 222 | 0.6 / 0.78 = 0.77 | ortholog_one2many: 608958 | 3 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q3 | +/+ | ENSP00000315949 | HOXD8 | q3 | q3 | 273 | 9 | 28202580 | 28204191 | -1 | 2 | 94.8% | 52.8% | 708 | 0.49 / 0.5 = 0.98 | between_species_paralog: 491059 | 2 |
| --- | q4 | gapp | - | - | - | q4 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q5 | gapp | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 222.5 | 9 | 28165652 | 28167383 | -1 | 2 | 75.3% | 58.8% | 501 | 0.51 / 0.59 = 0.86 | between_species_paralog: HOXB4 | 2 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 363.4 | 9 | 28138727 | 28140962 | -1 | 2 | 97.2% | 48.8% | 1260 | 0.56 / 0.6 = 0.93 | noComparaHits: ENSCAFG00000016845 | 3 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 115 | 9 | 28119861 | 28120124 | -1 | 1 | 28% | 65.2% | 264 | 0.79 / 0.81 = 0.98 | noComparaHits: 491052 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 44 (region 5 for Gacu)
 |
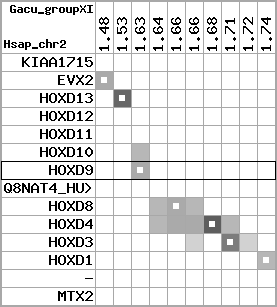 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l groupXI -i 1481474:1740599 -f 19e4 some_geneID_to_be_selected
scoreRatio_sum=6.01 log_scoreRatio_sum=-1.14 focal_scoreRatio_sum=1.89
AlnScore=1985 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 166.4 | groupXI | 1481474 | 1484334 | -1 | 2 | 38.4% | 46.9% | 540 | 0.62 / 0.79 = 0.78 | noComparaHits: | 3 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 264 | groupXI | 1524294 | 1526021 | 1 | 2 | 90.7% | 48.9% | 831 | 0.61 / 0.59 = 1.03 | between_species_paralog: ENSGACG00000005617 | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q3 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q3 | +/+ | ENSP00000249499 | HOXD9 | q3 | q3 | 146 | groupXI | 1626200 | 1626433 | 1 | 1 | 22.8% | 85.9% | 234 | 0.6 / 0.78 = 0.77 | noComparaHits: | 4 |
| --- | q4 | gapp | - | - | - | q4 | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q5 | +/+ | ENSP00000315949 | HOXD8 | q5 | q5 | 115 | groupXI | 1655562 | 1655828 | 1 | 1 | 32.1% | 64.5% | 267 | 0.49 / 0.78 = 0.63 | noComparaHits: ENSGACG00000005623 | 3 |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 253.7 | groupXI | 1677309 | 1679610 | 1 | 2 | 95.7% | 51.8% | 795 | 0.51 / 0.53 = 0.96 | between_species_paralog: ENSGACG00000005628 | 3 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 312 | groupXI | 1709777 | 1713575 | 1 | 2 | 79.9% | 49.7% | 1170 | 0.56 / 0.65 = 0.86 | between_species_paralog: ENSGACG00000005631 | 3 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 118 | groupXI | 1740279 | 1740566 | 1 | 1 | 32.3% | 61.3% | 288 | 0.79 / 0.81 = 0.98 | noComparaHits: ENSGACG00000005635 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 45 (region 4 for Tnig)
 |
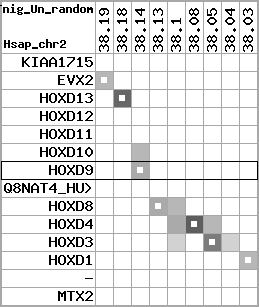 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l Un_random -i 38028536:38193032 -f 12e4 some_geneID_to_be_selected
scoreRatio_sum=5.98 log_scoreRatio_sum=-1.18 focal_scoreRatio_sum=1.88
AlnScore=1976 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 157.2 | Un_random | 38191031 | 38193032 | 1 | 2 | 30.9% | 51.9% | 432 | 0.62 / 0.8 = 0.78 | noComparaHits: GSTENG00020458001
; HOXB-EVX |
3 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 264 | Un_random | 38176504 | 38178141 | -1 | 2 | 89.6% | 47.2% | 870 | 0.61 / 0.59 = 1.03 | between_species_paralog: GSTENG00020457001
; HOXB13 |
2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q3 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q3 | +/+ | ENSP00000249499 | HOXD9 | q3 | q3 | 143 | Un_random | 38140064 | 38140297 | -1 | 1 | 22.8% | 84.6% | 234 | 0.6 / 0.79 = 0.76 | noComparaHits: GSTENG00020456001 | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q4 | +/+ | ENSP00000315949 | HOXD8 | q4 | q4 | 115 | Un_random | 38129985 | 38130416 | -1 | 1 | 66.2% | 38.3% | 432 | 0.49 / 0.78 = 0.63 | between_species_paralog: HOXB8 | 2 |
| --- | q5 | gapp | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q6 | +/+ | ENSP00000302548 | HOXD4 | q6 | q6 | 252 | Un_random | 38083014 | 38085184 | -1 | 2 | 89.8% | 55.1% | 723 | 0.51 / 0.54 = 0.94 | between_species_paralog: HOXB4 | 3 |
| p11 | q7 | +/+ | ENSP00000249440 | HOXD3 | q7 | q7 | 319.7 | Un_random | 38049854 | 38053317 | -1 | 3 | 99.3% | 43.6% | 1473 | 0.56 / 0.65 = 0.86 | between_species_paralog: GSTENG00020452001 | 4 |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q9 | +/+ | ENSP00000328598 | HOXD1 | q9 | q9 | 114 | Un_random | 38028536 | 38028751 | -1 | 1 | 21.6% | 77.8% | 216 | 0.79 / 0.81 = 0.98 | noComparaHits: GSTENG00005035001
; HOXB1 |
3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 46 (region 3 for Ggal)
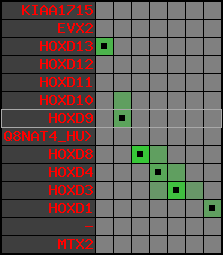 |
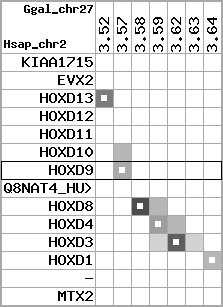 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l 27 -i 3518838:3641292 -f 92e3 some_geneID_to_be_selected
scoreRatio_sum=5.41 log_scoreRatio_sum=-0.68 focal_scoreRatio_sum=1.83
AlnScore=1924 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 225 | 27 | 3518838 | 3521210 | 1 | 2 | 71.3% | 48.4% | 720 | 0.61 / 0.65 = 0.94 | between_species_paralog: HOXB13 | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 144 | 27 | 3567989 | 3568210 | 1 | 1 | 21.6% | 89.2% | 222 | 0.6 / 0.78 = 0.77 | between_species_paralog: HOXB9 | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q3 | +/+ | ENSP00000315949 | HOXD8 | q3 | q3 | 279 | 27 | 3578995 | 3580514 | 1 | 2 | 99.7% | 50.7% | 744 | 0.49 / 0.48 = 1.02 | ortholog_one2many: HXB8_CHICK | 2 |
| p10 | q4 | +/+ | ENSP00000302548 | HOXD4 | q4 | q4 | 130 | 27 | 3586315 | 3586608 | 1 | 1 | 36.1% | 68.4% | 294 | 0.51 / 0.76 = 0.67 | noComparaHits: HXB5_CHICK
; HXB8_CHICK |
4 |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 406 | 27 | 3615199 | 3617444 | 1 | 2 | 96.3% | 52.2% | 1200 | 0.56 / 0.55 = 1.02 | between_species_paralog: HXB3_CHICK | 3 |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q7 | +/+ | ENSP00000328598 | HOXD1 | q7 | q7 | 124 | 27 | 3640885 | 3641226 | 1 | 1 | 38.7% | 55.1% | 342 | 0.79 / 0.8 = 0.99 | noComparaHits: HXB1_CHICK
; HXB8_CHICK |
2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 47 (region 4 for Olat)
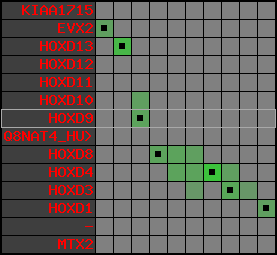 |
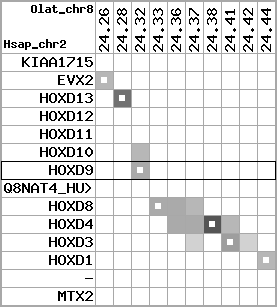 |
 |
retrieve target region: getEnsemblProteins.pl -h Oryzias_latipes -l 8 -i 24254536:24441881 -f 14e4 some_geneID_to_be_selected
scoreRatio_sum=5.91 log_scoreRatio_sum=-1.26 focal_scoreRatio_sum=1.86
AlnScore=1899 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Oryzias_latipes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 161 | 8 | 24254536 | 24257383 | -1 | 2 | 33% | 50.3% | 471 | 0.62 / 0.8 = 0.78 | noComparaHits: Q2WFT1_ORYLA | 3 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 254 | 8 | 24280310 | 24281755 | 1 | 2 | 90.1% | 48.4% | 828 | 0.61 / 0.6 = 1.02 | between_species_paralog: Q3V614_ORYLA | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q3 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q3 | +/+ | ENSP00000249499 | HOXD9 | q3 | q3 | 142 | 8 | 24318538 | 24318762 | 1 | 1 | 21.9% | 86.7% | 225 | 0.6 / 0.79 = 0.76 | between_species_paralog: Q3V615_ORYLA | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q4 | +/+ | ENSP00000315949 | HOXD8 | q4 | q4 | 122 | 8 | 24328909 | 24329400 | 1 | 1 | 70% | 38.4% | 492 | 0.49 / 0.77 = 0.64 | noComparaHits: Q3V617_ORYLA | 2 |
| --- | q5 | gapp | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 257.4 | 8 | 24381089 | 24383038 | 1 | 2 | 96.5% | 53.8% | 774 | 0.51 / 0.53 = 0.96 | between_species_paralog: Q3V619_ORYLA | 2 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 245 | 8 | 24414385 | 24415236 | 1 | 1 | 59% | 52.6% | 852 | 0.56 / 0.73 = 0.77 | between_species_paralog: Q3V620_ORYLA | 3 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 116 | 8 | 24441627 | 24441854 | 1 | 1 | 23.8% | 74.4% | 228 | 0.79 / 0.81 = 0.98 | noComparaHits: Q2WFU0_ORYLA | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 48 (region 4 for Xtro)
 |
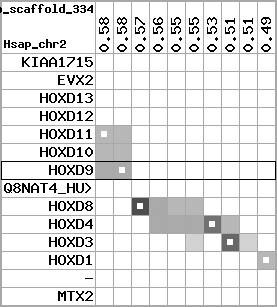 |
 |
retrieve target region: getEnsemblProteins.pl -h Xenopus_tropicalis -l scaffold_334 -i 486988:589462 -f 77e3 some_geneID_to_be_selected
scoreRatio_sum=5.25 log_scoreRatio_sum=-0.87 focal_scoreRatio_sum=1.89
AlnScore=1870 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Xenopus_tropicalis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q1 | +/+ | ENSP00000249504 | HOXD11 | q1,q2 | q1 | 100 | scaffold_334 | 584781 | 584996 | -1 | 1 | 21.3% | 61.1% | 216 | 0.53 / 0.82 = 0.65 | noComparaHits: HOXB9 | 6 |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q1,q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q1,q2 | q2 | 143 | scaffold_334 | 579382 | 579603 | -1 | 1 | 21.6% | 86.5% | 222 | 0.6 / 0.79 = 0.76 | between_species_paralog: HOXB9 | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q3 | +/+ | ENSP00000315949 | HOXD8 | q3 | q3 | 278 | scaffold_334 | 572897 | 573865 | -1 | 2 | 99.3% | 50.8% | 747 | 0.49 / 0.49 = 1 | noComparaHits: ENSXETG00000021993 | 2 |
| --- | q4 | gapp | - | - | - | q4 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q5 | gapp | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 215.6 | scaffold_334 | 533069 | 534165 | -1 | 2 | 88.2% | 53.1% | 642 | 0.51 / 0.6 = 0.85 | between_species_paralog: hoxb4 | 4 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 397 | scaffold_334 | 512901 | 514779 | -1 | 2 | 98.6% | 51.9% | 1185 | 0.56 / 0.56 = 1 | between_species_paralog: hoxb3 | 3 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 122 | scaffold_334 | 487045 | 487281 | -1 | 1 | 24.1% | 74.7% | 237 | 0.79 / 0.8 = 0.99 | noComparaHits: ENSXETG00000021964 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 49 (region 3 for Btau)
 |
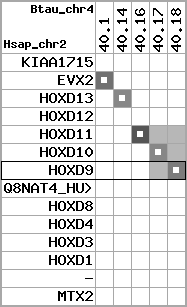 |
 |
retrieve target region: getEnsemblProteins.pl -h Bos_taurus -l 4 -i 40097176:40179364 -f 62e3 some_geneID_to_be_selected
scoreRatio_sum=4.73 log_scoreRatio_sum=-0.28 focal_scoreRatio_sum=2.05
AlnScore=1848 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Bos_taurus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 320 | 4 | 40097176 | 40098505 | -1 | 2 | 67.6% | 55.4% | 804 | 0.62 / 0.6 = 1.03 | between_species_paralog: EVX1 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 207.1 | 4 | 40142887 | 40144076 | 1 | 2 | 44.8% | 64.6% | 474 | 0.61 / 0.68 = 0.9 | between_species_paralog: HOXA13 | 3 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 262 | 4 | 40157129 | 40159489 | 1 | 2 | 83.7% | 54% | 738 | 0.53 / 0.52 = 1.02 | between_species_paralog: HOXA11 | 1 |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 214.7 | 4 | 40169001 | 40171084 | 1 | 2 | 43.8% | 71% | 465 | 0.58 / 0.69 = 0.84 | noComparaHits: HOXA10 | 2 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 244.5 | 4 | 40177480 | 40179364 | 1 | 2 | 88.3% | 47.5% | 834 | 0.6 / 0.64 = 0.94 | between_species_paralog: HOXA9 | 3 |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 50 (region 4 for Mmul)
 |
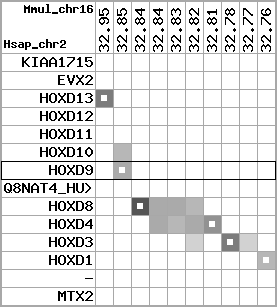 |
 |
retrieve target region: getEnsemblProteins.pl -h Macaca_mulatta -l 16 -i 32758415:32953191 -f 15e4 some_geneID_to_be_selected
scoreRatio_sum=5.27 log_scoreRatio_sum=-0.81 focal_scoreRatio_sum=1.8
AlnScore=1826 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Macaca_mulatta
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 228 | 16 | 32951572 | 32953191 | -1 | 2 | 71.3% | 47.7% | 669 | 0.61 / 0.64 = 0.95 | between_species_paralog: HOXB13 | 2 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 145 | 16 | 32852664 | 32852885 | -1 | 1 | 21.6% | 89.2% | 222 | 0.6 / 0.78 = 0.77 | between_species_paralog: HOXB9 | 5 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q3 | +/+ | ENSP00000315949 | HOXD8 | q3 | q3 | 269 | 16 | 32842665 | 32844111 | -1 | 2 | 94.8% | 53.3% | 672 | 0.49 / 0.5 = 0.98 | between_species_paralog: HOXB8 | 3 |
| --- | q4 | gapp | - | - | - | q4 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q5 | gapp | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q7 | +/+ | ENSP00000302548 | HOXD4 | q7 | q7 | 160 | 16 | 32806520 | 32806789 | -1 | 1 | 35.3% | 83.3% | 270 | 0.51 / 0.7 = 0.73 | between_species_paralog: HOXB4 | 3 |
| p11 | q8 | +/+ | ENSP00000249440 | HOXD3 | q8 | q8 | 311 | 16 | 32779070 | 32781210 | -1 | 2 | 85.6% | 47.5% | 1080 | 0.56 / 0.66 = 0.85 | noComparaHits: | 3 |
| --- | q9 | gapp | - | - | - | q9 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q10 | +/+ | ENSP00000328598 | HOXD1 | q10 | q10 | 120 | 16 | 32758415 | 32758756 | -1 | 1 | 33.8% | 58.5% | 342 | 0.79 / 0.8 = 0.99 | noComparaHits: HOXB1 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 51 (region 3 for Mdom)
 |
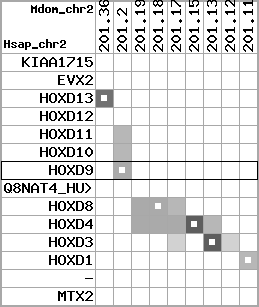 |
 |
retrieve target region: getEnsemblProteins.pl -h Monodelphis_domestica -l 2 -i 201105070:201365718 -f 20e4 some_geneID_to_be_selected
scoreRatio_sum=5.28 log_scoreRatio_sum=-0.84 focal_scoreRatio_sum=1.77
AlnScore=1823 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Monodelphis_domestica
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 239 | 2 | 201363760 | 201365718 | -1 | 2 | 97% | 39.1% | 900 | 0.61 / 0.63 = 0.97 | between_species_paralog: HOXB13 | 3 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 145 | 2 | 201204831 | 201205052 | -1 | 1 | 21.6% | 89.2% | 222 | 0.6 / 0.78 = 0.77 | between_species_paralog: HOXB9 | 2 |
| --- | q3 | gapp | - | - | - | q3 | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q4 | +/+ | ENSP00000315949 | HOXD8 | q4 | q4 | 113 | 2 | 201175565 | 201175924 | -1 | 1 | 41.7% | 51.2% | 360 | 0.49 / 0.79 = 0.62 | noComparaHits: HOXB6 | 3 |
| --- | q5 | gapp | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q6 | +/+ | ENSP00000302548 | HOXD4 | q6 | q6 | 252.4 | 2 | 201153490 | 201155052 | -1 | 2 | 91.4% | 57.1% | 693 | 0.51 / 0.54 = 0.94 | between_species_paralog: HOXB4 | 2 |
| p11 | q7 | +/+ | ENSP00000249440 | HOXD3 | q7 | q7 | 399 | 2 | 201123983 | 201126209 | -1 | 2 | 95.1% | 53.7% | 1203 | 0.56 / 0.56 = 1 | between_species_paralog: HOXB3 | 2 |
| --- | q8 | gapp | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q9 | +/+ | ENSP00000328598 | HOXD1 | q9 | q9 | 115 | 2 | 201105136 | 201105375 | -1 | 1 | 24.4% | 71.6% | 240 | 0.79 / 0.81 = 0.98 | noComparaHits: HOXB1 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 52 (region 6 for Gacu)
 |
 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l groupVI -i 16143064:16188136 -f 34e3 some_geneID_to_be_selected
scoreRatio_sum=6.6 log_scoreRatio_sum=1.21 focal_scoreRatio_sum=1.61
AlnScore=1758 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 288.8 | groupVI | 16143064 | 16145858 | -1 | 4 | 43.9% | 51.8% | 825 | 1 / 0.63 = 1.59 | ortholog_one2many: ENSGACG00000011901 | 1 |
| p2 | --- | gapq | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 134 | groupVI | 16162358 | 16162570 | 1 | 1 | 20.8% | 87.3% | 213 | 0.6 / 0.8 = 0.75 | noComparaHits: ENSGACG00000011902 | 6 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q3 | +/+ | ENSP00000302548 | HOXD4 | q3 | q3 | 208.9 | groupVI | 16181214 | 16182596 | 1 | 2 | 88.2% | 49% | 684 | 0.51 / 0.62 = 0.82 | noComparaHits: ENSGACG00000011907 | 5 |
| p11 | --- | gapq | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gapq | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q4 | +/+ | ENSP00000249442 | MTX2 | q4 | q4 | 381.3 | groupVI | 16186214 | 16188136 | 1 | 6 | 85.9% | 61.3% | 891 | 0.55 / 0.16 = 3.44 | ortholog_one2one: ENSGACG00000011909 | 1 |
region 53 (region 6 for Drer)
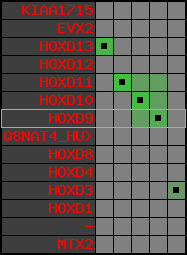 |
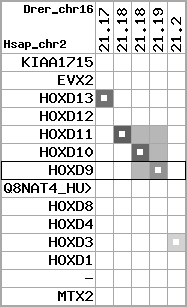 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 16 -i 21167839:21201078 -f 25e3 some_geneID_to_be_selected
scoreRatio_sum=4.44 log_scoreRatio_sum=-0.65 focal_scoreRatio_sum=2
AlnScore=1631 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 240 | 16 | 21167839 | 21168969 | 1 | 2 | 78.2% | 51.9% | 714 | 0.61 / 0.62 = 0.98 | between_species_paralog: hoxa13b | 4 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 252 | 16 | 21177655 | 21179247 | 1 | 2 | 85.8% | 49.5% | 765 | 0.53 / 0.54 = 0.98 | between_species_paralog: hoxa11b | 2 |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 280 | 16 | 21183004 | 21185153 | 1 | 2 | 74.7% | 59.7% | 759 | 0.58 / 0.59 = 0.98 | noComparaHits: hoxa10b | 3 |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 203.6 | 16 | 21188212 | 21189729 | 1 | 2 | 56.1% | 57% | 543 | 0.6 / 0.7 = 0.86 | between_species_paralog: hoxa9b | 2 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gapq | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 105 | 16 | 21200887 | 21201078 | 1 | 1 | 14.8% | 71.9% | 192 | 0.56 / 0.88 = 0.64 | noComparaHits: hoxa2b | 5 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 54 (region 5 for Olat)
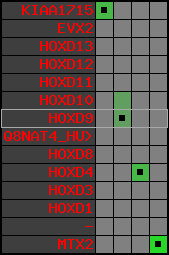 |
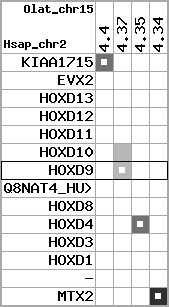 |
 |
retrieve target region: getEnsemblProteins.pl -h Oryzias_latipes -l 15 -i 4337486:4400015 -f 47e3 some_geneID_to_be_selected
scoreRatio_sum=4.74 log_scoreRatio_sum=0.45 focal_scoreRatio_sum=1.38
AlnScore=1586 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Oryzias_latipes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 297.7 | 15 | 4394562 | 4400015 | 1 | 5 | 57% | 51.4% | 849 | 1 / 0.62 = 1.61 | ortholog_one2many: ENSORLG00000001366 | 1 |
| p2 | --- | gapq | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 133 | 15 | 4374398 | 4374607 | -1 | 1 | 20.5% | 87.1% | 210 | 0.6 / 0.8 = 0.75 | noComparaHits: Q9PVR1_ORYLA | 6 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q3 | +/+ | ENSP00000302548 | HOXD4 | q3 | q3 | 217.9 | 15 | 4350793 | 4351949 | -1 | 2 | 91% | 52.1% | 699 | 0.51 / 0.6 = 0.85 | noComparaHits: Q3V5Z3_ORYLA | 5 |
| p11 | --- | gapq | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gapq | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q4 | +/+ | ENSP00000249442 | MTX2 | q4 | q4 | 290.7 | 15 | 4337486 | 4342821 | -1 | 4 | 65.8% | 67.2% | 603 | 0.55 / 0.36 = 1.53 | ortholog_one2one: ENSORLG00000001332 | 1 |
region 55 (region 5 for Trub)
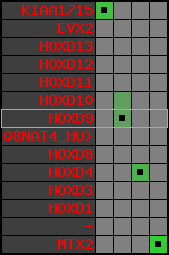 |
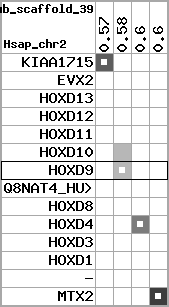 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_39 -i 568162:603585 -f 27e3 some_geneID_to_be_selected
scoreRatio_sum=4.76 log_scoreRatio_sum=0.42 focal_scoreRatio_sum=1.38
AlnScore=1570 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 348.3 | scaffold_39 | 568162 | 571367 | -1 | 6 | 55.1% | 54.5% | 960 | 1 / 0.55 = 1.82 | noComparaHits: NEWSINFRUG00000161011
; NEWSINFRUG00000164545 ; NEWSINFRUG00000148695 |
1 |
| p2 | --- | gapq | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 132 | scaffold_39 | 584702 | 584911 | 1 | 1 | 20.5% | 85.7% | 210 | 0.6 / 0.8 = 0.75 | noComparaHits: Q4VN43_FUGRU | 6 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q3 | +/+ | ENSP00000302548 | HOXD4 | q3 | q3 | 201.5 | scaffold_39 | 597373 | 598598 | 1 | 2 | 83.9% | 47.6% | 654 | 0.51 / 0.63 = 0.81 | noComparaHits: NEWSINFRUG00000162165 | 5 |
| p11 | --- | gapq | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gapq | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q4 | +/+ | ENSP00000249442 | MTX2 | q4 | q4 | 268.7 | scaffold_39 | 601883 | 603036 | 1 | 4 | 82.5% | 49.5% | 876 | 0.55 / 0.4 = 1.38 | ortholog_one2one: NEWSINFRUG00000144217 | 1 |
region 56 (region 7 for Gacu)
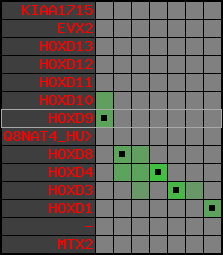 |
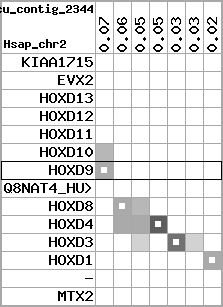 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_2344 -i 19871:66165 -f 35e3 some_geneID_to_be_selected
scoreRatio_sum=4.27 log_scoreRatio_sum=-0.85 focal_scoreRatio_sum=1.57
AlnScore=1459 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q1 | +/+ | ENSP00000249499 | HOXD9 | q1 | q1 | 144 | contig_2344 | 65914 | 66156 | -1 | 1 | 23.7% | 81.5% | 243 | 0.6 / 0.78 = 0.77 | noComparaHits: | 5 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q2 | +/+ | ENSP00000315949 | HOXD8 | q2 | q2 | 118 | contig_2344 | 60643 | 60930 | -1 | 1 | 32.4% | 61.5% | 288 | 0.49 / 0.78 = 0.63 | noComparaHits: ENSGACG00000007112 | 2 |
| --- | q3 | gapp | - | - | - | q3 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q4 | +/+ | ENSP00000302548 | HOXD4 | q4 | q4 | 250.6 | contig_2344 | 45010 | 46274 | -1 | 2 | 95.3% | 53.8% | 732 | 0.51 / 0.54 = 0.94 | noComparaHits: ENSGACG00000007100 | 4 |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 365.3 | contig_2344 | 30826 | 34266 | -1 | 2 | 96.1% | 49.2% | 1245 | 0.56 / 0.6 = 0.93 | noComparaHits: ENSGACG00000007094 | 2 |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q7 | +/+ | ENSP00000328598 | HOXD1 | q7 | q7 | 127 | contig_2344 | 19871 | 20197 | -1 | 1 | 32% | 60.6% | 327 | 0.79 / 0.79 = 1 | noComparaHits: ENSGACG00000007085 | 1 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 57 (region 6 for Olat)
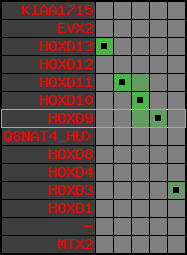 |
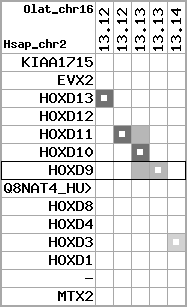 |
 |
retrieve target region: getEnsemblProteins.pl -h Oryzias_latipes -l 16 -i 13115399:13137005 -f 16e3 some_geneID_to_be_selected
scoreRatio_sum=4.18 log_scoreRatio_sum=-0.95 focal_scoreRatio_sum=1.83
AlnScore=1430 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Oryzias_latipes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 240 | 16 | 13115399 | 13116156 | 1 | 2 | 68.4% | 57.5% | 633 | 0.61 / 0.62 = 0.98 | between_species_paralog: Q3V623_ORYLA | 4 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 213.7 | 16 | 13120158 | 13121649 | 1 | 2 | 65.4% | 49.6% | 744 | 0.53 / 0.61 = 0.87 | between_species_paralog: Q3V624_ORYLA | 4 |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 259 | 16 | 13127649 | 13129140 | 1 | 2 | 95.3% | 48% | 927 | 0.58 / 0.62 = 0.94 | noComparaHits: Q3V625_ORYLA | 3 |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 134 | 16 | 13131568 | 13131795 | 1 | 1 | 22.2% | 78.9% | 228 | 0.6 / 0.8 = 0.75 | between_species_paralog: Q3V626_ORYLA | 5 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gapq | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 106 | 16 | 13136775 | 13137005 | 1 | 1 | 17.8% | 63.6% | 231 | 0.56 / 0.88 = 0.64 | noComparaHits: Q3V627_ORYLA | 7 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 58 (region 4 for Btau)
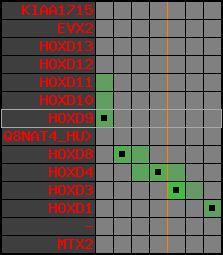 |
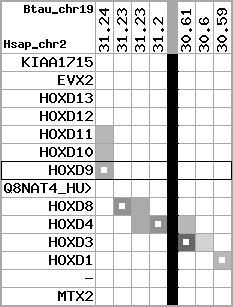 |
 |
retrieve target region: getEnsemblProteins.pl -h Bos_taurus -l 19 -i 30587828:31239752 -f 49e4 some_geneID_to_be_selected
scoreRatio_sum=4.11 log_scoreRatio_sum=-1.02 focal_scoreRatio_sum=1.53
AlnScore=1342 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Bos_taurus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q1 | +/+ | ENSP00000249499 | HOXD9 | q1 | q1 | 145 | 19 | 31239444 | 31239665 | -1 | 1 | 21.6% | 89.2% | 222 | 0.6 / 0.78 = 0.77 | between_species_paralog: HOXB9 | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q2 | +/+ | ENSP00000315949 | HOXD8 | q2 | q2 | 150 | 19 | 31230187 | 31230462 | -1 | 1 | 33.4% | 78.4% | 276 | 0.49 / 0.72 = 0.68 | noComparaHits: | 3 |
| --- | q3 | gapp | - | - | - | q3 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q4 | +/+ | ENSP00000302548 | HOXD4 | q4 | q4 | 160 | 19 | 31195664 | 31195918 | -1 | 1 | 33.3% | 87.1% | 255 | 0.51 / 0.7 = 0.73 | noComparaHits: HOXB5 | 4 |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 368 | 19 | 30606318 | 30608469 | -1 | 2 | 76.2% | 57.5% | 1002 | 0.56 / 0.59 = 0.95 | between_species_paralog: HOXB3 | 3 |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q7 | +/+ | ENSP00000328598 | HOXD1 | q7 | q7 | 118 | 19 | 30587828 | 30588133 | -1 | 1 | 32.3% | 60.4% | 306 | 0.79 / 0.81 = 0.98 | noComparaHits: HOXB1 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 59 (region 8 for Gacu)
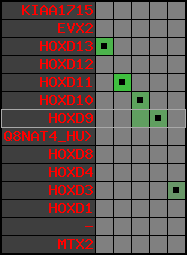 |
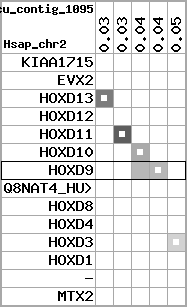 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_1095 -i 26570:49693 -f 17e3 some_geneID_to_be_selected
scoreRatio_sum=4.07 log_scoreRatio_sum=-1.08 focal_scoreRatio_sum=1.77
AlnScore=1333 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 237 | contig_1095 | 26570 | 27476 | 1 | 2 | 67.8% | 55.4% | 627 | 0.61 / 0.63 = 0.97 | noComparaHits: ENSGACG00000008296 | 5 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 249 | contig_1095 | 32620 | 34350 | 1 | 2 | 97.3% | 43.5% | 861 | 0.53 / 0.55 = 0.96 | noComparaHits: ENSGACG00000008297 | 3 |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 154 | contig_1095 | 39804 | 40055 | 1 | 1 | 24.7% | 88.1% | 252 | 0.58 / 0.77 = 0.75 | noComparaHits: ENSGACG00000008303 | 4 |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 130 | contig_1095 | 43335 | 43559 | 1 | 1 | 21.9% | 77.3% | 225 | 0.6 / 0.8 = 0.75 | noComparaHits: ENSGACG00000008310 | 7 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gapq | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 108 | contig_1095 | 49436 | 49675 | 1 | 1 | 18.5% | 62.5% | 240 | 0.56 / 0.88 = 0.64 | noComparaHits: ENSGACG00000008314 | 6 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 60 (region 9 for Gacu)
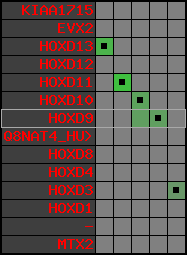 |
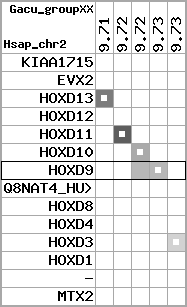 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l groupXX -i 9710810:9733933 -f 17e3 some_geneID_to_be_selected
scoreRatio_sum=4.07 log_scoreRatio_sum=-1.08 focal_scoreRatio_sum=1.77
AlnScore=1333 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 237 | groupXX | 9710810 | 9711716 | 1 | 2 | 67.8% | 55.4% | 627 | 0.61 / 0.63 = 0.97 | between_species_paralog: ENSGACG00000008296 | 5 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 249 | groupXX | 9716860 | 9718590 | 1 | 2 | 97.3% | 43.5% | 861 | 0.53 / 0.55 = 0.96 | between_species_paralog: ENSGACG00000008297 | 3 |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 154 | groupXX | 9724044 | 9724295 | 1 | 1 | 24.7% | 88.1% | 252 | 0.58 / 0.77 = 0.75 | noComparaHits: ENSGACG00000008303 | 4 |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 130 | groupXX | 9727575 | 9727799 | 1 | 1 | 21.9% | 77.3% | 225 | 0.6 / 0.8 = 0.75 | between_species_paralog: ENSGACG00000008310 | 7 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gapq | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 108 | groupXX | 9733676 | 9733915 | 1 | 1 | 18.5% | 62.5% | 240 | 0.56 / 0.88 = 0.64 | noComparaHits: ENSGACG00000008314 | 6 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 61 (region 10 for Gacu)
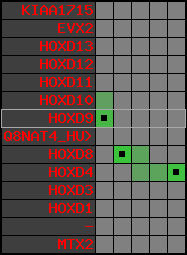 |
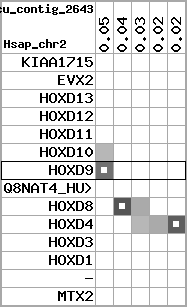 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_2643 -i 4952:46955 -f 32e3 some_geneID_to_be_selected
scoreRatio_sum=2.93 log_scoreRatio_sum=-0.06 focal_scoreRatio_sum=1.54
AlnScore=1318 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q1 | +/+ | ENSP00000249499 | HOXD9 | q1 | q1 | 254 | contig_2643 | 45188 | 46955 | -1 | 2 | 66.7% | 58.1% | 591 | 0.6 / 0.62 = 0.97 | noComparaHits: ENSGACG00000009396 | 1 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q2 | +/+ | ENSP00000315949 | HOXD8 | q2 | q2 | 274 | contig_2643 | 39207 | 41140 | -1 | 2 | 90.3% | 54.9% | 672 | 0.49 / 0.49 = 1 | noComparaHits: ENSGACG00000009401 | 1 |
| --- | q3 | gapp | - | - | - | q3 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q4 | gapp | - | - | - | q4 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q5 | +/+ | ENSP00000302548 | HOXD4 | q5 | q5 | 257.1 | contig_2643 | 14513 | 15813 | -1 | 2 | 100% | 53.8% | 792 | 0.51 / 0.53 = 0.96 | noComparaHits: ENSGACG00000009421 | 2 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 62 (region 6 for Trub)
 |
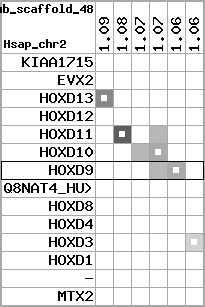 |
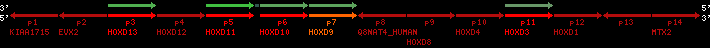 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_48 -i 1057087:1085777 -f 22e3 some_geneID_to_be_selected
scoreRatio_sum=4.04 log_scoreRatio_sum=-1.11 focal_scoreRatio_sum=1.77
AlnScore=1305 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 210 | scaffold_48 | 1084995 | 1085777 | -1 | 1 | 70.7% | 45.9% | 783 | 0.61 / 0.67 = 0.91 | between_species_paralog: NEWSINFRUG00000132089 | 6 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 255 | scaffold_48 | 1078635 | 1080372 | -1 | 2 | 94.7% | 44.7% | 858 | 0.53 / 0.54 = 0.98 | between_species_paralog: NEWSINFRUG00000138060 | 2 |
| --- | q3 | gapp | - | - | - | q3 | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | q4 | +/+ | ENSP00000249501 | HOXD10 | q4 | q4 | 155 | scaffold_48 | 1066192 | 1066455 | -1 | 1 | 26.2% | 85.4% | 264 | 0.58 / 0.77 = 0.75 | noComparaHits: NEWSINFRUG00000138061 | 4 |
| p7 | q5 | +/+ | ENSP00000249499 | HOXD9 | q5 | q5 | 137 | scaffold_48 | 1063108 | 1063341 | -1 | 1 | 22.8% | 79.5% | 234 | 0.6 / 0.79 = 0.76 | between_species_paralog: NEWSINFRUG00000138062 | 5 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gapq | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q6 | +/+ | ENSP00000249440 | HOXD3 | q6 | q6 | 108 | scaffold_48 | 1057105 | 1057341 | -1 | 1 | 18.3% | 62% | 237 | 0.56 / 0.88 = 0.64 | noComparaHits: NEWSINFRUG00000138063 | 6 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 63 (region 5 for Tnig)
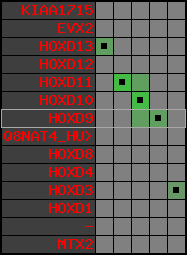 |
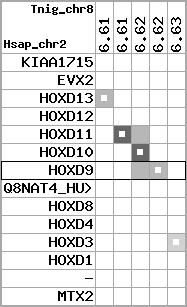 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l 8 -i 6607022:6627162 -f 15e3 some_geneID_to_be_selected
scoreRatio_sum=4.04 log_scoreRatio_sum=-1.12 focal_scoreRatio_sum=1.83
AlnScore=1305 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 115 | 8 | 6607043 | 6607240 | 1 | 1 | 19.7% | 84.8% | 198 | 0.61 / 0.82 = 0.74 | between_species_paralog: HOXAb13
; GSTENG00034119001 |
4 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 229.3 | 8 | 6611319 | 6613072 | 1 | 2 | 44.1% | 72.8% | 450 | 0.53 / 0.58 = 0.91 | between_species_paralog: GSTENG00034118001
; HOXAb11 |
3 |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 286.3 | 8 | 6616572 | 6618000 | 1 | 3 | 96.5% | 50% | 924 | 0.58 / 0.58 = 1 | noComparaHits: HOXAb10
; GSTENG00034117001 |
2 |
| p7 | q4 | +/+ | ENSP00000249499 | HOXD9 | q4 | q4 | 135 | 8 | 6620992 | 6621216 | 1 | 1 | 21.9% | 81.3% | 225 | 0.6 / 0.8 = 0.75 | between_species_paralog: HOXAb9 | 5 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gapq | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 104 | 8 | 6626881 | 6627078 | 1 | 1 | 15.3% | 69.7% | 198 | 0.56 / 0.88 = 0.64 | noComparaHits: HOXAb2
; GSTENG00034116001 |
7 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 64 (region 11 for Gacu)
 |
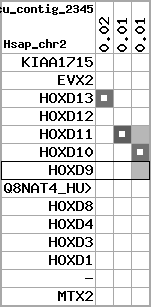 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_2345 -i 57:23907 -f 18e3 some_geneID_to_be_selected
scoreRatio_sum=2.95 log_scoreRatio_sum=-0.04 focal_scoreRatio_sum=1.01
AlnScore=1224 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 249 | contig_2345 | 22662 | 23907 | -1 | 2 | 72.2% | 56.9% | 687 | 0.61 / 0.61 = 1 | noComparaHits: Q9PWC4_GASAC | 4 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q2 | +/+ | ENSP00000249504 | HOXD11 | q2 | q2 | 251 | contig_2345 | 11804 | 13419 | -1 | 2 | 84.6% | 50.2% | 807 | 0.53 / 0.54 = 0.98 | noComparaHits: ENSGACG00000007132 | 2 |
| p6 | q3 | +/+ | ENSP00000249501 | HOXD10 | q3 | q3 | 271 | contig_2345 | 4545 | 6319 | -1 | 2 | 97.9% | 49.4% | 1038 | 0.58 / 0.6 = 0.97 | noComparaHits: ENSGACG00000007128 | 2 |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | q3 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 65 (region 6 for Tnig)
 |
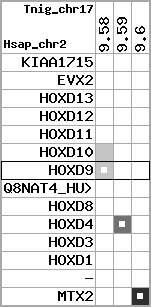 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l 17 -i 9564414:9598839 -f 26e3 some_geneID_to_be_selected
scoreRatio_sum=3.21 log_scoreRatio_sum=0 focal_scoreRatio_sum=1.13
AlnScore=1192 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q1 | +/+ | ENSP00000249499 | HOXD9 | q1 | q1 | 113 | 17 | 9576378 | 9576557 | 1 | 1 | 17.5% | 85% | 180 | 0.6 / 0.83 = 0.72 | noComparaHits: GSTENG00018723001
; HOXDb9 |
9 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q2 | +/+ | ENSP00000302548 | HOXD4 | q2 | q2 | 206.5 | 17 | 9593474 | 9594639 | 1 | 2 | 85.5% | 48.9% | 666 | 0.51 / 0.62 = 0.82 | noComparaHits: HOXDb4
; GSTENG00018723001 |
5 |
| p11 | --- | gapq | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gapq | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q3 | +/+ | ENSP00000249442 | MTX2 | q3 | q3 | 301.6 | 17 | 9597464 | 9598839 | 1 | 5 | 68.4% | 60.3% | 696 | 0.55 / 0.33 = 1.67 | ortholog_one2one: GSTENG00018722001 | 1 |
region 66 (region 5 for Btau)
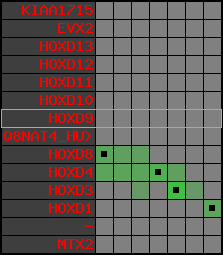 |
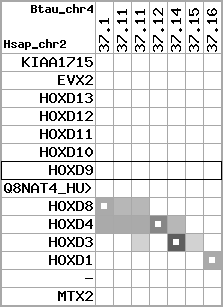 |
 |
retrieve target region: getEnsemblProteins.pl -h Bos_taurus -l 4 -i 37099203:37159173 -f 45e3 some_geneID_to_be_selected
scoreRatio_sum=3.42 log_scoreRatio_sum=-0.71 focal_scoreRatio_sum=0.77
AlnScore=1158 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Bos_taurus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q1 | +/+ | ENSP00000315949 | HOXD8 | q1 | q1 | 114 | 4 | 37099440 | 37099640 | 1 | 1 | 23.1% | 76.1% | 201 | 0.49 / 0.79 = 0.62 | noComparaHits: HOXA7 | 4 |
| --- | q2 | gapp | - | - | - | q2 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q3 | gapp | - | - | - | q3 | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q4 | +/+ | ENSP00000302548 | HOXD4 | q4 | q4 | 170 | 4 | 37124791 | 37125126 | 1 | 1 | 43.1% | 75.9% | 336 | 0.51 / 0.69 = 0.74 | between_species_paralog: HOXA4 | 3 |
| p11 | q5 | +/+ | ENSP00000249440 | HOXD3 | q5 | q5 | 426.7 | 4 | 37142956 | 37145704 | 1 | 3 | 96.3% | 53.5% | 1254 | 0.56 / 0.53 = 1.06 | between_species_paralog: HOXA3 | 2 |
| --- | q6 | gapp | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q7 | +/+ | ENSP00000328598 | HOXD1 | q7 | q7 | 130 | 4 | 37158853 | 37159134 | 1 | 1 | 33.8% | 62.2% | 282 | 0.79 / 0.79 = 1 | noComparaHits: HOXA1 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 67 (region 12 for Gacu)
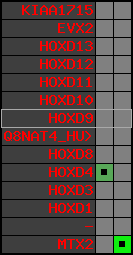 |
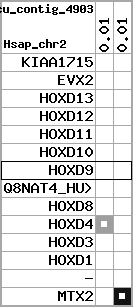 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_4903 -i 5131:10922 -f 43e2 some_geneID_to_be_selected
scoreRatio_sum=4.14 log_scoreRatio_sum=0.88 focal_scoreRatio_sum=0.61
AlnScore=1095 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q1 | +/+ | ENSP00000302548 | HOXD4 | q1 | q1 | 145 | contig_4903 | 10671 | 10922 | -1 | 1 | 32.9% | 77.4% | 252 | 0.51 / 0.73 = 0.7 | noComparaHits: | 6 |
| p11 | --- | gapq | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gapq | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | q2 | +/+ | ENSP00000249442 | MTX2 | q2 | q2 | 381.3 | contig_4903 | 5131 | 7053 | -1 | 6 | 85.9% | 61.3% | 891 | 0.55 / 0.16 = 3.44 | noComparaHits: ENSGACG00000011909 | 1 |
region 68 (region 7 for Drer)
 |
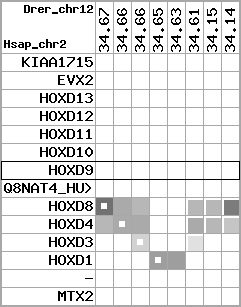 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 12 -i 34135896:34673873 -f 40e4 some_geneID_to_be_selected
scoreRatio_sum=3.14 log_scoreRatio_sum=-1.04 focal_scoreRatio_sum=0.74
AlnScore=937 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q1 | +/+ | ENSP00000315949 | HOXD8 | q1,q8 | q1 | 215.6 | 12 | 34671599 | 34673873 | -1 | 2 | 99% | 43% | 717 | 0.49 / 0.6 = 0.82 | between_species_paralog: hoxb8b | 3 |
| p10 | q2 | +/+ | ENSP00000302548 | HOXD4 | q3,q6 | q2 | 111 | 12 | 34663066 | 34663269 | -1 | 1 | 26.7% | 75% | 204 | 0.51 / 0.79 = 0.65 | noComparaHits: hoxb6b | 10 |
| p11 | q3 | +/+ | ENSP00000249440 | HOXD3 | q3,q6 | q3 | 100 | 12 | 34660365 | 34660568 | -1 | 1 | 15.7% | 70.6% | 204 | 0.56 / 0.89 = 0.63 | noComparaHits: hoxb5b | 7 |
| p12 | q4 | +/+ | ENSP00000328598 | HOXD1 | q5 | q4 | 150 | 12 | 34648904 | 34649358 | -1 | 2 | 41.5% | 57.2% | 372 | 0.79 / 0.76 = 1.04 | noComparaHits: ENSDARG00000054033 | 2 |
| p13 | --- | gapq | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gapq | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q5 | gape | - | - | - | q5 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q6 | gape | - | - | - | q6 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q7 | gape | - | - | - | q7 | - | - | - | - | - | - | - | - | - | - | - | - |
| --- | q8 | gape | - | - | - | q8 | - | - | - | - | - | - | - | - | - | - | - | - |
region 69 (region 7 for Trub)
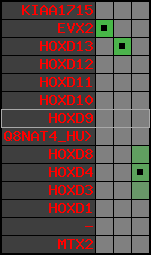 |
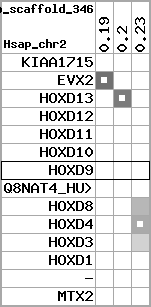 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_346 -i 186282:226492 -f 30e3 some_geneID_to_be_selected
scoreRatio_sum=2.58 log_scoreRatio_sum=-0.49 focal_scoreRatio_sum=0.52
AlnScore=925 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | q1 | +/+ | ENSP00000312385 | EVX2 | q1 | q1 | 307 | scaffold_346 | 186282 | 187332 | -1 | 2 | 62.6% | 54.2% | 699 | 0.62 / 0.62 = 1 | between_species_paralog: NEWSINFRUG00000165271 | 2 |
| p3 | q2 | +/+ | ENSP00000249505 | HOXD13 | q2 | q2 | 219.4 | scaffold_346 | 195273 | 196163 | 1 | 2 | 45.7% | 69% | 474 | 0.61 / 0.66 = 0.92 | between_species_paralog: NEWSINFRUG00000162059 | 5 |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gapq | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gapq | ENSP00000315949 | HOXD8 | q3 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q3 | +/+ | ENSP00000302548 | HOXD4 | q3 | q3 | 123 | scaffold_346 | 226202 | 226399 | 1 | 1 | 25.9% | 87.9% | 198 | 0.51 / 0.77 = 0.66 | noComparaHits: NEWSINFRUG00000149630 | 6 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | q3 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 70 (region 7 for Olat)
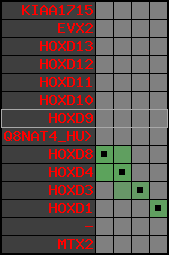 |
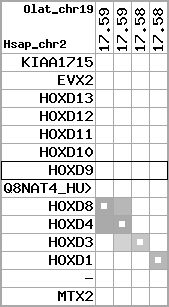 |
 |
retrieve target region: getEnsemblProteins.pl -h Oryzias_latipes -l 19 -i 17578385:17592929 -f 11e3 some_geneID_to_be_selected
scoreRatio_sum=2.9 log_scoreRatio_sum=-1.34 focal_scoreRatio_sum=0.66
AlnScore=755 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Oryzias_latipes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q1 | +/+ | ENSP00000315949 | HOXD8 | q1 | q1 | 113 | 19 | 17592687 | 17592911 | -1 | 1 | 24.8% | 72% | 225 | 0.49 / 0.79 = 0.62 | noComparaHits: Q9PVR5_ORYLA | 3 |
| p10 | q2 | +/+ | ENSP00000302548 | HOXD4 | q2 | q2 | 123 | 19 | 17590258 | 17590455 | -1 | 1 | 25.9% | 87.9% | 198 | 0.51 / 0.77 = 0.66 | noComparaHits: Q3V611_ORYLA | 7 |
| p11 | q3 | +/+ | ENSP00000249440 | HOXD3 | q3 | q3 | 135 | 19 | 17580611 | 17581051 | -1 | 1 | 34.3% | 54.6% | 441 | 0.56 / 0.85 = 0.66 | noComparaHits: Q3V612_ORYLA | 4 |
| p12 | q4 | +/+ | ENSP00000328598 | HOXD1 | q4 | q4 | 113 | 19 | 17578472 | 17578684 | -1 | 1 | 21.6% | 76.1% | 213 | 0.79 / 0.82 = 0.96 | noComparaHits: Q3V613_ORYLA | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 71 (region 8 for Trub)
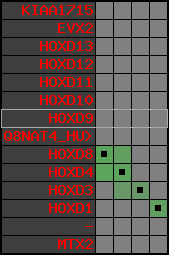 |
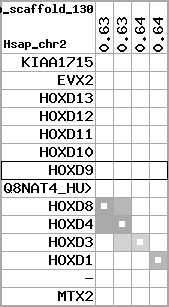 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_130 -i 628804:642318 -f 10e3 some_geneID_to_be_selected
scoreRatio_sum=2.91 log_scoreRatio_sum=-1.33 focal_scoreRatio_sum=0.67
AlnScore=744 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q1 | +/+ | ENSP00000315949 | HOXD8 | q1 | q1 | 114 | scaffold_130 | 628828 | 629052 | 1 | 1 | 24.8% | 73.3% | 225 | 0.49 / 0.79 = 0.62 | noComparaHits: NEWSINFRUG00000156875 | 4 |
| p10 | q2 | +/+ | ENSP00000302548 | HOXD4 | q2 | q2 | 123 | scaffold_130 | 631301 | 631498 | 1 | 1 | 25.9% | 87.9% | 198 | 0.51 / 0.77 = 0.66 | noComparaHits: NEWSINFRUG00000132774 | 6 |
| p11 | q3 | +/+ | ENSP00000249440 | HOXD3 | q3 | q3 | 122 | scaffold_130 | 639531 | 639938 | 1 | 1 | 32.2% | 53.2% | 408 | 0.56 / 0.86 = 0.65 | noComparaHits: NEWSINFRUG00000161068 | 4 |
| p12 | q4 | +/+ | ENSP00000328598 | HOXD1 | q4 | q4 | 114 | scaffold_130 | 642031 | 642252 | 1 | 1 | 22.6% | 73% | 222 | 0.79 / 0.81 = 0.98 | noComparaHits: NEWSINFRUG00000131120 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 72 (region 7 for Tnig)
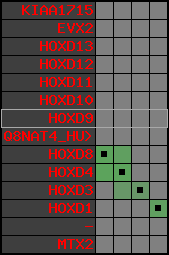 |
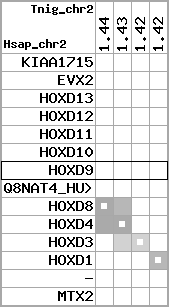 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l 2 -i 1421792:1435644 -f 10e3 some_geneID_to_be_selected
scoreRatio_sum=2.9 log_scoreRatio_sum=-1.35 focal_scoreRatio_sum=0.66
AlnScore=724 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q1 | +/+ | ENSP00000315949 | HOXD8 | q1 | q1 | 112 | 2 | 1435444 | 1435620 | -1 | 1 | 20.3% | 86.4% | 177 | 0.49 / 0.79 = 0.62 | noComparaHits: HOXBb6
; GSTENG00019143001 |
3 |
| p10 | q2 | +/+ | ENSP00000302548 | HOXD4 | q2 | q2 | 123 | 2 | 1432934 | 1433131 | -1 | 1 | 25.9% | 87.9% | 198 | 0.51 / 0.77 = 0.66 | noComparaHits: HOXBb5
; GSTENG00019143001 |
7 |
| p11 | q3 | +/+ | ENSP00000249440 | HOXD3 | q3 | q3 | 105 | 2 | 1424212 | 1424418 | -1 | 1 | 16% | 76.8% | 207 | 0.56 / 0.88 = 0.64 | noComparaHits: HOXBb3
; GSTENG00019144001 |
6 |
| p12 | q4 | +/+ | ENSP00000328598 | HOXD1 | q4 | q4 | 115 | 2 | 1421876 | 1422091 | -1 | 1 | 22% | 75% | 216 | 0.79 / 0.81 = 0.98 | noComparaHits: HOXBb1
; GSTENG00019145001 |
2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 73 (region 8 for Drer)
 |
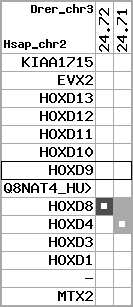 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 3 -i 24709958:24717334 -f 55e2 some_geneID_to_be_selected
scoreRatio_sum=1.65 log_scoreRatio_sum=-0.42 focal_scoreRatio_sum=0.5
AlnScore=710 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q1 | +/+ | ENSP00000315949 | HOXD8 | q1 | q1 | 275 | 3 | 24715898 | 24717334 | -1 | 2 | 95.9% | 52.7% | 681 | 0.49 / 0.49 = 1 | between_species_paralog: hoxb8a | 2 |
| p10 | q2 | +/+ | ENSP00000302548 | HOXD4 | q2 | q2 | 114 | 3 | 24709982 | 24710179 | -1 | 1 | 25.9% | 77.3% | 198 | 0.51 / 0.79 = 0.65 | noComparaHits: hoxb7a | 9 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 74 (region 13 for Gacu)
 |
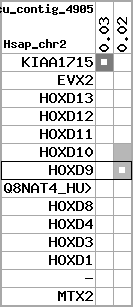 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_4905 -i 15850:35383 -f 15e3 some_geneID_to_be_selected
scoreRatio_sum=2.34 log_scoreRatio_sum=0.18 focal_scoreRatio_sum=0.98
AlnScore=551 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | q1 | +/+ | ENSP00000272748 | KIAA1715 | q1 | q1 | 288.8 | contig_4905 | 32589 | 35383 | 1 | 4 | 43.9% | 51.8% | 825 | 1 / 0.63 = 1.59 | noComparaHits: ENSGACG00000011901 | 1 |
| p2 | --- | gapq | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gapq | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gapq | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gapq | ENSP00000249501 | HOXD10 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q2 | +/+ | ENSP00000249499 | HOXD9 | q2 | q2 | 134 | contig_4905 | 15877 | 16089 | -1 | 1 | 20.8% | 87.3% | 213 | 0.6 / 0.8 = 0.75 | noComparaHits: ENSGACG00000011902 | 6 |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 75 (region 14 for Gacu)
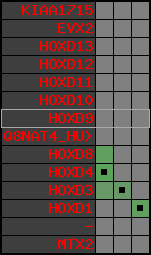 |
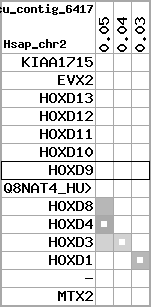 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_6417 -i 33855:46196 -f 93e2 some_geneID_to_be_selected
scoreRatio_sum=2.29 log_scoreRatio_sum=-0.86 focal_scoreRatio_sum=0.46
AlnScore=536 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q1 | +/+ | ENSP00000302548 | HOXD4 | q1 | q1 | 123 | contig_6417 | 45996 | 46193 | -1 | 1 | 25.9% | 87.9% | 198 | 0.51 / 0.77 = 0.66 | noComparaHits: | 8 |
| p11 | q2 | +/+ | ENSP00000249440 | HOXD3 | q2 | q2 | 122 | contig_6417 | 36453 | 36818 | -1 | 1 | 26.2% | 56.6% | 366 | 0.56 / 0.86 = 0.65 | noComparaHits: ENSGACG00000003942 | 4 |
| p12 | q3 | +/+ | ENSP00000328598 | HOXD1 | q3 | q3 | 114 | contig_6417 | 33927 | 34151 | -1 | 1 | 22.9% | 70.7% | 225 | 0.79 / 0.81 = 0.98 | noComparaHits: ENSGACG00000003939 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 76 (region 15 for Gacu)
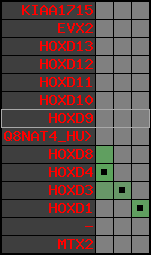 |
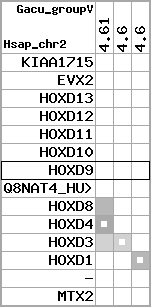 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l groupV -i 4598337:4610678 -f 93e2 some_geneID_to_be_selected
scoreRatio_sum=2.29 log_scoreRatio_sum=-0.86 focal_scoreRatio_sum=0.46
AlnScore=536 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q1 | +/+ | ENSP00000302548 | HOXD4 | q1 | q1 | 123 | groupV | 4610478 | 4610675 | -1 | 1 | 25.9% | 87.9% | 198 | 0.51 / 0.77 = 0.66 | noComparaHits: ENSGACG00000003945 | 8 |
| p11 | q2 | +/+ | ENSP00000249440 | HOXD3 | q2 | q2 | 122 | groupV | 4600935 | 4601300 | -1 | 1 | 26.2% | 56.6% | 366 | 0.56 / 0.86 = 0.65 | noComparaHits: ENSGACG00000003942 | 4 |
| p12 | q3 | +/+ | ENSP00000328598 | HOXD1 | q3 | q3 | 114 | groupV | 4598409 | 4598633 | -1 | 1 | 22.9% | 70.7% | 225 | 0.79 / 0.81 = 0.98 | noComparaHits: ENSGACG00000003939 | 3 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 77 (region 8 for Tnig)
 |
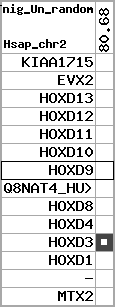 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l Un_random -i 80676474:80677664 -f 89e1 some_geneID_to_be_selected
scoreRatio_sum=1.22 log_scoreRatio_sum=0.2 focal_scoreRatio_sum=0.24
AlnScore=535 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q1 | +/+ | ENSP00000249440 | HOXD3 | q1 | q1 | 491 | Un_random | 80676474 | 80677664 | -1 | 3 | 88.9% | 65.2% | 1092 | 0.56 / 0.46 = 1.22 | ortholog_one2many: GSTENG00003053001 | 2 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 78 (region 16 for Gacu)
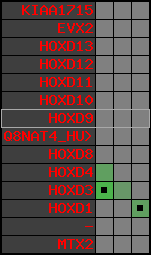 |
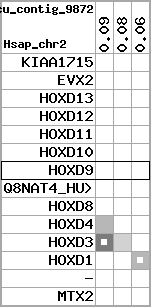 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_9872 -i 61563:92385 -f 23e3 some_geneID_to_be_selected
scoreRatio_sum=1.84 log_scoreRatio_sum=-0.16 focal_scoreRatio_sum=0.34
AlnScore=517 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q1 | +/+ | ENSP00000249440 | HOXD3 | q1 | q1 | 312 | contig_9872 | 88587 | 92385 | -1 | 2 | 79.9% | 49.7% | 1170 | 0.56 / 0.65 = 0.86 | noComparaHits: ENSGACG00000005631 | 3 |
| --- | q2 | gapp | - | - | - | q2 | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | q3 | +/+ | ENSP00000328598 | HOXD1 | q3 | q3 | 118 | contig_9872 | 61596 | 61883 | -1 | 1 | 32.3% | 61.3% | 288 | 0.79 / 0.81 = 0.98 | noComparaHits: ENSGACG00000005635 | 2 |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 79 (region 9 for Tnig)
 |
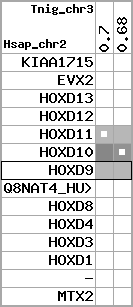 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l 3 -i 679911:697538 -f 13e3 some_geneID_to_be_selected
scoreRatio_sum=1.48 log_scoreRatio_sum=-0.61 focal_scoreRatio_sum=0.63
AlnScore=486 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q1 | +/+ | ENSP00000249504 | HOXD11 | q1 | q1 | 105 | 3 | 695268 | 695510 | -1 | 1 | 23.7% | 61.7% | 243 | 0.53 / 0.81 = 0.65 | between_species_paralog: GSTENG00031863001 | 5 |
| p6 | q2 | +/+ | ENSP00000249501 | HOXD10 | q1 | q2 | 206.8 | 3 | 679911 | 682163 | -1 | 2 | 81.5% | 44.8% | 840 | 0.58 / 0.7 = 0.83 | noComparaHits: GSTENG00031864001 | 5 |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 80 (region 6 for Btau)
 |
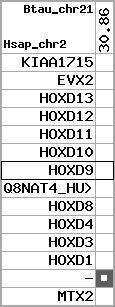 |
 |
retrieve target region: getEnsemblProteins.pl -h Bos_taurus -l 21 -i 30860717:31473122 -f 46e4 some_geneID_to_be_selected
scoreRatio_sum=0.28 log_scoreRatio_sum=-1.26 focal_scoreRatio_sum=0.04
AlnScore=462 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Bos_taurus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gaps | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gaps | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | q1 | +/+ | ENSP00000346209 | - | q1 | q1 | 105 | 21 | 30860717 | 30860908 | -1 | 1 | 61.4% | 78.6% | 192 | 0.15 / 0.53 = 0.28 | noComparaHits: | noData |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 81 (region 9 for Drer)
 |
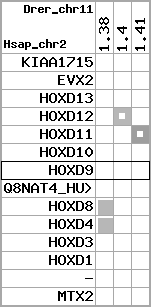 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 11 -i 1379031:1406535 -f 21e3 some_geneID_to_be_selected
scoreRatio_sum=1.55 log_scoreRatio_sum=-0.5 focal_scoreRatio_sum=0.45
AlnScore=446 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| --- | q1 | gaps | - | - | - | q1 | - | - | - | - | - | - | - | - | - | - | - | - |
| p1 | --- | gapq | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gapq | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gapq | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | q2 | +/+ | ENSP00000303578 | HOXD12 | q2 | q2 | 109 | 11 | 1398052 | 1398255 | 1 | 1 | 24.5% | 73.5% | 204 | 0.66 / 0.8 = 0.83 | noComparaHits: hoxc12b | 3 |
| p5 | q3 | +/+ | ENSP00000249504 | HOXD11 | q3 | q3 | 143 | 11 | 1406308 | 1406535 | 1 | 1 | 22.5% | 89.5% | 228 | 0.53 / 0.74 = 0.72 | between_species_paralog: hoxc11b | 5 |
| p6 | --- | gape | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 82 (region 17 for Gacu)
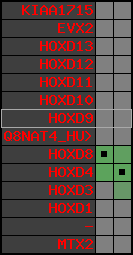 |
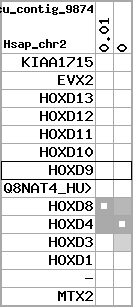 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_9874 -i 2487:8413 -f 44e2 some_geneID_to_be_selected
scoreRatio_sum=1.28 log_scoreRatio_sum=-0.88 focal_scoreRatio_sum=0.37
AlnScore=430 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q1 | +/+ | ENSP00000315949 | HOXD8 | q1 | q1 | 115 | contig_9874 | 8132 | 8398 | -1 | 1 | 32.1% | 64.5% | 267 | 0.49 / 0.78 = 0.63 | noComparaHits: ENSGACG00000005623 | 3 |
| p10 | q2 | +/+ | ENSP00000302548 | HOXD4 | q1 | q2 | 121 | contig_9874 | 2487 | 2714 | -1 | 1 | 29.8% | 76.3% | 228 | 0.51 / 0.78 = 0.65 | noComparaHits: ENSGACG00000005626 | 9 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 83 (region 9 for Trub)
 |
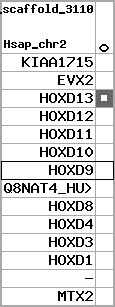 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_3110 -i 2177:3814 -f 12e2 some_geneID_to_be_selected
scoreRatio_sum=1.03 log_scoreRatio_sum=0.03 focal_scoreRatio_sum=0.21
AlnScore=408 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 265 | scaffold_3110 | 2177 | 3805 | 1 | 2 | 89% | 47.8% | 858 | 0.61 / 0.59 = 1.03 | between_species_paralog: NEWSINFRUG00000160125 | 2 |
| p4 | --- | gape | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gape | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gape | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 84 (region 18 for Gacu)
 |
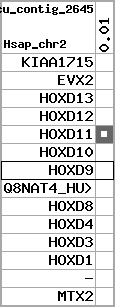 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_2645 -i 14:19677 -f 15e3 some_geneID_to_be_selected
scoreRatio_sum=0.9 log_scoreRatio_sum=-0.1 focal_scoreRatio_sum=0.3
AlnScore=407 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q1 | +/+ | ENSP00000249504 | HOXD11 | q1 | q1 | 226.5 | contig_2645 | 7504 | 9697 | -1 | 2 | 76.9% | 48.9% | 660 | 0.53 / 0.59 = 0.9 | noComparaHits: ENSGACG00000009392 | 4 |
| p6 | --- | gape | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 85 (region 19 for Gacu)
 |
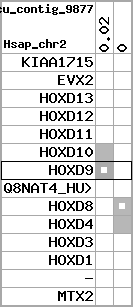 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_9877 -i 4242:26330 -f 17e3 some_geneID_to_be_selected
scoreRatio_sum=1.38 log_scoreRatio_sum=-0.75 focal_scoreRatio_sum=0.97
AlnScore=406 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q1 | +/+ | ENSP00000249499 | HOXD9 | q1 | q1 | 146 | contig_9877 | 17464 | 17697 | -1 | 1 | 22.8% | 85.9% | 234 | 0.6 / 0.78 = 0.77 | noComparaHits: | 4 |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q2 | +/+ | ENSP00000315949 | HOXD8 | q2 | q2 | 106 | contig_9877 | 4242 | 4511 | -1 | 1 | 30% | 60% | 270 | 0.49 / 0.8 = 0.61 | noComparaHits: | 5 |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 86 (region 4 for Ggal)
 |
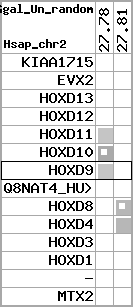 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l Un_random -i 27778788:27807228 -f 21e3 some_geneID_to_be_selected
scoreRatio_sum=1.39 log_scoreRatio_sum=-0.73 focal_scoreRatio_sum=0.59
AlnScore=402 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | q1 | /+ | ENSP00000249501 | HOXD10 | q1 | q1 | 169 | Un_random | 27778788 | 27779063 | -1 | 1 | 27.1% | 87% | 276 | 0.58 / 0.75 = 0.77 | noComparaHits: 430550 | 3 |
| p7 | --- | gapq | ENSP00000249499 | HOXD9 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gapq | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q2 | /+ | ENSP00000315949 | HOXD8 | q2 | q2 | 114 | Un_random | 27806971 | 27807210 | -1 | 1 | 27.6% | 68.8% | 240 | 0.49 / 0.79 = 0.62 | noComparaHits: HOXB7 | 5 |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | q2 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 87 (region 5 for Ggal)
 |
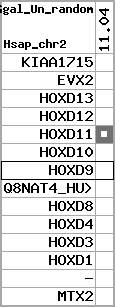 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l Un_random -i 11038122:11040291 -f 16e2 some_geneID_to_be_selected
scoreRatio_sum=0.9 log_scoreRatio_sum=-0.1 focal_scoreRatio_sum=0.3
AlnScore=400 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | q1 | +/+ | ENSP00000249504 | HOXD11 | q1 | q1 | 222.7 | Un_random | 11038122 | 11040291 | -1 | 2 | 99.1% | 41.8% | 849 | 0.53 / 0.59 = 0.9 | between_species_paralog: HOXC11 | 1 |
| p6 | --- | gape | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 88 (region 6 for Ggal)
 |
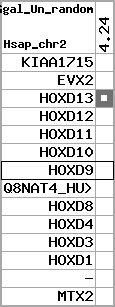 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l Un_random -i 4242278:4245456 -f 24e2 some_geneID_to_be_selected
scoreRatio_sum=1 log_scoreRatio_sum=0 focal_scoreRatio_sum=0.2
AlnScore=385 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 250 | Un_random | 4242278 | 4245456 | 1 | 2 | 60% | 56.8% | 618 | 0.61 / 0.61 = 1 | between_species_paralog: HOXC13 | 1 |
| p4 | --- | gape | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gape | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gape | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 89 (region 10 for Drer)
 |
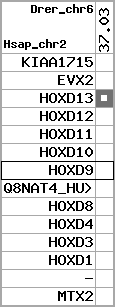 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 6 -i 37029844:37032258 -f 18e2 some_geneID_to_be_selected
scoreRatio_sum=1 log_scoreRatio_sum=0 focal_scoreRatio_sum=0.2
AlnScore=384 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | q1 | +/+ | ENSP00000249505 | HOXD13 | q1 | q1 | 249 | 6 | 37029844 | 37032258 | -1 | 2 | 89.9% | 44.6% | 780 | 0.61 / 0.61 = 1 | between_species_paralog: hoxc13b | 3 |
| p4 | --- | gape | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gape | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gape | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 90 (region 7 for Ggal)
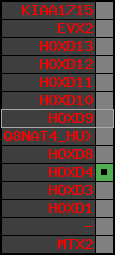 |
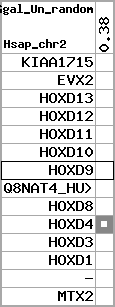 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l Un_random -i 376714:377022 -f 23e1 some_geneID_to_be_selected
scoreRatio_sum=0.74 log_scoreRatio_sum=-0.29 focal_scoreRatio_sum=0.19
AlnScore=309 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q1 | +/+ | ENSP00000302548 | HOXD4 | q1 | q1 | 170 | Un_random | 376735 | 377022 | -1 | 1 | 37.6% | 85.4% | 288 | 0.51 / 0.69 = 0.74 | between_species_paralog: HOXB4 | 3 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 91 (region 8 for Ggal)
 |
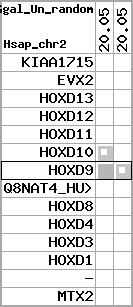 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l Un_random -i 20045093:20048409 -f 25e2 some_geneID_to_be_selected
scoreRatio_sum=1.43 log_scoreRatio_sum=-0.66 focal_scoreRatio_sum=1.07
AlnScore=304 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | q1 | /+ | ENSP00000249501 | HOXD10 | q1 | q1 | 125 | Un_random | 20045126 | 20045365 | -1 | 1 | 23.5% | 70% | 240 | 0.58 / 0.81 = 0.72 | noComparaHits: HXD9_CHICK | 6 |
| p7 | q2 | /+ | ENSP00000249499 | HOXD9 | q1 | q2 | 108 | Un_random | 20047888 | 20048409 | -1 | 1 | 53.2% | 41.8% | 522 | 0.6 / 0.84 = 0.71 | ortholog_one2many: HXD9_CHICK | 7 |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 92 (region 5 for Mmul)
 |
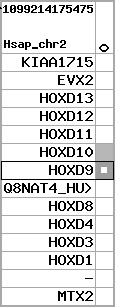 |
 |
retrieve target region: getEnsemblProteins.pl -h Macaca_mulatta -l 1099214175475 -i 300:578 -f 21e1 some_geneID_to_be_selected
scoreRatio_sum=0.8 log_scoreRatio_sum=-0.21 focal_scoreRatio_sum=0.8
AlnScore=248 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Macaca_mulatta
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q1 | +/+ | ENSP00000249499 | HOXD9 | q1 | q1 | 169 | 1099214175475 | 321 | 578 | 1 | 1 | 25.1% | 94.2% | 258 | 0.6 / 0.75 = 0.8 | noComparaHits: | 4 |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 93 (region 10 for Trub)
 |
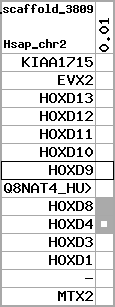 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_3809 -i 5460:5702 -f 18e1 some_geneID_to_be_selected
scoreRatio_sum=0.66 log_scoreRatio_sum=-0.41 focal_scoreRatio_sum=0.17
AlnScore=221 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q1 | +/+ | ENSP00000302548 | HOXD4 | q1 | q1 | 122 | scaffold_3809 | 5472 | 5702 | 1 | 1 | 30.2% | 75.3% | 231 | 0.51 / 0.77 = 0.66 | noComparaHits: NEWSINFRUG00000159821 | 7 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 94 (region 11 for Drer)
 |
 |
 |
retrieve target region: getEnsemblProteins.pl -h Danio_rerio -l 24 -i 25215421:25218746 -f 25e2 some_geneID_to_be_selected
scoreRatio_sum=0.77 log_scoreRatio_sum=-0.25 focal_scoreRatio_sum=0.77
AlnScore=211 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Danio_rerio
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q1 | +/+ | ENSP00000249499 | HOXD9 | q1 | q1 | 144 | 24 | 25218450 | 25218674 | 1 | 1 | 21.9% | 86.7% | 225 | 0.6 / 0.78 = 0.77 | between_species_paralog: hoxb9a | 3 |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 95 (region 11 for Trub)
 |
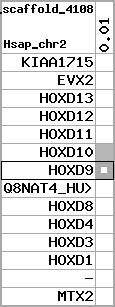 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_4108 -i 5942:6187 -f 18e1 some_geneID_to_be_selected
scoreRatio_sum=0.76 log_scoreRatio_sum=-0.26 focal_scoreRatio_sum=0.76
AlnScore=208 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | q1 | +/+ | ENSP00000249499 | HOXD9 | q1 | q1 | 142 | scaffold_4108 | 5963 | 6187 | 1 | 1 | 21.9% | 86.7% | 225 | 0.6 / 0.79 = 0.76 | noComparaHits: NEWSINFRUG00000159744 | 4 |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 96 (region 20 for Gacu)
 |
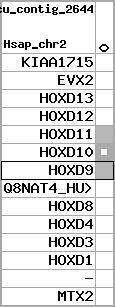 |
 |
retrieve target region: getEnsemblProteins.pl -h Gasterosteus_aculeatus -l contig_2644 -i 2418:2705 -f 22e1 some_geneID_to_be_selected
scoreRatio_sum=0.73 log_scoreRatio_sum=-0.3 focal_scoreRatio_sum=0.37
AlnScore=204 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gasterosteus_aculeatus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | q1 | +/+ | ENSP00000249501 | HOXD10 | q1 | q1 | 142 | contig_2644 | 2418 | 2693 | -1 | 1 | 27.1% | 72.8% | 276 | 0.58 / 0.79 = 0.73 | noComparaHits: | 5 |
| p7 | --- | gape | ENSP00000249499 | HOXD9 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gape | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gape | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 97 (region 10 for Tnig)
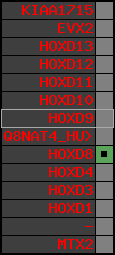 |
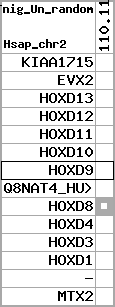 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l Un_random -i 110113334:110113540 -f 16e1 some_geneID_to_be_selected
scoreRatio_sum=0.62 log_scoreRatio_sum=-0.47 focal_scoreRatio_sum=0.21
AlnScore=201 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | q1 | +/+ | ENSP00000315949 | HOXD8 | q1 | q1 | 110 | Un_random | 110113334 | 110113540 | 1 | 1 | 23.8% | 79.7% | 207 | 0.49 / 0.79 = 0.62 | noComparaHits: GSTENG00008821001 | 4 |
| p10 | --- | gape | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 98 (region 4 for Mdom)
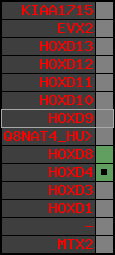 |
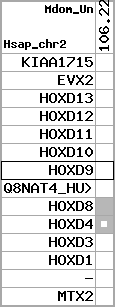 |
 |
retrieve target region: getEnsemblProteins.pl -h Monodelphis_domestica -l Un -i 106219307:106219567 -f 20e1 some_geneID_to_be_selected
scoreRatio_sum=0.64 log_scoreRatio_sum=-0.44 focal_scoreRatio_sum=0.16
AlnScore=200 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Monodelphis_domestica
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | q1 | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q1 | +/+ | ENSP00000302548 | HOXD4 | q1 | q1 | 110 | Un | 106219328 | 106219567 | 1 | 1 | 31.4% | 63.8% | 240 | 0.51 / 0.8 = 0.64 | noComparaHits: HOXC6 | 9 |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 99 (region 9 for Ggal)
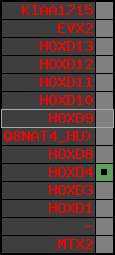 |
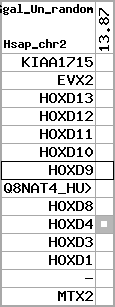 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l Un_random -i 13865410:13865634 -f 17e1 some_geneID_to_be_selected
scoreRatio_sum=0.63 log_scoreRatio_sum=-0.45 focal_scoreRatio_sum=0.16
AlnScore=181 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q1 | +/+ | ENSP00000302548 | HOXD4 | q1 | q1 | 100 | Un_random | 13865410 | 13865601 | 1 | 1 | 25.1% | 71.9% | 192 | 0.51 / 0.81 = 0.63 | noComparaHits: | noData |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 100 (region 12 for Trub)
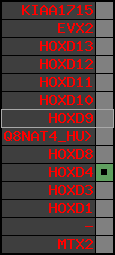 |
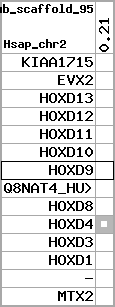 |
 |
retrieve target region: getEnsemblProteins.pl -h Takifugu_rubripes -l scaffold_95 -i 213369:213722 -f 27e1 some_geneID_to_be_selected
scoreRatio_sum=0.63 log_scoreRatio_sum=-0.45 focal_scoreRatio_sum=0.16
AlnScore=181 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Takifugu_rubripes
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | q1 | +/+ | ENSP00000302548 | HOXD4 | q1 | q1 | 100 | scaffold_95 | 213384 | 213611 | 1 | 1 | 29.8% | 63.2% | 228 | 0.51 / 0.81 = 0.63 | noComparaHits: NEWSINFRUG00000149044 | noData |
| p11 | --- | gape | ENSP00000249440 | HOXD3 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 101 (region 7 for Btau)
 |
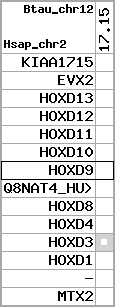 |
 |
retrieve target region: getEnsemblProteins.pl -h Bos_taurus -l 12 -i 17059616:17152307 -f 70e3 some_geneID_to_be_selected
scoreRatio_sum=0.64 log_scoreRatio_sum=-0.44 focal_scoreRatio_sum=0.13
AlnScore=117 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Bos_taurus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q1 | +/+ | ENSP00000249440 | HOXD3 | q1 | q1 | 108 | 12 | 17151894 | 17152307 | 1 | 1 | 31.3% | 47.5% | 414 | 0.56 / 0.88 = 0.64 | noComparaHits: IPF1 | 5 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 102 (region 5 for Mdom)
 |
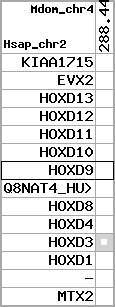 |
 |
retrieve target region: getEnsemblProteins.pl -h Monodelphis_domestica -l 4 -i 288288681:288480685 -f 14e4 some_geneID_to_be_selected
scoreRatio_sum=0.64 log_scoreRatio_sum=-0.44 focal_scoreRatio_sum=0.13
AlnScore=114 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Monodelphis_domestica
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q1 | +/+ | ENSP00000249440 | HOXD3 | q1 | q1 | 105 | 4 | 288439997 | 288440407 | 1 | 1 | 30.8% | 46.4% | 411 | 0.56 / 0.88 = 0.64 | noComparaHits: IPF1 | 6 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 103 (region 10 for Ggal)
 |
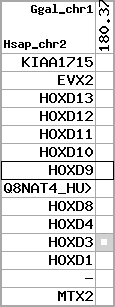 |
 |
retrieve target region: getEnsemblProteins.pl -h Gallus_gallus -l 1 -i 180355088:180368171 -f 98e2 some_geneID_to_be_selected
scoreRatio_sum=0.64 log_scoreRatio_sum=-0.44 focal_scoreRatio_sum=0.13
AlnScore=113 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Gallus_gallus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q1 | +/+ | ENSP00000249440 | HOXD3 | q1 | q1 | 104 | 1 | 180367578 | 180368063 | -1 | 1 | 37% | 43.3% | 486 | 0.56 / 0.88 = 0.64 | noComparaHits: IPF1 | 6 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 104 (region 5 for Cfam)
 |
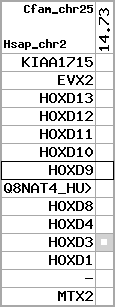 |
 |
retrieve target region: getEnsemblProteins.pl -h Canis_familiaris -l 25 -i 14692875:14842159 -f 11e4 some_geneID_to_be_selected
scoreRatio_sum=0.64 log_scoreRatio_sum=-0.44 focal_scoreRatio_sum=0.13
AlnScore=112 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Canis_familiaris
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q1 | +/+ | ENSP00000249440 | HOXD3 | q1 | q1 | 103 | 25 | 14730016 | 14730396 | -1 | 1 | 26.6% | 48% | 381 | 0.56 / 0.88 = 0.64 | noComparaHits: 493994 | 5 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 105 (region 5 for Mmus)
 |
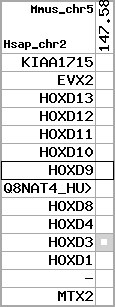 |
 |
retrieve target region: getEnsemblProteins.pl -h Mus_musculus -l 5 -i 147500123:147613649 -f 85e3 some_geneID_to_be_selected
scoreRatio_sum=0.64 log_scoreRatio_sum=-0.44 focal_scoreRatio_sum=0.13
AlnScore=110 (based on relative scores)
AlnType: normal (increasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Mus_musculus
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q1 | +/+ | ENSP00000249440 | HOXD3 | q1 | q1 | 101 | 5 | 147584758 | 147585171 | 1 | 1 | 31.3% | 44.3% | 414 | 0.56 / 0.88 = 0.64 | noComparaHits: Ipf1 | 6 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
region 106 (region 11 for Tnig)
 |
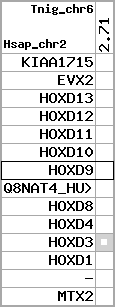 |
 |
retrieve target region: getEnsemblProteins.pl -h Tetraodon_nigroviridis -l 6 -i 2710849:2711238 -f 29e1 some_geneID_to_be_selected
scoreRatio_sum=0.63 log_scoreRatio_sum=-0.45 focal_scoreRatio_sum=0.13
AlnScore=109 (based on relative scores)
AlnType: reversed (decreasing chromosome positions)
QueryProtein(s)=ENSP00000249499 (contained in p7)
TargetGenome=Tetraodon_nigroviridis
| ref-loci | target-loci | aln | protID | geneName | optQloci | target-loci | score | chr | startPos | endPos | ori | hits | queryCov | meanIdentity | hitsLength | intra_inter | compara | globalRank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| p1 | --- | gaps | ENSP00000272748 | KIAA1715 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p2 | --- | gaps | ENSP00000312385 | EVX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p3 | --- | gaps | ENSP00000249505 | HOXD13 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p4 | --- | gaps | ENSP00000303578 | HOXD12 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p5 | --- | gaps | ENSP00000249504 | HOXD11 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p6 | --- | gaps | ENSP00000249501 | HOXD10 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p7 | --- | gaps | ENSP00000249499 | HOXD9 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p8 | --- | gaps | ENSP00000316098 | Q8NAT4_HUMAN | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p9 | --- | gaps | ENSP00000315949 | HOXD8 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p10 | --- | gaps | ENSP00000302548 | HOXD4 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p11 | q1 | +/+ | ENSP00000249440 | HOXD3 | q1 | q1 | 100 | 6 | 2710849 | 2711220 | -1 | 1 | 26.4% | 48.4% | 372 | 0.56 / 0.89 = 0.63 | noComparaHits: GSTENG00023870001 | 10 |
| p12 | --- | gape | ENSP00000328598 | HOXD1 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p13 | --- | gape | ENSP00000346209 | - | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |
| p14 | --- | gape | ENSP00000249442 | MTX2 | noMatch | --- | - | - | - | - | - | - | - | - | - | - | - | - |