
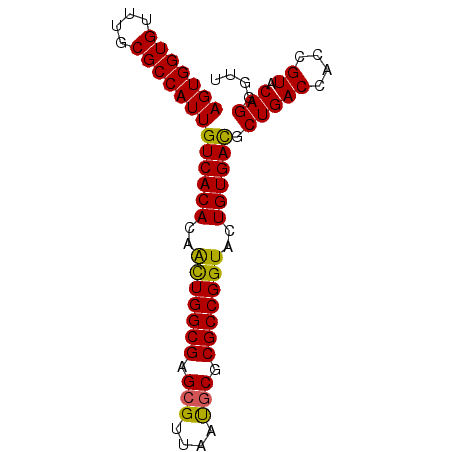
| Sequence ID | Eco_1 |
|---|---|
| Location | 4,390,606 – 4,390,686 |
| Length | 80 |
| Max. P | 0.999982 |

| Location | 4,390,606 – 4,390,686 |
|---|---|
| Length | 80 |
| Sequences | 6 |
| Columns | 80 |
| Reading direction | forward |
| Mean pairwise identity | 96.16 |
| Mean single sequence MFE | -33.37 |
| Consensus MFE | -33.22 |
| Energy contribution | -32.83 |
| Covariance contribution | -0.39 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.60 |
| Structure conservation index | 1.00 |
| SVM decision value | 5.30 |
| SVM RNA-class probability | 0.999982 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>Eco_1 4390606 80 + 1 AGUGGUGUUUGCGCCAUUGUCACACAACUGGCGAGCGUUAAUGCGCGCCGGUACUGUGACGCUGACCACCGUACAGGGUU (((((((....)))))))((((((..(((((((.(((....))).)))))))..)))))).(((((....)).))).... ( -33.90) >Hin_1 1 80 + 1 AGUGGUGUUUGCGCCAUUGUCACACAACUGGCGAGCGUUAACGCGCGCCGGUACUGUGACGCUGACCACCGUACAGGGUU (((((((....)))))))((((((..(((((((.(((....))).)))))))..)))))).(((((....)).))).... ( -36.50) >Lpn_1 1 80 + 1 AGUGGUGUUUGCGCCAUUGUCACACAAUUGGCGAGCGUUAAUACGCGCCGGUACUGUGAUGCUGACCACCGUACAGGAUU (((((((....)))))))((((((..((((((..((((....))))))))))..)))))).(((((....)).))).... ( -27.80) >Sfl_1 1 78 + 1 AGUGGUGUUUGCGCCAUUGUCACACAACUGGCGAGCGUUAAUGCGCGCCGGUACUGUGACGCUGACCACCGUACAGGG-- (((((((....)))))))((((((..(((((((.(((....))).)))))))..)))))).(((((....)).)))..-- ( -33.90) >Sty_1 1 78 + 1 AGUGGUGUUUGCGCCAUUGUCACACAACUGGCGAGCGUUAAUGCGCGCCGGUACUGUGACGCUGACCACCGUACAGGG-- (((((((....)))))))((((((..(((((((.(((....))).)))))))..)))))).(((((....)).)))..-- ( -33.90) >Ype_1 1 80 + 1 AGUGGUGUUUGCGCCAUUGUCACACAGCUGGCGAGCGUUAAUGCGCGCCGGUACUGUGACGCUGACCACCGUACAGGGUU (((((((....)))))))((((((..(((((((.(((....))).)))))))..)))))).(((((....)).))).... ( -34.20) >consensus AGUGGUGUUUGCGCCAUUGUCACACAACUGGCGAGCGUUAAUGCGCGCCGGUACUGUGACGCUGACCACCGUACAGGGUU (((((((....)))))))((((((..(((((((.(((....))).)))))))..)))))).(((((....)).))).... (-33.22 = -32.83 + -0.39)


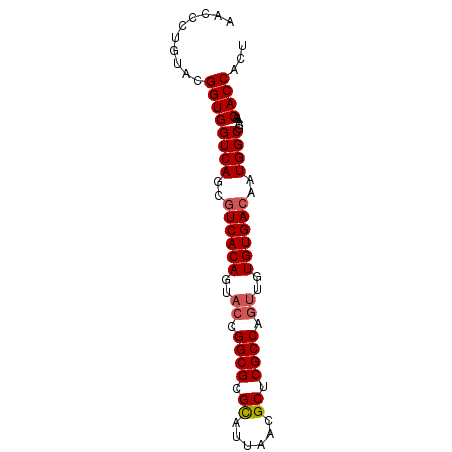
| Location | 4,390,606 – 4,390,686 |
|---|---|
| Length | 80 |
| Sequences | 6 |
| Columns | 80 |
| Reading direction | reverse |
| Mean pairwise identity | 96.16 |
| Mean single sequence MFE | -28.70 |
| Consensus MFE | -27.37 |
| Energy contribution | -27.73 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.63 |
| SVM RNA-class probability | 0.999465 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>Eco_1 4390606 80 - 1 AACCCUGUACGGUGGUCAGCGUCACAGUACCGGCGCGCAUUAACGCUCGCCAGUUGUGUGACAAUGGCGCAAACACCACU ..........((((((((..((((((..((.((((.((......)).)))).))..))))))..)))).....))))... ( -29.80) >Hin_1 1 80 - 1 AACCCUGUACGGUGGUCAGCGUCACAGUACCGGCGCGCGUUAACGCUCGCCAGUUGUGUGACAAUGGCGCAAACACCACU ..........((((((((..((((((..((.((((.(((....))).)))).))..))))))..)))).....))))... ( -32.40) >Lpn_1 1 80 - 1 AAUCCUGUACGGUGGUCAGCAUCACAGUACCGGCGCGUAUUAACGCUCGCCAAUUGUGUGACAAUGGCGCAAACACCACU ...((.((((.(((((....))))).)))).)).((((....)))).(((((.(((.....))))))))........... ( -23.10) >Sfl_1 1 78 - 1 --CCCUGUACGGUGGUCAGCGUCACAGUACCGGCGCGCAUUAACGCUCGCCAGUUGUGUGACAAUGGCGCAAACACCACU --........((((((((..((((((..((.((((.((......)).)))).))..))))))..)))).....))))... ( -29.80) >Sty_1 1 78 - 1 --CCCUGUACGGUGGUCAGCGUCACAGUACCGGCGCGCAUUAACGCUCGCCAGUUGUGUGACAAUGGCGCAAACACCACU --........((((((((..((((((..((.((((.((......)).)))).))..))))))..)))).....))))... ( -29.80) >Ype_1 1 80 - 1 AACCCUGUACGGUGGUCAGCGUCACAGUACCGGCGCGCAUUAACGCUCGCCAGCUGUGUGACAAUGGCGCAAACACCACU ..........((((....((((((((((...((((.((......)).)))).))))))........))))...))))... ( -27.30) >consensus AACCCUGUACGGUGGUCAGCGUCACAGUACCGGCGCGCAUUAACGCUCGCCAGUUGUGUGACAAUGGCGCAAACACCACU ..........((((((((..((((((..((.((((.((......)).)))).))..))))))..)))).....))))... (-27.37 = -27.73 + 0.36)


Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Dec 7 14:13:11 2006