
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 2,644,670 – 2,644,785 |
| Length | 115 |
| Max. P | 0.595244 |

| Location | 2,644,670 – 2,644,785 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.83 |
| Mean single sequence MFE | -37.03 |
| Consensus MFE | -19.10 |
| Energy contribution | -18.47 |
| Covariance contribution | -0.63 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.595244 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2644670 115 + 22224390 UGGUUCAUGCGGUACCGCUGAUGCUUGAUCUCAUUGAAGCAGAUUUGUUGAUGGCUGUGCAGCUGUUCCAGCAGCUCGAUGUACCGCCUGUUGCAGCUCUGUG-----AUCGGUGGGAAU .((((((.(((....)))))).)))...(((((((((.(((((.((((....(((.((((((((((....)))))....))))).)))....)))).))))).-----.))))))))).. ( -45.10) >DroVir_CAF1 82749 109 + 1 UGAUUCAUACGAUAACGUUGAUGUUUGAUCUCGUUGAAGCAUUUCUGUUGAUGCUGCUGCAGCUGUUCCAGCAGCUCCAUGUAGCGUCUGUUGCAUGUCUGCA-----AA--GAUA---- .............((((..(((((((.(......).)))))))..))))((((((((((.((((((....)))))).)).))))))))..(((((....))))-----).--....---- ( -33.50) >DroPse_CAF1 84331 120 + 1 UGGUUCAUGCGAUACCGUUGAUGCUUAAUUUCAUUGAAGCAGAUCUGUUGGCGGCUGUGCAGCUGCUCCAGCAGCUCGAUGUAGCGUCUGUUGCAGUUCUGUCGCAAAAACAGUAGGAGA ..(((..((((((((.((.((((((......((((((.(((.(.(((....))).).))).(((((....))))))))))).))))))....)).))...))))))..)))......... ( -37.80) >DroGri_CAF1 75213 110 + 1 UGAUUCAUGCGAUAUCGCUGAUGUUUGAUCUCGUUGAAGCAUUUCUGCUGGUGUUGAUACAGCUGUUCCAGCAGCUCAAUGUAGCGUCUGUUGCACGUCUACA-----AAA-GGCA---- .......(((((((.(((((((((((.(......).))))))..(((((((.((((...))))....))))))).......)))))..))))))).((((...-----..)-))).---- ( -32.70) >DroMoj_CAF1 96012 113 + 1 UGAUUCAUACGAUAUCGCUGAUGCUUGAUCUCGUUAAAGCAUUUCUGUUGAUGCUGAUGCAGCUGCUCCAGCAGCUCAAUGUUGCGUCUGUUGCACUCCUGCA-----CA--GAGGGAGA .(((((....)).)))((.(((((........((...((((((......))))))...))((((((....)))))).......)))))....)).(((((...-----..--..))))). ( -33.10) >DroAna_CAF1 74091 112 + 1 UGGUUCAUGCGGUAGCGCUGGUGCUUGAUCUCGUUGAAGCACAUCUGUUGGUGACUGUGCAGCUGUUCCAGCAGCUCUAUGUACCGACCGUUGCAGCUCUACA-----AUUGA---AAAA ...((((....((((.((((((((((.(......).))))))....(((((((...(((.((((((....)))))).))).))))))).....)))).)))).-----..)))---)... ( -40.00) >consensus UGAUUCAUGCGAUACCGCUGAUGCUUGAUCUCGUUGAAGCAUAUCUGUUGAUGCCGGUGCAGCUGUUCCAGCAGCUCAAUGUAGCGUCUGUUGCAGCUCUGCA_____AA__GAGGGA_A ....(((.(((....))))))(((((.(......).)))))....(((.(((((......((((((....)))))).......)))))....)))......................... (-19.10 = -18.47 + -0.63)

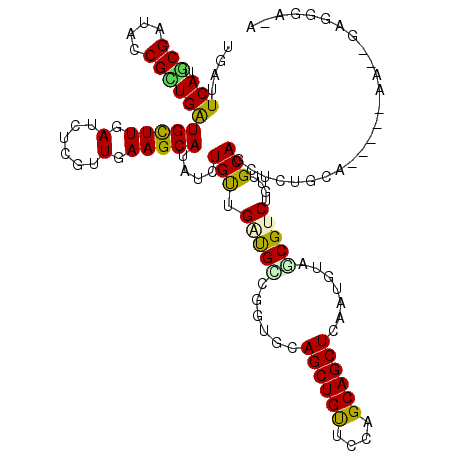
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:57:46 2006