
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 2,515,660 – 2,515,913 |
| Length | 253 |
| Max. P | 0.999935 |

| Location | 2,515,660 – 2,515,780 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.17 |
| Mean single sequence MFE | -47.50 |
| Consensus MFE | -46.48 |
| Energy contribution | -45.84 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.34 |
| Structure conservation index | 0.98 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.911007 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2515660 120 - 22224390 CGUGGUGGUGGCAACUGCAGUUGCUGCUGCGAUGCACUGGAAACAAUUACACCGCUAUCCGCCGAGCCCGUCGAUGUUUGCGAUGAGAAGCGCUCGGUUAGCGGUAUUUUAAUCGGCUGG .((.(..(..(((((....)))))..)..).))((..((....)).....(((((((...(((((((((((((.......)))))....).))))))))))))))..........))... ( -46.40) >DroSec_CAF1 13877 120 - 1 CGUGGUGGUGGCAACUGCAGUUGCUGCUGCGAUGCACUGGAAACAAUUACACCGCUGUCCGCCGAGCCCGUCGAUGUUUGCGAUGAGAAGCGCUCGGUCAGCGGUAUUUUAAUCGGCUGG .((.(..(..(((((....)))))..)..).))((..((....)).....(((((((...(((((((((((((.......)))))....).))))))))))))))..........))... ( -47.70) >DroSim_CAF1 13866 120 - 1 CGUGGUGGUGGCAACUGCAGUUGCUGCUGCGAUGCACUGGAAACAAUUACACCGCUGUCCGCCGAGCCCGUCGAUGUUUGCGAUGAGAAGCGCUCGGUCAGCGGUAUUUUAAUCGGCUGG .((.(..(..(((((....)))))..)..).))((..((....)).....(((((((...(((((((((((((.......)))))....).))))))))))))))..........))... ( -47.70) >DroEre_CAF1 14617 120 - 1 CGUGGUGGCGGCAAUUGCAGUUGCUGCUGCGAUGCACUGGAAACAAUUACACCGCUGUCCGCCGAACCCGUCGAUGUUUGCGAUGAGAAGCGCUCGGUCAGCGGUAUUUUAAUCGGCUGG .((.(..((((((((....))))))))..).))((..((....)).....(((((((.(((.((...((((((.......))))).)...))..))).)))))))..........))... ( -45.20) >DroYak_CAF1 15850 120 - 1 CGCGGUGGCGGCAACUGCAGUUGCUGCUGCGAUGCACUGGAAACAAUUACGCCGCUGUCCGCCGAGCCCGUUGAUGUUUGCGAUGAGAAGCGCUCGGUCAGCGGUAUUUUAAUCGGCUGG .((((((((((((..((((((....)))))).)))..((....))....)))))))))(((((((..((((((((....(((........)))...))))))))........))))).)) ( -50.50) >consensus CGUGGUGGUGGCAACUGCAGUUGCUGCUGCGAUGCACUGGAAACAAUUACACCGCUGUCCGCCGAGCCCGUCGAUGUUUGCGAUGAGAAGCGCUCGGUCAGCGGUAUUUUAAUCGGCUGG .((.(..((((((((....))))))))..).))((..((....)).....(((((((...(((((((((((((.......)))))....).))))))))))))))..........))... (-46.48 = -45.84 + -0.64)

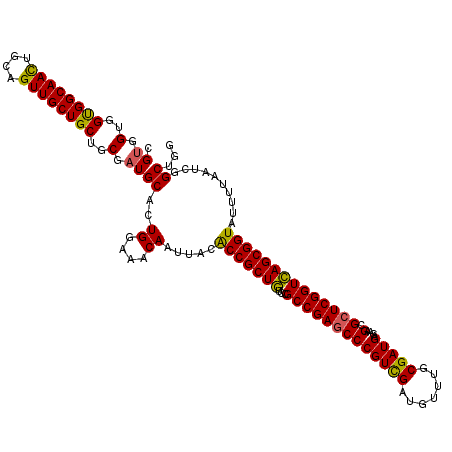
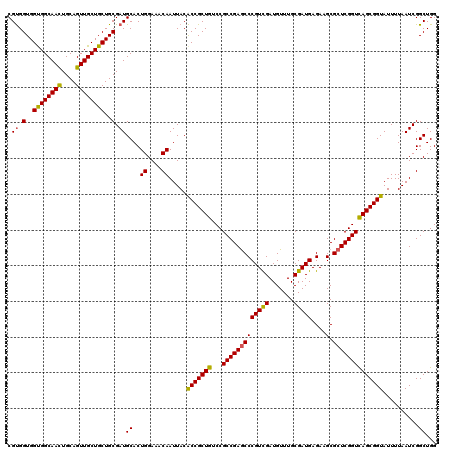
| Location | 2,515,700 – 2,515,820 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -47.92 |
| Consensus MFE | -43.38 |
| Energy contribution | -43.66 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.98 |
| SVM RNA-class probability | 0.984569 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2515700 120 + 22224390 CAAACAUCGACGGGCUCGGCGGAUAGCGGUGUAAUUGUUUCCAGUGCAUCGCAGCAGCAACUGCAGUUGCCACCACCACGCAGUAGCAGUGGAUCGCUGAGUCUGCCACAAGCGCCACCU ........(.((((((((((((...((((((((.(((....)))))))))))....(((((....)))))..)).((((((....)).))))...)))))))))).)............. ( -44.90) >DroSec_CAF1 13917 120 + 1 CAAACAUCGACGGGCUCGGCGGACAGCGGUGUAAUUGUUUCCAGUGCAUCGCAGCAGCAACUGCAGUUGCCACCACCACGCAGUAGCAGUGGCUCGCUGAGCCUGCCGCAGGCGCCACCU .......((.(((((((((((....((((((((.(((....)))))))))))...(((.(((((..((((.........))))..))))).)))))))))))))).)).(((.....))) ( -50.00) >DroSim_CAF1 13906 120 + 1 CAAACAUCGACGGGCUCGGCGGACAGCGGUGUAAUUGUUUCCAGUGCAUCGCAGCAGCAACUGCAGUUGCCACCACCACGCAGUAGCAGUGGCUCGCUGAGCCUGCCGCAGGCGCCACCU .......((.(((((((((((....((((((((.(((....)))))))))))...(((.(((((..((((.........))))..))))).)))))))))))))).)).(((.....))) ( -50.00) >DroEre_CAF1 14657 120 + 1 CAAACAUCGACGGGUUCGGCGGACAGCGGUGUAAUUGUUUCCAGUGCAUCGCAGCAGCAACUGCAAUUGCCGCCACCACGCAGUAGCAGUGGCUCACUGAGCCUGCCCCAGGCGCCACCU ...........(((((((((((.((((((((((.(((....))))))))))).((((...))))...))))))).((((((....)).))))......))))))(((...)))....... ( -45.30) >DroYak_CAF1 15890 120 + 1 CAAACAUCAACGGGCUCGGCGGACAGCGGCGUAAUUGUUUCCAGUGCAUCGCAGCAGCAACUGCAGUUGCCGCCACCGCGCAGUAGCAGUGGCUCGCUGAGCCUGCCACAGGCGCCACCU ...........(((((((((((.((((.(((..((((....))))....))).((((...)))).))))))(((((.((......)).)))))..)))))))))(((...)))....... ( -49.40) >consensus CAAACAUCGACGGGCUCGGCGGACAGCGGUGUAAUUGUUUCCAGUGCAUCGCAGCAGCAACUGCAGUUGCCACCACCACGCAGUAGCAGUGGCUCGCUGAGCCUGCCACAGGCGCCACCU ...........((((((((((....((((((((.(((....)))))))))))...(((.(((((..((((.........))))..))))).)))))))))))))((.....))....... (-43.38 = -43.66 + 0.28)

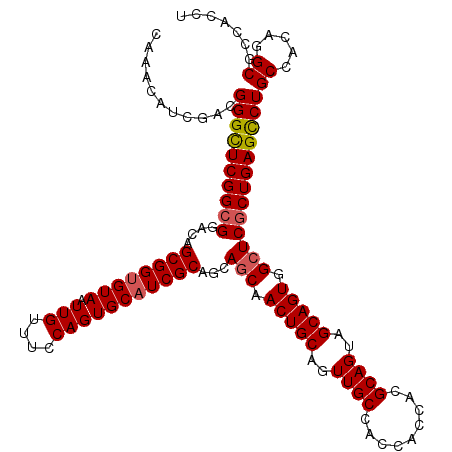
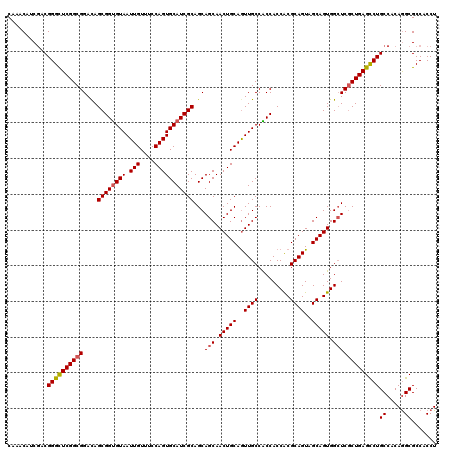
| Location | 2,515,740 – 2,515,860 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -54.82 |
| Consensus MFE | -48.88 |
| Energy contribution | -49.20 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.85 |
| SVM RNA-class probability | 0.997377 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2515740 120 + 22224390 CCAGUGCAUCGCAGCAGCAACUGCAGUUGCCACCACCACGCAGUAGCAGUGGAUCGCUGAGUCUGCCACAAGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAACAGUUGC ....(((......)))(((((((..(.(((.....((((((....)).))))...((((..((((((.....((((....))))((.....))))))))..)))).))).)..))))))) ( -49.70) >DroSec_CAF1 13957 120 + 1 CCAGUGCAUCGCAGCAGCAACUGCAGUUGCCACCACCACGCAGUAGCAGUGGCUCGCUGAGCCUGCCGCAGGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGC ....(((......)))((((((((.(((((..(......)..)))))..((((.(((((...(((((((.(.((((....)))).).....)))))))...)))).).)))))))))))) ( -54.60) >DroSim_CAF1 13946 120 + 1 CCAGUGCAUCGCAGCAGCAACUGCAGUUGCCACCACCACGCAGUAGCAGUGGCUCGCUGAGCCUGCCGCAGGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGC ....(((......)))((((((((.(((((..(......)..)))))..((((.(((((...(((((((.(.((((....)))).).....)))))))...)))).).)))))))))))) ( -54.60) >DroEre_CAF1 14697 120 + 1 CCAGUGCAUCGCAGCAGCAACUGCAAUUGCCGCCACCACGCAGUAGCAGUGGCUCACUGAGCCUGCCCCAGGCGCCACCUGGCGGCAAGCGGCGGCAGAAGCAGCAGCGCCAGCAAUUGC .....(((..((.((.((..((((..((((((((......(((((((....))).)))).((.(((((((((.....))))).)))).))))))))))..))))..))))..))...))) ( -55.60) >DroYak_CAF1 15930 120 + 1 CCAGUGCAUCGCAGCAGCAACUGCAGUUGCCGCCACCGCGCAGUAGCAGUGGCUCGCUGAGCCUGCCACAGGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGCUGC ....(((.((.((((.(((((....))))).(((((.((......)).)))))..)))).(((.(((((.(.((((....)))).)..)))))))).)).)))(((((.......))))) ( -59.60) >consensus CCAGUGCAUCGCAGCAGCAACUGCAGUUGCCACCACCACGCAGUAGCAGUGGCUCGCUGAGCCUGCCACAGGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGC ....(((.((.(((((((.(((((..((((.........))))..))))).))).)))).(((.(((((..(((((....))).))..)))))))).)).)))(((((.......))))) (-48.88 = -49.20 + 0.32)

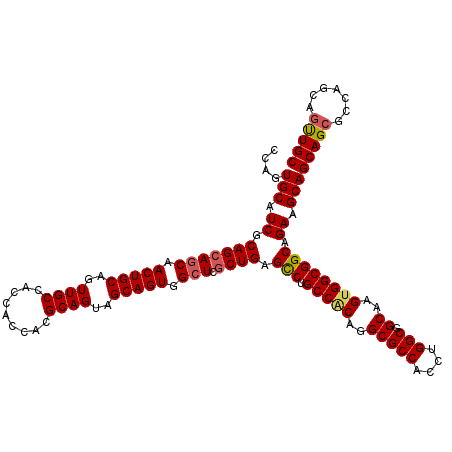
| Location | 2,515,740 – 2,515,860 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -64.44 |
| Consensus MFE | -59.10 |
| Energy contribution | -58.78 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.10 |
| Structure conservation index | 0.92 |
| SVM decision value | 4.66 |
| SVM RNA-class probability | 0.999935 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2515740 120 - 22224390 GCAACUGUUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCUUGUGGCAGACUCAGCGAUCCACUGCUACUGCGUGGUGGUGGCAACUGCAGUUGCUGCUGCGAUGCACUGG ((((((((.(.((((((((((.(...(((((((((....)))))))))...).)))))...)))))..((((..((((...))))..))))...).))))))))(((......))).... ( -55.60) >DroSec_CAF1 13957 120 - 1 GCAACUGCUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCCUGCGGCAGGCUCAGCGAGCCACUGCUACUGCGUGGUGGUGGCAACUGCAGUUGCUGCUGCGAUGCACUGG ((((((((.(.(((((..(((((((((((((((((....)))))))))...)))).)))).))))).(((((..((((...))))..)))))..).))))))))(((......))).... ( -64.80) >DroSim_CAF1 13946 120 - 1 GCAACUGCUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCCUGCGGCAGGCUCAGCGAGCCACUGCUACUGCGUGGUGGUGGCAACUGCAGUUGCUGCUGCGAUGCACUGG ((((((((.(.(((((..(((((((((((((((((....)))))))))...)))).)))).))))).(((((..((((...))))..)))))..).))))))))(((......))).... ( -64.80) >DroEre_CAF1 14697 120 - 1 GCAAUUGCUGGCGCUGCUGCUUCUGCCGCCGCUUGCCGCCAGGUGGCGCCUGGGGCAGGCUCAGUGAGCCACUGCUACUGCGUGGUGGCGGCAAUUGCAGUUGCUGCUGCGAUGCACUGG (((.((((.(((((.(((((...((((((((((((((.(((((.....)))))))))))).......(((((.((....))))))))))))))...))))).)).))))))))))..... ( -70.00) >DroYak_CAF1 15930 120 - 1 GCAGCUGCUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCCUGUGGCAGGCUCAGCGAGCCACUGCUACUGCGCGGUGGCGGCAACUGCAGUUGCUGCUGCGAUGCACUGG (((..(((.(((((.(((((...((((((((((.(((....))).((((..(((((((((((...)))))..)))))).))))))))))))))...))))).)).)))))).)))..... ( -67.00) >consensus GCAACUGCUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCCUGCGGCAGGCUCAGCGAGCCACUGCUACUGCGUGGUGGUGGCAACUGCAGUUGCUGCUGCGAUGCACUGG (((..(((.(((((.(((((...((((((((((...(((..(((((((((...))).(((((...)))))...))))))))).))))))))))...))))).)).)))))).)))..... (-59.10 = -58.78 + -0.32)


| Location | 2,515,780 – 2,515,897 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.26 |
| Mean single sequence MFE | -54.42 |
| Consensus MFE | -50.48 |
| Energy contribution | -50.64 |
| Covariance contribution | 0.16 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.22 |
| SVM RNA-class probability | 0.999839 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2515780 117 + 22224390 CAGUAGCAGUGGAUCGCUGAGUCUGCCACAAGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAACAGUUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA---UGGUGUCAC ..((..(((((...))))).(((((((((..(((((....))).))..)))))(((.((.((((((((.......)))))))))).))).)))).))((((((.....---.)))))).. ( -52.30) >DroSec_CAF1 13997 117 + 1 CAGUAGCAGUGGCUCGCUGAGCCUGCCGCAGGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA---UGGUGUCAC ..(.(((....))))((((..((((((((.(.((((....)))).)..)))))(((.((.((((((((.......)))))))))).)))))).))))((((((.....---.)))))).. ( -55.90) >DroSim_CAF1 13986 117 + 1 CAGUAGCAGUGGCUCGCUGAGCCUGCCGCAGGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA---UGGUGUCAC ..(.(((....))))((((..((((((((.(.((((....)))).)..)))))(((.((.((((((((.......)))))))))).)))))).))))((((((.....---.)))))).. ( -55.90) >DroEre_CAF1 14737 120 + 1 CAGUAGCAGUGGCUCACUGAGCCUGCCCCAGGCGCCACCUGGCGGCAAGCGGCGGCAGAAGCAGCAGCGCCAGCAAUUGCUGCUCAGCCAGGACAGCGGCAUUGAAAAUAAUGGUGUCAC (((((((....))).)))).((((((((((((.....))))).))))...)))(((.((.((((((((....))...)))))))).)))..((((.((....(....)...)).)))).. ( -49.30) >DroYak_CAF1 15970 117 + 1 CAGUAGCAGUGGCUCGCUGAGCCUGCCACAGGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGCUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA---UGGUGUCAC ..(.(((....))))((((..((((((((.(.((((....)))).)..)))))(((.((.((((((((.......)))))))))).)))))).))))((((((.....---.)))))).. ( -58.70) >consensus CAGUAGCAGUGGCUCGCUGAGCCUGCCACAGGCGCCACCUGGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA___UGGUGUCAC ..(.(((....))))((((..((((((((..(((((....))).))..)))))(((.((.((((((((.......)))))))))).)))))).))))(((((((.......))))))).. (-50.48 = -50.64 + 0.16)

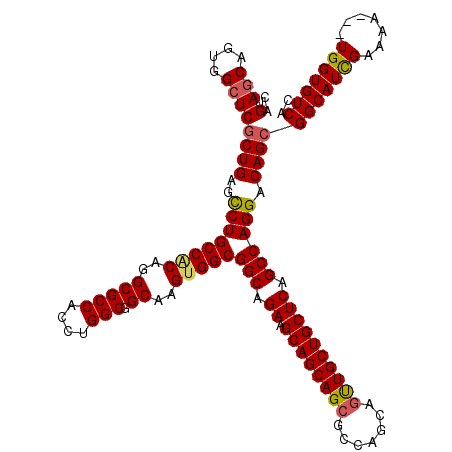
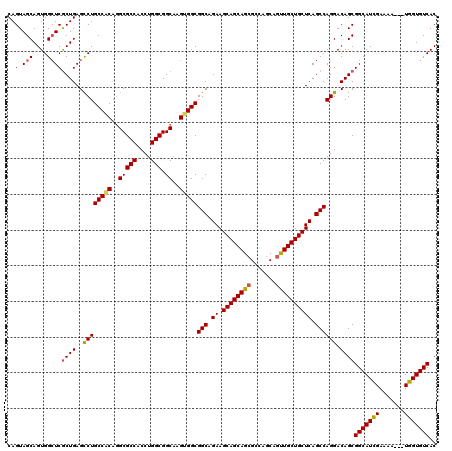
| Location | 2,515,780 – 2,515,897 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.26 |
| Mean single sequence MFE | -50.32 |
| Consensus MFE | -46.98 |
| Energy contribution | -47.10 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.34 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.899039 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2515780 117 - 22224390 GUGACACCA---UUUUCGAUGCCGCUGUCCUGGCUGAGCAGCAACUGUUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCUUGUGGCAGACUCAGCGAUCCACUGCUACUG (((....((---((...)))).(((((..(((((.((((((((.(.......).))))))))..)))(((((..((.(((....)))))..)))))))...)))))...)))........ ( -44.60) >DroSec_CAF1 13997 117 - 1 GUGACACCA---UUUUCGAUGCCGCUGUCCUGGCUGAGCAGCAACUGCUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCCUGCGGCAGGCUCAGCGAGCCACUGCUACUG (((.((...---.......((((((......(((.((((((((...((....))))))))))..)))((((((((....))))))))....))))))(((((...)))))..)).))).. ( -51.30) >DroSim_CAF1 13986 117 - 1 GUGACACCA---UUUUCGAUGCCGCUGUCCUGGCUGAGCAGCAACUGCUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCCUGCGGCAGGCUCAGCGAGCCACUGCUACUG (((.((...---.......((((((......(((.((((((((...((....))))))))))..)))((((((((....))))))))....))))))(((((...)))))..)).))).. ( -51.30) >DroEre_CAF1 14737 120 - 1 GUGACACCAUUAUUUUCAAUGCCGCUGUCCUGGCUGAGCAGCAAUUGCUGGCGCUGCUGCUUCUGCCGCCGCUUGCCGCCAGGUGGCGCCUGGGGCAGGCUCAGUGAGCCACUGCUACUG ....................(((((((....(((.((((((((...((....))))))))))..)))...(((((((.(((((.....)))))))))))).))))).))........... ( -49.50) >DroYak_CAF1 15970 117 - 1 GUGACACCA---UUUUCGAUGCCGCUGUCCUGGCUGAGCAGCAGCUGCUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCCUGUGGCAGGCUCAGCGAGCCACUGCUACUG .........---........(((((((.((((((.((((((((((.......))))))))))..)))(((((..((.(((....)))))..))))))))..))))).))........... ( -54.90) >consensus GUGACACCA___UUUUCGAUGCCGCUGUCCUGGCUGAGCAGCAACUGCUGGCGCUGCUGCUUCUGCCGCCACUUGCCGCCAGGUGGCGCCUGCGGCAGGCUCAGCGAGCCACUGCUACUG (((.((.............((((((......(((.((((((((...((....))))))))))..)))((((((((....))))))))....))))))(((((...)))))..)).))).. (-46.98 = -47.10 + 0.12)

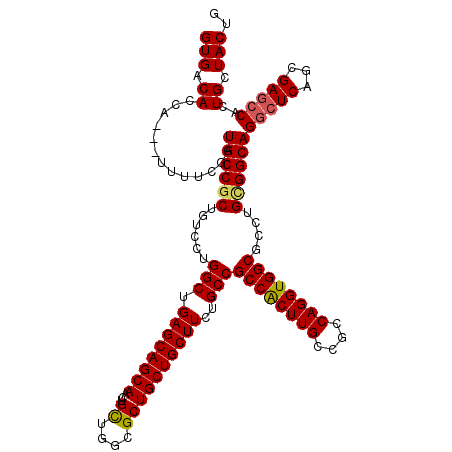
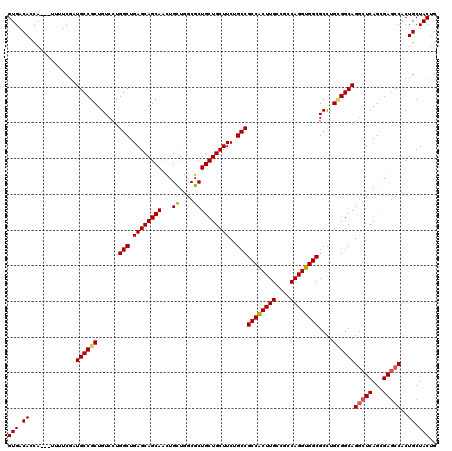
| Location | 2,515,820 – 2,515,913 |
|---|---|
| Length | 93 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.42 |
| Mean single sequence MFE | -38.74 |
| Consensus MFE | -33.02 |
| Energy contribution | -32.74 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.76 |
| SVM RNA-class probability | 0.975835 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2515820 93 + 22224390 GGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAACAGUUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA---UGGUGUCACCA---------------------CUCGUCCAUCGAAA--- .(.(((.(((((.(((.((.((((((((.......)))))))))).)))........((((((.....---.)))))).)))---------------------)).)))).......--- ( -37.60) >DroSec_CAF1 14037 93 + 1 GGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA---UGGUGUCACCA---------------------CCCGUCCAUCGAAA--- (((((...((((.(((.((.((((((((.......)))))))))).)))........((((((.....---.)))))).)))---------------------))))))........--- ( -38.70) >DroSim_CAF1 14026 93 + 1 GGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA---UGGUGUCACCA---------------------CCCGUCCAUCGAAA--- (((((...((((.(((.((.((((((((.......)))))))))).)))........((((((.....---.)))))).)))---------------------))))))........--- ( -38.70) >DroEre_CAF1 14777 102 + 1 GGCGGCAAGCGGCGGCAGAAGCAGCAGCGCCAGCAAUUGCUGCUCAGCCAGGACAGCGGCAUUGAAAAUAAUGGUGUCACCACCA------------------CCCGUCAAUCGAAAGCC ((((((((((.(((((.((.((((((((....))...)))))))).)))......)).)).))).......(((((....)))))------------------.)))))...(....).. ( -35.10) >DroYak_CAF1 16010 117 + 1 GGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGCUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA---UGGUGUCACCACCAACACCACCACCACCUCCUCCCGUCAAUCAAAAGCC (((((...((((.((..(((((((((((.......)))))))))..(((........))).)).....---((((((........)))))))).))))......)))))........... ( -43.60) >consensus GGCGGCAAGUGGCGGCAGAAGCAGCAGCGCCAGCAGUUGCUGCUCAGCCAGGACAGCGGCAUCGAAAA___UGGUGUCACCA_____________________CCCGUCCAUCGAAA___ (((((........(((.((.((((((((.......)))))))))).)))........(((((((.......)))))))..........................)))))........... (-33.02 = -32.74 + -0.28)

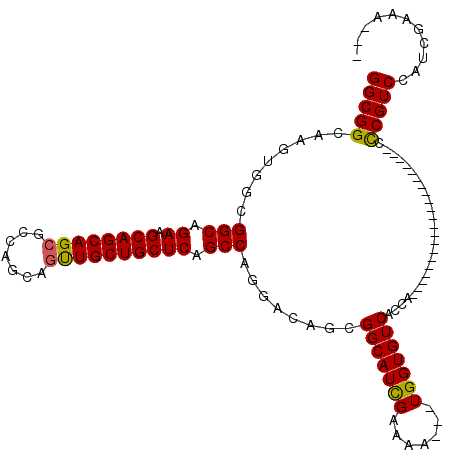
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:56:48 2006