
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 21,812,089 – 21,812,184 |
| Length | 95 |
| Max. P | 0.842162 |

| Location | 21,812,089 – 21,812,184 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 84.57 |
| Mean single sequence MFE | -25.09 |
| Consensus MFE | -19.41 |
| Energy contribution | -19.60 |
| Covariance contribution | 0.19 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.842162 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
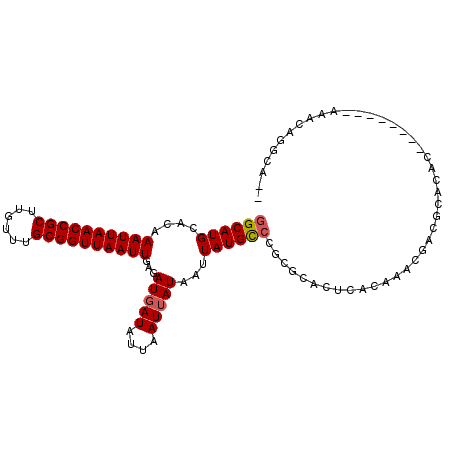
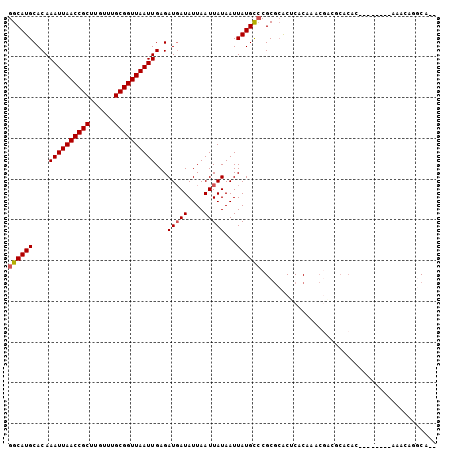
>X_DroMel_CAF1 21812089 95 + 22224390 GGCAUGCACAAAUUAACCGCUUGUUUGCGGUUAAUUGAGAUGAUAUUAAUUAUAAUUAUGCCCGCGCACUCACAAACGACUCACAC--------ACAGAGGCA-- ((((((....((((((((((......))))))))))...(((((....)))))...))))))...((.(((...............--------...))))).-- ( -24.57) >DroPse_CAF1 36094 98 + 1 GGCAUGCACAAAUUAACCGCUUGUUUGCGGUUAAUUGAGAUGAUAUUAAUUAUAAUUAUGUGCGC--ACACACAGAG-ACACACACACAA----AACAAAUCACA .((..(((((((((((((((......))))))))))...(((((....))))).....)))))))--..........-............----........... ( -21.60) >DroSec_CAF1 28040 95 + 1 GGCAUGCACAAAUUAACCGCUUGUUUGCGGUUAAUUGAGAUUAUAUUAAUUAUAAUUAUGCCCGCGCACUCACAAACGACUCGCAC--------ACACAGGCA-- (((((.....((((((((((......))))))))))..(((((((.....)))))))))))).(((...((......))..)))..--------.........-- ( -26.30) >DroEre_CAF1 27866 95 + 1 GGCAUGCACAAAUUAACCGCUUGUUUGCGGUUAAUUGAGAUGAUAUUAAUUAUAAUUAUGCCCGCGCACUCACAAACGGCGCACAC--------AAACAGGCG-- ((((((....((((((((((......))))))))))...(((((....)))))...)))))).((((.(........)))))....--------.........-- ( -27.30) >DroYak_CAF1 26137 103 + 1 GGCAUGCACAAAUUAACCGCUUGUUUGCGGUUAAUUGAGAUGAUAUUAAUUAUAAUUAUGCCCGCGCACUCACAAACGACGCACACACAGGCACAAACUGGCA-- ((((((....((((((((((......))))))))))...(((((....)))))...)))))).((((..((......)).)).....(((.......))))).-- ( -26.70) >DroAna_CAF1 28979 97 + 1 GGCAUGCACAAAUUAACCGCUUGUUAGCGGUUAAUUGAGAUGAUAUUAAUUAUAAUUAUGCCAGCGCGCGCACAAACAAUCAAAUCAU------ACAAACACA-- ((((((....(((((((((((....)))))))))))...(((((....)))))...)))))).((....)).................------.........-- ( -24.10) >consensus GGCAUGCACAAAUUAACCGCUUGUUUGCGGUUAAUUGAGAUGAUAUUAAUUAUAAUUAUGCCCGCGCACUCACAAACGACGCACAC________AAACAGGCA__ ((((((....((((((((((......))))))))))...(((((....)))))...))))))........................................... (-19.41 = -19.60 + 0.19)

| Location | 21,812,089 – 21,812,184 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 84.57 |
| Mean single sequence MFE | -29.15 |
| Consensus MFE | -19.05 |
| Energy contribution | -19.38 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.65 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.512520 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
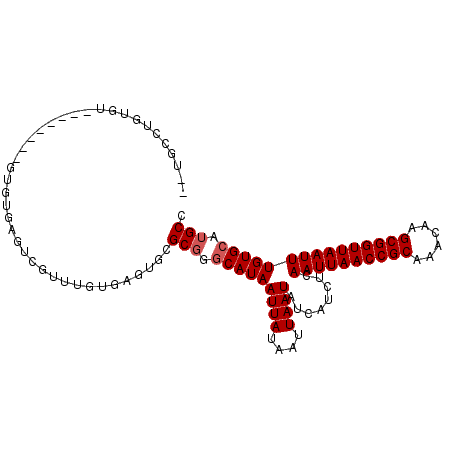
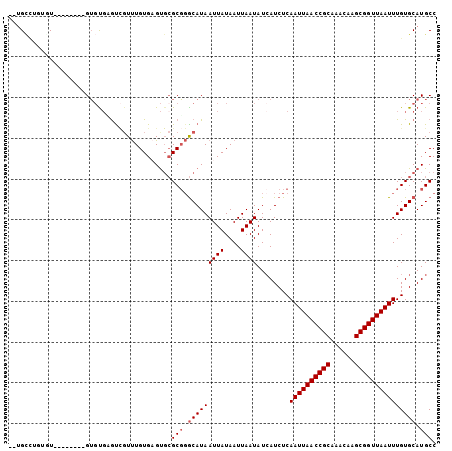
>X_DroMel_CAF1 21812089 95 - 22224390 --UGCCUCUGU--------GUGUGAGUCGUUUGUGAGUGCGCGGGCAUAAUUAUAAUUAAUAUCAUCUCAAUUAACCGCAAACAAGCGGUUAAUUUGUGCAUGCC --.......((--------(((((((..(((((((....)))))))...((((....)))).....)))((((((((((......))))))))))...)))))). ( -25.30) >DroPse_CAF1 36094 98 - 1 UGUGAUUUGUU----UUGUGUGUGUGU-CUCUGUGUGU--GCGCACAUAAUUAUAAUUAAUAUCAUCUCAAUUAACCGCAAACAAGCGGUUAAUUUGUGCAUGCC ..(((((....----(((((((((..(-........).--.)))))))))....)))))....((((.(((((((((((......)))))))).))).).))).. ( -27.40) >DroSec_CAF1 28040 95 - 1 --UGCCUGUGU--------GUGCGAGUCGUUUGUGAGUGCGCGGGCAUAAUUAUAAUUAAUAUAAUCUCAAUUAACCGCAAACAAGCGGUUAAUUUGUGCAUGCC --.....((((--------(..((((..(((((((....)))))))...((((((.....)))))))))((((((((((......)))))))))).)..))))). ( -30.30) >DroEre_CAF1 27866 95 - 1 --CGCCUGUUU--------GUGUGCGCCGUUUGUGAGUGCGCGGGCAUAAUUAUAAUUAAUAUCAUCUCAAUUAACCGCAAACAAGCGGUUAAUUUGUGCAUGCC --.........--------((((((((.(((((((....))))))).......................((((((((((......)))))))))).)))))))). ( -28.90) >DroYak_CAF1 26137 103 - 1 --UGCCAGUUUGUGCCUGUGUGUGCGUCGUUUGUGAGUGCGCGGGCAUAAUUAUAAUUAAUAUCAUCUCAAUUAACCGCAAACAAGCGGUUAAUUUGUGCAUGCC --(((((..(((((((((((..((((.....)))..)..)))))))))))...................((((((((((......)))))))))))).))).... ( -34.40) >DroAna_CAF1 28979 97 - 1 --UGUGUUUGU------AUGAUUUGAUUGUUUGUGCGCGCGCUGGCAUAAUUAUAAUUAAUAUCAUCUCAAUUAACCGCUAACAAGCGGUUAAUUUGUGCAUGCC --...((.(((------(((((((((((((((((((........))))))..)))))))).))).....(((((((((((....))))))))))).))))).)). ( -28.60) >consensus __UGCCUGUGU________GUGUGAGUCGUUUGUGAGUGCGCGGGCAUAAUUAUAAUUAAUAUCAUCUCAAUUAACCGCAAACAAGCGGUUAAUUUGUGCAUGCC ........................................(((.(((((((((....))))........((((((((((......))))))))))))))).))). (-19.05 = -19.38 + 0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:09:18 2006