
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 21,735,642 – 21,735,762 |
| Length | 120 |
| Max. P | 0.603931 |

| Location | 21,735,642 – 21,735,762 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.67 |
| Mean single sequence MFE | -46.88 |
| Consensus MFE | -30.27 |
| Energy contribution | -30.72 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.46 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.603931 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 21735642 120 + 22224390 GUCACUCUGUACUCUGUUUACGAGGAUGCGGGGUCCGCAUAUCUUGUGAUGGAGCUGCUUAAGGGUGGCGAGCUUCUCGAUCGGAUACUUGCCGUGGGCCAGAUGUGCGAGAGUGAGGCC ((((((((((((((((((.((((((((((((...)))))).))))))))))))).)))...))))))))..(((((.(..(((.(((((.(((...))).)).))).)))..).))))). ( -51.30) >DroVir_CAF1 31166 120 + 1 GUGACGCUCUAUUCUGUGUACGAGGACACCAGCUCCGCGUAUCUGGUCAUGGAGCUGCUUAAGGGCGGCGAACUGCUCGAUCGCAUUCUGGCCGUUGGCCAAAUGUGCGAGAGCGAGGCU ((..(((((......((((......))))((((((((.(........).)))))))).....((((((....))))))..((((((..(((((...)))))...)))))))))))..)). ( -46.90) >DroGri_CAF1 31959 120 + 1 GUUACCCUCUACUCGGUGUACGAGGACACAAACUCCGCAUAUCUGGUCAUGGAGCUGCUCAAGGGCGGCGAACUCCUCGAUCGCAUCCUCGCCGUCGGCCAGAUGUGCGAGAGCGAGGCA .....((((..((((.....))))........((((((((((((((((..(((((((((....))))))....)))..(((.((......)).)))))))))))))))).))).)))).. ( -54.00) >DroWil_CAF1 25259 120 + 1 GUUACCCUCUAUUCCGUCUAUGAGGAUGCGAGUUCAGCGUAUCUGGUUAUGGAACUACUCAAGGGUGGUGAACUACUCGAUCGCAUUCUGGCCGUUGGCCAAAUGUGUGAAAGUGAAGCC ...(((((........((((((((((((((.......)))))))..)))))))........)))))(((....((((....((((((.(((((...)))))))))))....))))..))) ( -36.99) >DroMoj_CAF1 99556 120 + 1 GUGACAUUAUAUUCAGUGUACGAAGAUGCCAGCUCUGCGUAUUUAGUGAUGGAGCUGCUCAAGGGCGGCGAACUGCUCGAUCGCAUCCUGGCAGUUCAACAAAUGUGCGAGAGUGAGGCU (((.((((......)))))))......(((.(((((.(((((....((.....((((((....))))))((((((((.(........).))))))))..))...))))))))))..))). ( -42.90) >DroAna_CAF1 21415 120 + 1 GUGACGCUGUACUCCGUGUACGAGGACGCGGGCUCUGCCUACCUGGUGAUGGAGCUGCUGAAGGGCGGAGAGCUGCUGGAUCGCAUCCUGGCCGUGGGCCAGAUGUGCGAGAGCGAGGCG ((..(((((((((((((.(((.(((....((((...)))).))).))))))))).))).....(((((....)))))...((((((.((((((...))))))..)))))).))))..)). ( -49.20) >consensus GUGACCCUCUACUCCGUGUACGAGGACGCGAGCUCCGCGUAUCUGGUGAUGGAGCUGCUCAAGGGCGGCGAACUGCUCGAUCGCAUCCUGGCCGUUGGCCAAAUGUGCGAGAGCGAGGCC ...........(((.((((......))))..(((((((((((((((((((((.((((((....))))))....(((......)))......)))).))))))))))))).)))))))... (-30.27 = -30.72 + 0.45)

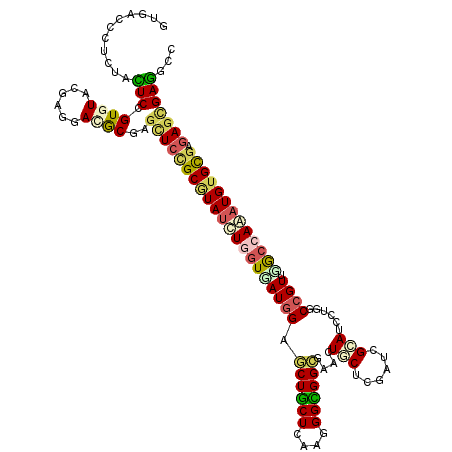
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:09:07 2006