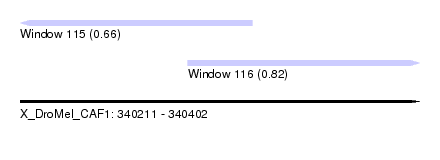
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 340,211 – 340,402 |
| Length | 191 |
| Max. P | 0.820807 |

| Location | 340,211 – 340,322 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 82.52 |
| Mean single sequence MFE | -41.87 |
| Consensus MFE | -29.48 |
| Energy contribution | -29.21 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.47 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.662213 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
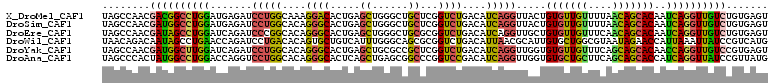
>X_DroMel_CAF1 340211 111 - 22224390 GCCAGGAUCUCAUCCAGGCCGUCGUUGGCUAUCGGGCGGAAAGAAAGCAGACACAGGACAUAAGUGAAACGGAGCUGGAUGGUUUGGCGGACUUGGGUCUGAAACUGGCUA (((((..((..(((((((((((((..(((((((.(((.....(....)...(((.........))).......))).))))))))))))).)))))))..))..))))).. ( -43.10) >DroSim_CAF1 4877 111 - 1 GCCAGGAUCUCAUCCAGGCCAUCGUUGGCUAUCGGGCGGAAAGAAAGCAGACACAGGACAUAAGUGAAACGGAGCUGGAUGGUUUGGCGGACUUGGGUCUGAAACUGGCUA (((((........((((((((((...((((.(((.((.........))...(((.........)))...))))))).))))))))))(((((....)))))...))))).. ( -38.00) >DroEre_CAF1 7486 111 - 1 GCCGGGAUCUGAUCCAGGCUAUCGUUGGCUAUCGCGCGGAAAGAAAACAGACGCAGGACAUCAGUGAGACGGAGCUGGAUGGUUUGGCGGACUUGGGUCUGAAGCUGGCUA (((((..((.((((((((...((((..((((((..(((.............))).......((((........))))))))).)..)))).)))))))).))..))))).. ( -40.02) >DroWil_CAF1 1039 111 - 1 GUCAGGAUCUGGUUCAGGCUAUUGUCUGUUAUCGGUGCGAACGCAAACAAGCUCACGACCUUAGUGAAACGGAAUUGGAUGGCCUGGGGGAGUUGGGCUUAAAACUGGCCA (((((...(((((.(((((....)))))..)))))(((....)))...((((((((..((((((.(...((........)).)))))))..).)))))))....))))).. ( -35.30) >DroYak_CAF1 4933 111 - 1 GCCAGGAUCUGAUCCAAGCCAUCGUUGGCUAUCGGUCGGAAAGAAAGCAGACACAGGACAUCAGUGAAACGGAGCUGGAUGGCUUGGCCGACUUGGGUCUGAAGCUGGCCA (((((..(((((((..(((((....)))))...))))))).......(((((.((((...(((((........))))).((((...)))).)))).)))))...))))).. ( -42.30) >DroAna_CAF1 2076 111 - 1 GCCAGGACCUGGUCCAGGCCAUAGUGGGCUAUCGGGCGGAGCGCAAGCAGGCCCAGGAUCUGACCGAAACGGAGCUGGAUGGUCUGGCGGAUCUGGGCCUGAAGCUGGCCA ((((((.((((((...((((......)))))))))))...((....))((((((((.((((..(((...))).((..(.....)..))))))))))))))....))))).. ( -52.50) >consensus GCCAGGAUCUGAUCCAGGCCAUCGUUGGCUAUCGGGCGGAAAGAAAGCAGACACAGGACAUAAGUGAAACGGAGCUGGAUGGUUUGGCGGACUUGGGUCUGAAACUGGCCA (((((..((.((((((((((.(((..(((((((.(((.........((...............))........))).)))))))))).)).)))))))).))..))))).. (-29.48 = -29.21 + -0.27)


| Location | 340,291 – 340,402 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 80.12 |
| Mean single sequence MFE | -41.15 |
| Consensus MFE | -27.12 |
| Energy contribution | -26.52 |
| Covariance contribution | -0.60 |
| Combinations/Pair | 1.57 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.820807 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 340291 111 + 22224390 UAGCCAACGACGGCCUGGAUGAGAUCCUGGCAAAGGACACUGAGCUGGGCUGCUCGGUCUGACAUCAGGUUACUGUGUUGUUUUAACAGCACAAUCAGGUUGUCUGUGAGU ..((..(((((((((((((((...((((.....)))).(((((((......)))))))....)))).......((((((((....))))))))..))))))))).))..)) ( -42.70) >DroSim_CAF1 4957 111 + 1 UAGCCAACGAUGGCCUGGAUGAGAUCCUGGCACAGGGCACUGAGCUGGGCUGCUCGGUCUGACAUCAGGUUACUGUGUUGUUUUAACAGCACAAUCAGGUUGUCUGUGAGU ..((..((((..((((((..(((..((..((.(((....))).))..))...)))(..(((....)))..)..((((((((....)))))))).))))))..)).))..)) ( -42.90) >DroEre_CAF1 7566 111 + 1 UAGCCAACGAUAGCCUGGAUCAGAUCCCGGCACAGGGCACUGAGCUGGGCUGCGCGGUCUGACAUCAGGUUGCUGUGUUGUUUCAACAGCACAAUCAGGUUGUCUGUGAGU ..((..((((((((((((..(((..((((((.(((....))).))))))))).(((..(((....)))..)))((((((((....)))))))).)))))))))).))..)) ( -48.70) >DroWil_CAF1 1119 111 + 1 UAACAGACAAUAGCCUGAACCAGAUCCUGACACAGUGCUGUCAUUUGGGCAGCGCGGUCUGACAUUAACGCAUUGUGCUGGCGUAAUAGAACCAUUAAAUUAUCCGUCAUG .....(((....(((.....((((((........((((((((.....))))))))))))))........((.....)).))).((((......))))........)))... ( -26.40) >DroYak_CAF1 5013 111 + 1 UAGCCAACGAUGGCUUGGAUCAGAUCCUGGCACAGGGCACUGAGCUGCGCCGCUCGGUCUGACAUCAGGUUGGUGUGUUGUUUCAGCAGCACAACCAGGUUGUCCGUGAGU .(((((....))))).(((((((((((((...)))))..((((((......)))))))))))...(((.((((((((((((....))))))).))))).))))))...... ( -42.90) >DroAna_CAF1 2156 111 + 1 UAGCCCACUAUGGCCUGGACCAGGUCCUGGCACAGGGCACUCAGCUGAGCGGCCCGGUCCGACAUCAGGUUGGUGUGCUGCUUCAGCAGCACCAUCAGGUUAUCCGUUAUG (((((.......(((((((((..((....))...((((.(((....)))..)))))))))(....)))))(((((((((((....)))))).))))))))))......... ( -43.30) >consensus UAGCCAACGAUGGCCUGGAUCAGAUCCUGGCACAGGGCACUGAGCUGGGCUGCUCGGUCUGACAUCAGGUUACUGUGUUGUUUCAACAGCACAAUCAGGUUGUCCGUGAGU ........((((((((((.......(((((....((((.....((......))...))))....))))).....(((((((....)))))))..))))))))))....... (-27.12 = -26.52 + -0.60)
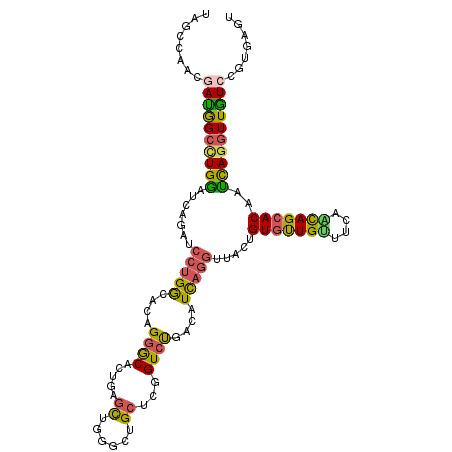
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:33:46 2006