
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 21,163,944 – 21,164,075 |
| Length | 131 |
| Max. P | 0.856497 |

| Location | 21,163,944 – 21,164,056 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 80.89 |
| Mean single sequence MFE | -48.33 |
| Consensus MFE | -34.88 |
| Energy contribution | -35.83 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.81 |
| SVM RNA-class probability | 0.856497 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
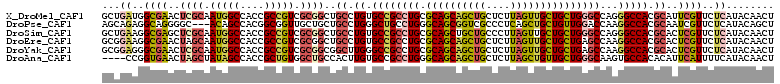
>X_DroMel_CAF1 21163944 112 - 22224390 GCUGAUGGCGAACUCGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCAGCUGCUCUUAGUUGCUGCUGGGCCAGGGCCACGCAUUCGUUCUCAUACAACU ..(((..(((((..((...(((((...(((((.(((((.......)))))..))((((((((((....))))))))))..)))...)))))))..)))))..)))....... ( -49.70) >DroPse_CAF1 1677 109 - 1 AGCAGAGGCAGGGGC---ACAGCCACGGCGGUUGCUGCUGCCUGGGCUGCCUGGGCAGCGGUCGCCCUCAGCUGCUGUUGGACCAAGGCCACGCAAUCGUUCUCAUACAGCU (((.(((((...(((---...)))...))(((.(((((((((..((...))..))))))))).)))))).)))(((((.((((....((...))....))))....))))). ( -49.60) >DroSim_CAF1 1450 112 - 1 GCUGAAGGCGAGCUCGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCUGCUGCCCUUAGUUGCUGCUGGGCCAGGGCCACGCACUCGUUCUCAUACAACU ..(((..(((((..((...(((((...(((((.(((((.......)))))..))(((((.((((....)))).)))))..)))...)))))))..)))))..)))....... ( -48.70) >DroEre_CAF1 1411 112 - 1 GCGGAAGGCGAACUAGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCAGCUGCUCUUAGUUGCUGCUGAGCCAAGGCCACGCACUCGUUCUCAUACAACU ...((..((((.........(((..((((....))))..))).((((.((((((.(((((((.((.....)).))))))).))..))))...))))))))..))........ ( -46.40) >DroYak_CAF1 1428 112 - 1 GCGGAGGGCGAACUCGCAAUGGCCACCGCCGUCGCGGCGGCUUGGGCCGCCUGCGCAGCAGCUGCUCUUAGUUGCUGCUGAGCCAAGGCCACGCACUCGUUCUCAUACAACU ...((((((((....((.(((((....))))).))((((((....))))))(((((((((((((....))))))))))((.((....)))).))).))))))))........ ( -54.30) >DroAna_CAF1 2597 108 - 1 ----CCGGUGAACUAGCUAUAGCCACCGCUGUGGCUGCCACUUGUGCCGCCUGGGCAGCAGCUGCUCUUAGCUGUUGCUGGGCAAGUGCCACACAUUCAUUUUCAUACAACU ----...(((((.((((((((((....)))))))))).....((((.(((((.(((((((((((....))))))))))).))...))).))))........)))))...... ( -41.30) >consensus GCGGAAGGCGAACUCGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCAGCUGCUCUUAGUUGCUGCUGGGCCAAGGCCACGCACUCGUUCUCAUACAACU ...((..((((..((((.(((((....))))).))))..((.((.((((((((.((((((((((....)))))))))))))))...))))).))..))))..))........ (-34.88 = -35.83 + 0.95)
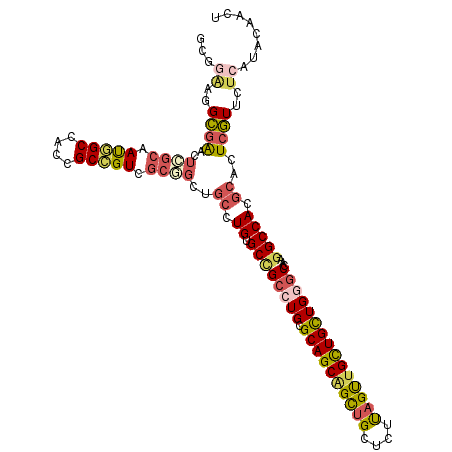
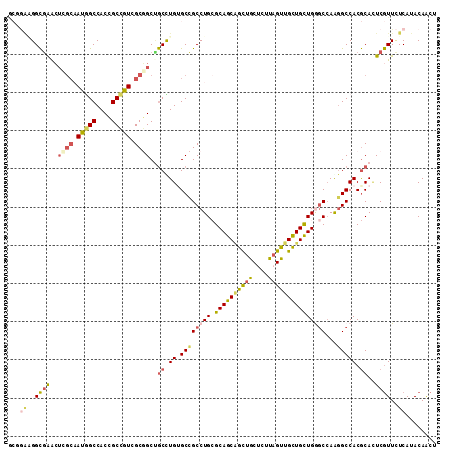
| Location | 21,163,976 – 21,164,075 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 81.27 |
| Mean single sequence MFE | -42.82 |
| Consensus MFE | -29.31 |
| Energy contribution | -28.45 |
| Covariance contribution | -0.86 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.653254 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 21163976 99 + 22224390 CAGCAGCAACUAAGAGCAGCUGCUGCGCAGGCGGCACAGGCAGCCGCGACGGCGGUGGCCAUUGCGAGUUCGCCAUCAGCUAAGGCUAUGGCCGAACCA (((((((..(.....)..)))))))((((((((((.......)))))...(((....))).))))).(((((((((.(((....))))))).))))).. ( -45.10) >DroPse_CAF1 1709 93 + 1 CAACAGCAGCUGAGGGCGACCGCUGCCCAGGCAGCCCAGGCAGCAGCAACCGCCGUGGCUGU---GCCCCUGCCUCUGCUUAUGCCUGUGGCUU---CC .....(((((...((....)))))))...((.((((((((((..((((...((.(.(((...---))).).))...))))..)))))).)))).---)) ( -40.00) >DroSec_CAF1 2161 99 + 1 CAGCAGCAACUAAGGGCAGCAGCUGCGCAGGCGGCACAGGCAGCCGCGACGGCGGUGGCCAUUGCGAGUUCGCCUUCAGCUAAGACUAUGGCCGAACCA .....((((.....(((.((.(((((((.(((.((....)).)))))).)))).)).))).))))(.(((((((...((......))..)).)))))). ( -36.60) >DroSim_CAF1 1482 99 + 1 CAGCAGCAACUAAGGGCAGCAGCUGCGCAGGCGGCACAGGCAGCCGCGACGGCGGUGGCCAUUGCGAGCUCGCCUUCAGCUAAGGCUAUGGCCGAACCA ..(((((..((......))..)))))...((((((..((((((((((((.(((....))).))))).))).))))..(((....)))...))))..)). ( -41.70) >DroEre_CAF1 1443 96 + 1 CAGCAGCAACUAAGAGCAGCUGCUGCGCAGGCGGCACAGGCAGCCGCGACGGCGGUGGCCAUUGCUAGUUCGCCUUCCGCCAAGACGGCGGCCGAA--- ..((((((.((......)).))))))((..((((((..(((.(((((....))))).)))..)))).))..))...(((((.....))))).....--- ( -43.20) >DroYak_CAF1 1460 99 + 1 CAGCAGCAACUAAGAGCAGCUGCUGCGCAGGCGGCCCAAGCCGCCGCGACGGCGGUGGCCAUUGCGAGUUCGCCCUCCGCUAAGGCGACGGCCGAACCA (((((((..(.....)..)))))))(((.((((((....))))))))).((((.((.(((...(((((......)).)))...))).)).))))..... ( -50.30) >consensus CAGCAGCAACUAAGAGCAGCUGCUGCGCAGGCGGCACAGGCAGCCGCGACGGCGGUGGCCAUUGCGAGUUCGCCUUCAGCUAAGGCUAUGGCCGAACCA .............((((....(((((....)))))...(((.(((((....))))).))).......))))(((...((......))..)))....... (-29.31 = -28.45 + -0.86)

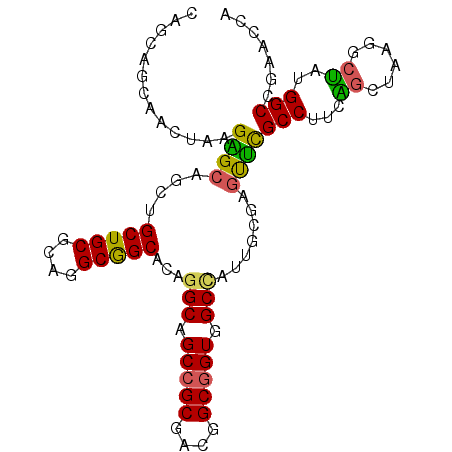

| Location | 21,163,976 – 21,164,075 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 81.27 |
| Mean single sequence MFE | -46.93 |
| Consensus MFE | -34.61 |
| Energy contribution | -36.28 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.841209 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 21163976 99 - 22224390 UGGUUCGGCCAUAGCCUUAGCUGAUGGCGAACUCGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCAGCUGCUCUUAGUUGCUGCUG .((((((.(((((((....))).)))))))))).((((((((....))))).(((((.......))))).)))((((((((((....)))))))))).. ( -47.40) >DroPse_CAF1 1709 93 - 1 GG---AAGCCACAGGCAUAAGCAGAGGCAGGGGC---ACAGCCACGGCGGUUGCUGCUGCCUGGGCUGCCUGGGCAGCGGUCGCCCUCAGCUGCUGUUG ..---..(((.((((((...((..((((((.(((---(..((....))...)))).))))))..)))))))))))((((((.((.....)).)))))). ( -48.10) >DroSec_CAF1 2161 99 - 1 UGGUUCGGCCAUAGUCUUAGCUGAAGGCGAACUCGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCUGCUGCCCUUAGUUGCUGCUG .((((((.(..((((....))))..).)))))).((((((((....))))).(((((.......))))).)))(((((.((((....)))).))))).. ( -39.50) >DroSim_CAF1 1482 99 - 1 UGGUUCGGCCAUAGCCUUAGCUGAAGGCGAGCUCGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCUGCUGCCCUUAGUUGCUGCUG .((..(((((.((((....)))).((((.(((.(((.(((((....))))).))))))))))).))))))...(((((.((((....)))).))))).. ( -43.60) >DroEre_CAF1 1443 96 - 1 ---UUCGGCCGCCGUCUUGGCGGAAGGCGAACUAGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCAGCUGCUCUUAGUUGCUGCUG ---...(((.(((((..((((((..(((.(........).))).))))))..))))).)))...((.....))((((((((((....)))))))))).. ( -47.30) >DroYak_CAF1 1460 99 - 1 UGGUUCGGCCGUCGCCUUAGCGGAGGGCGAACUCGCAAUGGCCACCGCCGUCGCGGCGGCUUGGGCCGCCUGCGCAGCAGCUGCUCUUAGUUGCUGCUG .((((((.((..(((....)))..)).)))))).((.(((((....))))).))((((((....))))))...((((((((((....)))))))))).. ( -55.70) >consensus UGGUUCGGCCAUAGCCUUAGCUGAAGGCGAACUCGCAAUGGCCACCGCCGUCGCGGCUGCCUGUGCCGCCUGCGCAGCAGCUGCCCUUAGUUGCUGCUG .(((((.(((.((((....))))..)))))))).((((((((....))))).(((((.......))))).)))((((((((((....)))))))))).. (-34.61 = -36.28 + 1.67)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:06:59 2006