
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 21,136,126 – 21,136,322 |
| Length | 196 |
| Max. P | 0.967637 |

| Location | 21,136,126 – 21,136,242 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 80.46 |
| Mean single sequence MFE | -53.57 |
| Consensus MFE | -29.05 |
| Energy contribution | -28.30 |
| Covariance contribution | -0.75 |
| Combinations/Pair | 1.28 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.667131 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 21136126 116 + 22224390 CUCCGGCUGAG-GGACGUAUGCAAGGCGCACAACAUGUGGCUGCACGCCAGUGGCCACGGACUGGCCGCCUUGGUGUGCGCCCAAAAUCAGGGCCAUGUGGAGGAGGUACUCCACUC ....(((((((-((.(((((((((((((..((...((((((..(......)..))))))...))..)))))).))))))))))....))..))))..((((((......)))))).. ( -54.70) >DroVir_CAF1 73939 116 + 1 CAGAGGCUGCG-CGAUGUCUGUAAGGCGCACAACAUGUGGCUGCAUGCCAGUGGCCAUGGCCUGGCCGCACUCGUUUGCGCCCAGAGUCCGGGCAAUGUGCACGAUGUGCUGCACUC ..(((((.(((-((((.((((...((((((.(((...((((.....))))(((((((.....)))))))....))))))))))))))))((.((.....)).))..)))).)).))) ( -50.20) >DroGri_CAF1 83709 116 + 1 CAGCUUUUGCG-GGAUGUCUGCAAGGCGCAAAAUAUGUGGCUACAUGCCAGCGGCCAUGGAUUGGCCGCACUCGUUUGUGCCCAGCGGCCGGGCAAUGUUCAUGAUGUCCUGCAUUC .......((((-((((((((((..((((((((.....((((.....))))(((((((.....))))))).....))))))))..)))...((((...))))..)))))))))))... ( -52.70) >DroMoj_CAF1 17884 116 + 1 CAGCGCCUGCG-CGACGUCUGCAAGGCCCACAACAUGUGGCUGCACGCCAGCGGCCACGGCCUGGCCGCACUCGUCUGCGCCCAGAGUCCGGGCAGCGUGCACGACGUGCUCCACUC ..(((....))-).(((((((((..((((.......((((((((......))))))))(((((((.((((......)))).)))).))).))))....)))).)))))......... ( -56.20) >DroAna_CAF1 742 116 + 1 AUCCGGCUGCG-GGAUGUGUGCAAGGCGAGCAACAUGUGGCUCCAUGCCAGUGGCCACGGCCUGGCCGCCCUGGUCUGUGCCCAGUAUCCGGGCCAUGUGGAGGAUGUCCUGCACUC ....((((.((-((.((.(..((.(((.((.......((((.....))))(((((((.....))))))).)).)))))..).))...))))))))..(((.(((....))).))).. ( -48.50) >DroPer_CAF1 65687 117 + 1 CAGCGACUACGUGGACGUGUGCAAGCCGCACAACAUGUGGCUUCAUGCCAGUGGCCAUGGCCUGGCCGCUCUCGUGUGUGCCCAGAGCCAGGGCCAUGUCGACGAUGUCCUGCACUC (((.(((..(((.((((((((.((((((((.....)))))))))))))..((((((.((((((((.(((.(....).))).)))).)))).))))))))).)))..))))))..... ( -59.10) >consensus CAGCGGCUGCG_GGACGUCUGCAAGGCGCACAACAUGUGGCUGCAUGCCAGUGGCCACGGCCUGGCCGCACUCGUCUGCGCCCAGAGUCCGGGCAAUGUGCACGAUGUCCUGCACUC ..............((((..(((.(((..........((((.....))))(((((((.....)))))))....))))))((((.......)))).)))).................. (-29.05 = -28.30 + -0.75)

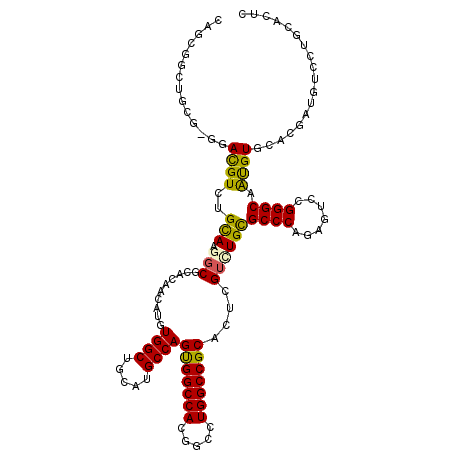
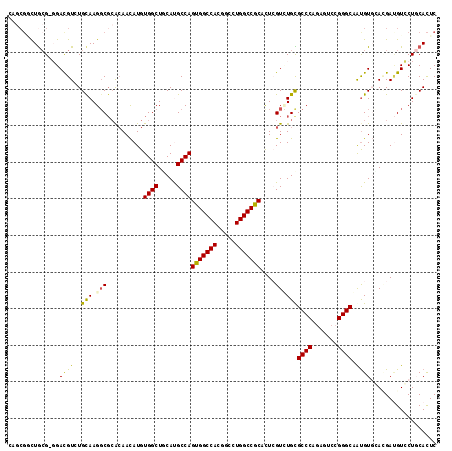
| Location | 21,136,165 – 21,136,282 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.11 |
| Mean single sequence MFE | -58.82 |
| Consensus MFE | -38.54 |
| Energy contribution | -38.97 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.30 |
| Mean z-score | -3.12 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967637 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 21136165 117 + 22224390 CUGCACGCCAGUGGCCACGGACUGGCCGCCUUGGUGUGCGCCCAAAAUCAGGGCCAUGUGGAGGAGG---UACUCCACUCGAUGGCCCUGAAUUUGGGCAGCUGGCUGGGUGUGCCCAGU ..(((((((((((((((.....)))))))..))))))))(((((((.((((((((((((((((....---..))))))...)))))))))).))))))).....((((((....)))))) ( -78.50) >DroVir_CAF1 73978 117 + 1 CUGCAUGCCAGUGGCCAUGGCCUGGCCGCACUCGUUUGCGCCCAGAGUCCGGGCAAUGUGCACGAUG---UGCUGCACUCCAUGGCGCUAAAUCUGGGCAGCUGGCUGGGCGUGCCCAGU ..(((((((.(((((((.....)))))))....((..(((((((((....(.((...(((((.....---...)))))......)).)....))))))).))..))..)))))))..... ( -53.40) >DroGri_CAF1 83748 117 + 1 CUACAUGCCAGCGGCCAUGGAUUGGCCGCACUCGUUUGUGCCCAGCGGCCGGGCAAUGUUCAUGAUG---UCCUGCAUUCUAUGGCCCUCAAUUUAGGCAGCUGGUUGGGCGUGCCCAGU ......(((((((((((((((..((((((....((....))...))))))(((((..........))---))).....)))))))))((......))...))))))((((....)))).. ( -47.50) >DroWil_CAF1 376 120 + 1 CUACAUGCCAGCGGUCAUGGAUUGGCCGCUUUGGUCUGUCAAUCGAAUGCCGGCCAGGUGGUGGAUGAUUUCGUCCAUUCGAUGGCACUAAAUUUGGGCAGUUGGCUGGGUGUGCCCAGU ..(((.(((((((((((.....)))))))..)))).)))....(((.((((.((((...((((((((....))))))))...)))).(.......))))).)))((((((....)))))) ( -50.90) >DroMoj_CAF1 17923 117 + 1 CUGCACGCCAGCGGCCACGGCCUGGCCGCACUCGUCUGCGCCCAGAGUCCGGGCAGCGUGCACGACG---UGCUCCACUCGAUGGCCCUCAACCUGGGCAGCUGGCUGGGCGUGCCCAGU ..(((((((.(((((((.....)))))))....(((.((((((((.((..((((..((.((((...)---)))......))...))))...)))))))).)).)))..)))))))..... ( -60.10) >DroAna_CAF1 781 117 + 1 CUCCAUGCCAGUGGCCACGGCCUGGCCGCCCUGGUCUGUGCCCAGUAUCCGGGCCAUGUGGAGGAUG---UCCUGCACUCGAUGGCCCUGAAUUUGGGCAGCUGGCUGGGUGUGCCCAGU ......(((((((((((.(((......))).)))))..(((((((..((.((((((((((.(((...---.))).)))...))))))).))..)))))))))))))((((....)))).. ( -62.50) >consensus CUGCAUGCCAGCGGCCACGGACUGGCCGCACUCGUCUGCGCCCAGAAUCCGGGCAAUGUGCAGGAUG___UCCUCCACUCGAUGGCCCUAAAUUUGGGCAGCUGGCUGGGCGUGCCCAGU ......(((((((((((.....)))))............((((((.....((((...(((((...........)))))......)))).....)))))).))))))((((....)))).. (-38.54 = -38.97 + 0.42)

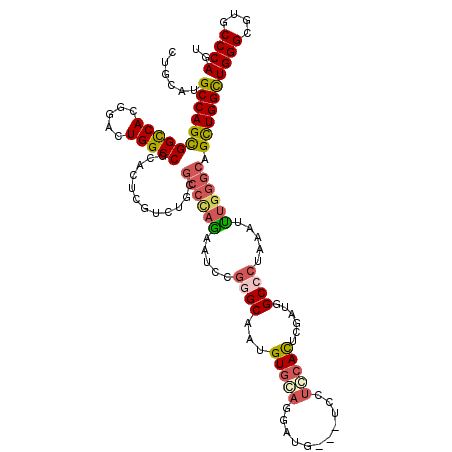
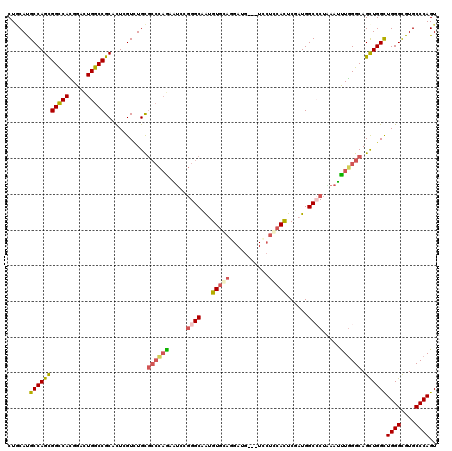
| Location | 21,136,165 – 21,136,282 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.11 |
| Mean single sequence MFE | -52.39 |
| Consensus MFE | -31.25 |
| Energy contribution | -33.17 |
| Covariance contribution | 1.92 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.914655 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 21136165 117 - 22224390 ACUGGGCACACCCAGCCAGCUGCCCAAAUUCAGGGCCAUCGAGUGGAGUA---CCUCCUCCACAUGGCCCUGAUUUUGGGCGCACACCAAGGCGGCCAGUCCGUGGCCACUGGCGUGCAG .(((((....)))))......(((((((.((((((((((...((((((..---....)))))))))))))))).)))))))((((.(((.(..(((((.....))))).)))).)))).. ( -68.60) >DroVir_CAF1 73978 117 - 1 ACUGGGCACGCCCAGCCAGCUGCCCAGAUUUAGCGCCAUGGAGUGCAGCA---CAUCGUGCACAUUGCCCGGACUCUGGGCGCAAACGAGUGCGGCCAGGCCAUGGCCACUGGCAUGCAG .(((((....)))))...((((((.((.....((((......)))).(((---(.((((......(((((((...)))))))...))))))))(((((.....))))).)))))).)).. ( -52.20) >DroGri_CAF1 83748 117 - 1 ACUGGGCACGCCCAACCAGCUGCCUAAAUUGAGGGCCAUAGAAUGCAGGA---CAUCAUGAACAUUGCCCGGCCGCUGGGCACAAACGAGUGCGGCCAAUCCAUGGCCGCUGGCAUGUAG ..((((....)))).(((((.(((........((((......(((.....---...))).......))))((((((....(......)...)))))).......))).)))))....... ( -40.72) >DroWil_CAF1 376 120 - 1 ACUGGGCACACCCAGCCAACUGCCCAAAUUUAGUGCCAUCGAAUGGACGAAAUCAUCCACCACCUGGCCGGCAUUCGAUUGACAGACCAAAGCGGCCAAUCCAUGACCGCUGGCAUGUAG .(((((....)))))...((((((..((((.((((((......((((.(....).))))((....))..)))))).))))..........(((((.((.....)).))))))))).)).. ( -35.50) >DroMoj_CAF1 17923 117 - 1 ACUGGGCACGCCCAGCCAGCUGCCCAGGUUGAGGGCCAUCGAGUGGAGCA---CGUCGUGCACGCUGCCCGGACUCUGGGCGCAGACGAGUGCGGCCAGGCCGUGGCCGCUGGCGUGCAG .....(((((((.((((((((.....)))))..((((((.(..(((.(((---(.((((.(..((.((((((...)))))))).)))))))))..)))..).)))))))))))))))).. ( -61.00) >DroAna_CAF1 781 117 - 1 ACUGGGCACACCCAGCCAGCUGCCCAAAUUCAGGGCCAUCGAGUGCAGGA---CAUCCUCCACAUGGCCCGGAUACUGGGCACAGACCAGGGCGGCCAGGCCGUGGCCACUGGCAUGGAG .(((((....)))))(((((((((((...((.(((((((...(((.(((.---...))).)))))))))).))...))))))(((.(((.(((......))).)))...))))).))).. ( -56.30) >consensus ACUGGGCACACCCAGCCAGCUGCCCAAAUUCAGGGCCAUCGAGUGCAGGA___CAUCCUCCACAUGGCCCGGACUCUGGGCACAGACCAGUGCGGCCAGGCCAUGGCCACUGGCAUGCAG ..((((....))))(((((.((((((......((((...((.(((((...........))))).))))))......))))))...........(((((.....))))).)))))...... (-31.25 = -33.17 + 1.92)

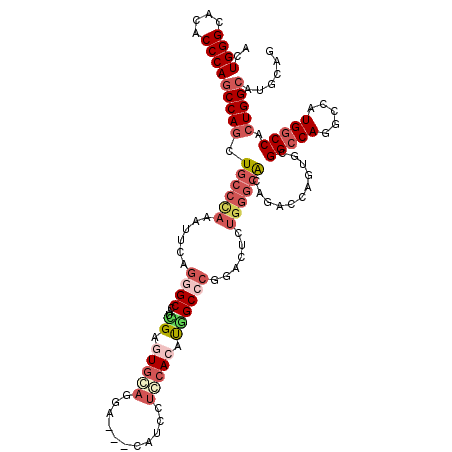
| Location | 21,136,205 – 21,136,322 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 83.36 |
| Mean single sequence MFE | -51.87 |
| Consensus MFE | -30.61 |
| Energy contribution | -30.45 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.59 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 21136205 117 - 22224390 CGCUGUUCUGCAGCGGACGGUGCAGCAGGACGAUGGGCAGACUGGGCACACCCAGCCAGCUGCCCAAAUUCAGGGCCAUCGAGUGGAGUACCUCCUCCACAUGGCCCUGAUUUUGGG ...((((((((.(((.....))).)))))))).(((((((.(((((....)))))....)))))))...((((((((((...((((((......))))))))))))))))....... ( -61.20) >DroVir_CAF1 74018 117 - 1 CGCUAUUCUGUAGCGGGCGAUGGAGCAGCACAAUGGGUAAACUGGGCACGCCCAGCCAGCUGCCCAGAUUUAGCGCCAUGGAGUGCAGCACAUCGUGCACAUUGCCCGGACUCUGGG .(((((...)))))((((((((.....((((.((((((((((((((((.((.......))))))))).))).)).))))...)))).((((...)))).)))))))).......... ( -49.30) >DroPse_CAF1 65554 117 - 1 CACUGUUCAGGAGUGGACGAUGCAACAGGACAAUGGGCAGACUGGGAACUCCCAGCCAGCUGCCCAAAUUCAGGGCCAUCGAGUGCAGGACAUCGUCGACAUGGCCCUGGCUCUGGG ((.((((((....)))))).))...(((..(..(((((((.(((((....)))))....)))))))....(((((((((((((((.....)))..))))..)))))))))..))).. ( -50.10) >DroGri_CAF1 83788 117 - 1 CGCUGUUCUGCAGCGGCCGAUGAAGUAACACAAUGGGCAGACUGGGCACGCCCAACCAGCUGCCUAAAUUGAGGGCCAUAGAAUGCAGGACAUCAUGAACAUUGCCCGGCCGCUGGG ..........((((((((............((((((((((.((((..........))))))))))..)))).((((......(((........))).......)))))))))))).. ( -43.92) >DroMoj_CAF1 17963 117 - 1 CGCUGUUCUGGAGCGGCCGAUGGAGCAGCACAAUGGGCAGACUGGGCACGCCCAGCCAGCUGCCCAGGUUGAGGGCCAUCGAGUGGAGCACGUCGUGCACGCUGCCCGGACUCUGGG ......(((((.(((((((((((..((((....(((((((.(((((....)))))....))))))).))))....))))))......((((...))))..))))))))))....... ( -56.60) >DroPer_CAF1 65767 117 - 1 CACUGUUCAGGAGUGGACGAUGCAACAGGACAAUGGGCAGACUGGGAACUCCCAGCCAGCUGCCCAAAUUCAGGGCCAUCGAGUGCAGGACAUCGUCGACAUGGCCCUGGCUCUGGG ((.((((((....)))))).))...(((..(..(((((((.(((((....)))))....)))))))....(((((((((((((((.....)))..))))..)))))))))..))).. ( -50.10) >consensus CGCUGUUCUGGAGCGGACGAUGCAGCAGCACAAUGGGCAGACUGGGCACGCCCAGCCAGCUGCCCAAAUUCAGGGCCAUCGAGUGCAGGACAUCGUCCACAUGGCCCGGACUCUGGG ((((.......))))..(((((..((..((...(((((((.(((((....)))))....)))))))...))...))))))).......................(((.......))) (-30.61 = -30.45 + -0.16)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:06:43 2006