| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 2,357,270 – 2,357,409 |
| Length | 139 |
| Max. P | 0.850519 |

| Location | 2,357,270 – 2,357,390 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.67 |
| Mean single sequence MFE | -46.27 |
| Consensus MFE | -30.35 |
| Energy contribution | -29.67 |
| Covariance contribution | -0.69 |
| Combinations/Pair | 1.47 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.66 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.518646 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
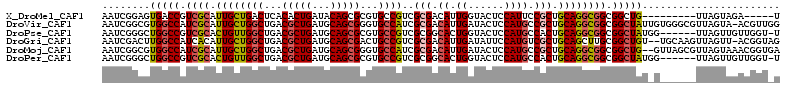
>X_DroMel_CAF1 2357270 120 - 22224390 CUGCAGCGGAAUGGAGUACCAAUGUCGCGACGGCACGCGCUGUAUCAGUGUGAGUCAGCAAUGCGACGGUCACUCCGAUUGCAGUGACGGCGACGACGAGGAGCAUUGCGACGGAAGUGA ..(..((((..(((....)))...))))..)(((.(((((((...))))))).))).(((((((..(.((((((........))))))(....).....)..))))))).((....)).. ( -43.40) >DroPse_CAF1 2059 120 - 1 CUGCAGUGGCAUGGAGUACCAGUGCCGCGACGGCACGCGCUGCAUCAGCGUCAGCCAACAGUGCGACGGCCAGCCCGAUUGCAGCGACGGCGACGACGAAGAGCACUGCGACGGAAGUAA .(((.(((((((((....))).))))))....)))((((.(((.((..((((.(((..(..((((((((.....))).)))))..)..)))...))))..))))).))))((....)).. ( -47.90) >DroGri_CAF1 2766 120 - 1 CUGCAGCGACAUGGAAUAUCAAUGUCGCGACGGCAGUCGCUGCAUCAGCGUCAGCCAGCAAUGUGAUGGCCAAGUCGAUUGCAACGAUGGCGACGAUGAGGACCACUGCGAUGGAAGUGA ((((.(((((((.(.....).)))))))....))))(((((.((((.((........))...((..(((((..((((.((((.......))))))))..)).)))..))))))..))))) ( -44.20) >DroWil_CAF1 3567 120 - 1 CUGCAGUGCCAUGGAAUAUCAAUGUCGCGAUGGCACUCGCUGCAUCAGCUCAAGCCAACAAUGCGAUGGCCAUCCUGAUUGCAGUGAUGGCGAUGAUGAGGAGCACUGCGAUGGAAGUGA .(((((((((......((((...((((((.((((....((((...))))....))))....)))))).(((((((((....))).)))))))))).....).)))))))).......... ( -44.30) >DroAna_CAF1 2017 120 - 1 AUGCAGUGCUAUGGAGUAUCAGUGCAGCGACGGUACGCGCUGUAUUAGUGUUAGCCAGACCUGUGACGGUCGCAUAGACUGCACCGGAGGCGACGACGAGGAGCACUGCGACGGAAGUGA .(((((((((.(((..(((.(((((((((.((...))))))))))).)))....)))..((((((.(((((.....))))))))((..(....)..))))))))))))))((....)).. ( -49.90) >DroPer_CAF1 2049 120 - 1 CUGCAGUGGCAUGGAGUACCAGUGCCGCGACGGCACGCGCUGCAUCAGCGUCAGCCAACAGUGCGACGGCCAGCCCGAUUGCAGCGACGGCGACGACGAAGAGCACUGCGACGGAAGUAA .(((.(((((((((....))).))))))....)))((((.(((.((..((((.(((..(..((((((((.....))).)))))..)..)))...))))..))))).))))((....)).. ( -47.90) >consensus CUGCAGUGGCAUGGAGUACCAAUGCCGCGACGGCACGCGCUGCAUCAGCGUCAGCCAACAAUGCGACGGCCACCCCGAUUGCAGCGACGGCGACGACGAGGAGCACUGCGACGGAAGUGA .(((.(((((((((....)).)))))))....)))((((.(((.((..((((.(((.....((((((((.....))).))))).....)))...))))..))))).))))((....)).. (-30.35 = -29.67 + -0.69)

| Location | 2,357,310 – 2,357,409 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 79.14 |
| Mean single sequence MFE | -44.56 |
| Consensus MFE | -26.58 |
| Energy contribution | -26.72 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.60 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
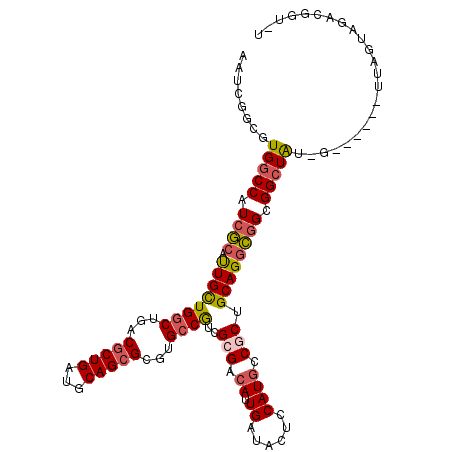
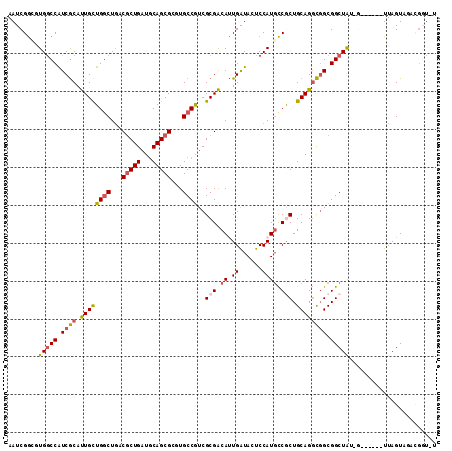
>X_DroMel_CAF1 2357310 99 + 22224390 AAUCGGAGUGACCGUCGCAUUGCUGACUCACACUGAUACAGCGCGUGCCGUCGCGACAUUGGUACUCCAUUCCGCUGCAGGCGGCGGCUG---------UUAGUAGA-----U .(((((((((.(((((((...((((..((.....))..))))(((...))).)))))...))))))))...((((((....))))))...---------......))-----) ( -34.70) >DroVir_CAF1 2679 112 + 1 AAUCGGCGUGGCCAUCGCAUUGCUGGCUGACGCUGAUGCAGCGGGUGCCAUCGCGACAUUGAUACUCCAUGCCGCUGCAGGCGGCGGCUAUUGUGGGCGUUAGUA-ACGUUGG .(((((((((((((.(.....).))))).)))))))).(((((.((((....)).))(((((..(((((.(((((((....)))))))...)).)))..))))).-.))))). ( -46.50) >DroPse_CAF1 2099 106 + 1 AAUCGGGCUGGCCGUCGCACUGUUGGCUGACGCUGAUGCAGCGCGUGCCGUCGCGGCACUGGUACUCCAUGCCACUGCAGGCGGCGGCUAUGG------UUAGUUGUUGGU-U ((((((((((((((((((.((((.(((....)))...)))).))).((((((((.(((.(((((.....))))).)))..)))))))).))))------)))))..)))))-) ( -49.30) >DroGri_CAF1 2806 110 + 1 AAUCGACUUGGCCAUCACAUUGCUGGCUGACGCUGAUGCAGCGACUGCCGUCGCGACAUUGAUAUUCCAUGUCGCUGCAGCUUGCGGCUGU--UGCAAGUUAGUU-ACGGUAG .......(..((((.(.....).))))..)(((((...))))).(((((((.(((((((.(.....).)))))((.(((((.....)))))--.))......)).-))))))) ( -41.30) >DroMoj_CAF1 3119 111 + 1 AAUCGGCGUGGCCAUCGCAUUGCUGGCUGACGCUGAUGCAGCGGGUGCCAUCGCGACAUUGAUACUCCAUGCCGCUGCAGGCGGCGGCUG--GUUAGCGUUAGUAAACGGUGA .(((((((((((((.(.....).))))).)))))))).((.((..(((...(((............(((.(((((((....)))))))))--)...)))...)))..)).)). ( -46.26) >DroPer_CAF1 2089 106 + 1 AAUCGGGCUGGCCGUCGCACUGUUGGCUGACGCUGAUGCAGCGCGUGCCGUCGCGGCACUGGUACUCCAUGCCACUGCAGGCGGCGGCUAUGG------UUAGUUGUUGGU-U ((((((((((((((((((.((((.(((....)))...)))).))).((((((((.(((.(((((.....))))).)))..)))))))).))))------)))))..)))))-) ( -49.30) >consensus AAUCGGCGUGGCCAUCGCAUUGCUGGCUGACGCUGAUGCAGCGCGUGCCGUCGCGACAUUGAUACUCCAUGCCGCUGCAGGCGGCGGCUAU_G______UUAGUAGACGGU_U ........(((((.((((.((((((((...(((((...)))))...))))..(((.((.((......)))).))).)))))))).)))))....................... (-26.58 = -26.72 + 0.14)
| Location | 2,357,310 – 2,357,409 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 79.14 |
| Mean single sequence MFE | -38.02 |
| Consensus MFE | -31.43 |
| Energy contribution | -30.27 |
| Covariance contribution | -1.16 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.79 |
| SVM RNA-class probability | 0.850519 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
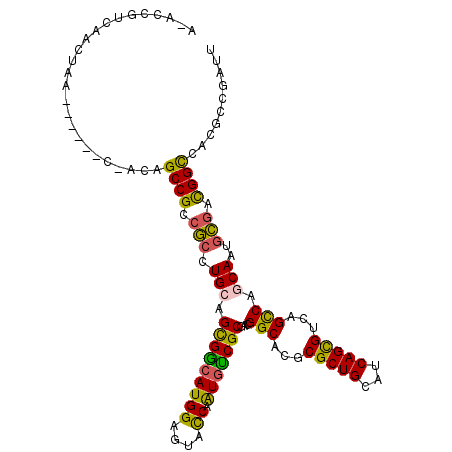
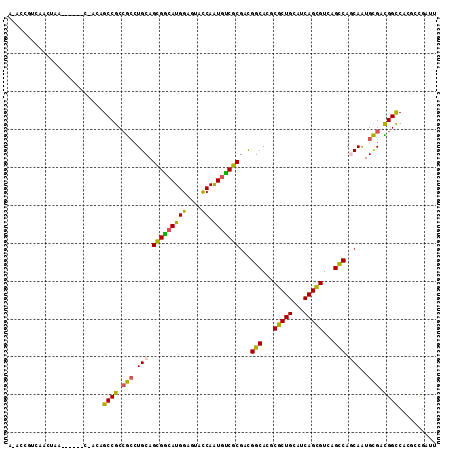
>X_DroMel_CAF1 2357310 99 - 22224390 A-----UCUACUAA---------CAGCCGCCGCCUGCAGCGGAAUGGAGUACCAAUGUCGCGACGGCACGCGCUGUAUCAGUGUGAGUCAGCAAUGCGACGGUCACUCCGAUU .-----........---------...((((.(....).))))..(((((((((...((((((..(((.(((((((...))))))).))).....))))))))).))))))... ( -35.90) >DroVir_CAF1 2679 112 - 1 CCAACGU-UACUAACGCCCACAAUAGCCGCCGCCUGCAGCGGCAUGGAGUAUCAAUGUCGCGAUGGCACCCGCUGCAUCAGCGUCAGCCAGCAAUGCGAUGGCCACGCCGAUU ....(((-.....))).........((((.(((.(((.(((((((.(.....).)))))))..((((...(((((...)))))...)))))))..))).)))).......... ( -35.80) >DroPse_CAF1 2099 106 - 1 A-ACCAACAACUAA------CCAUAGCCGCCGCCUGCAGUGGCAUGGAGUACCAGUGCCGCGACGGCACGCGCUGCAUCAGCGUCAGCCAACAGUGCGACGGCCAGCCCGAUU .-............------.....((((.((((((..(((((((((....))).))))))...(((..((((((...))))))..)))..))).))).)))).......... ( -41.20) >DroGri_CAF1 2806 110 - 1 CUACCGU-AACUAACUUGCA--ACAGCCGCAAGCUGCAGCGACAUGGAAUAUCAAUGUCGCGACGGCAGUCGCUGCAUCAGCGUCAGCCAGCAAUGUGAUGGCCAAGUCGAUU .......-.....(((((..--...((((((...(((.(((((((.(.....).)))))))...(((.(.(((((...))))).).))).)))...)).)))))))))..... ( -36.10) >DroMoj_CAF1 3119 111 - 1 UCACCGUUUACUAACGCUAAC--CAGCCGCCGCCUGCAGCGGCAUGGAGUAUCAAUGUCGCGAUGGCACCCGCUGCAUCAGCGUCAGCCAGCAAUGCGAUGGCCACGCCGAUU ....((((....))))....(--(((((((.(....).))))).)))...(((...((((((.((((...(((((...)))))...))))....))))))(((...)))))). ( -37.90) >DroPer_CAF1 2089 106 - 1 A-ACCAACAACUAA------CCAUAGCCGCCGCCUGCAGUGGCAUGGAGUACCAGUGCCGCGACGGCACGCGCUGCAUCAGCGUCAGCCAACAGUGCGACGGCCAGCCCGAUU .-............------.....((((.((((((..(((((((((....))).))))))...(((..((((((...))))))..)))..))).))).)))).......... ( -41.20) >consensus A_ACCGUCAACUAA______C_ACAGCCGCCGCCUGCAGCGGCAUGGAGUACCAAUGUCGCGACGGCACGCGCUGCAUCAGCGUCAGCCAGCAAUGCGACGGCCACGCCGAUU .........................((((.(((.(((.(((((((((....)).)))))))...(((...(((((...)))))...))).)))..))).)))).......... (-31.43 = -30.27 + -1.16)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:55:05 2006